Extrakromoszomális DNS
Az extrakromoszomális DNS (röviden ecDNS) a kromoszómákon kívüli DNS, vagy a sejtmagon belül, vagy azon kívül. Egy genom DNS-ének nagy része a sejtmagon belül kromoszómákban található meg. Számos extrakromoszomális DNS-változat van, és míg egyesek fontos biológiai funkciókat látnak el,[1] szerepet játszhatnak betegségekben, például a rákban.[2][3][4]
A prokarióták elsősorban plazmidokban, az eukarióták sejtszervecskékben tartalmaznak nem virális ecDNS-t.[1] A mitokondriális DNS az eukarióták extrakromoszomális DNS-ének fő forrása.[5] E sejtszervecske saját DNS-e alátámasztja, hogy a mitokondriumok eukarióták által bekebelezett baktériumsejtek voltak.[6] Az ecDNS-t gyakran használják a replikáció kutatásában, mivel könnyen azonosítható és izolálható.[1]
Bár extrakromoszomális körkörös DNS (eccDNS) megtalálható normál eukarióta sejtekben, a nem körkörös ecDNS ettől eltérő egység, melyet ráksejtek magjában azonosítottak, és sok onkogént hordozhat.[7][8][3] Az ecDNS a génerősítés fő mechanizmusa, sok onkogénmásolatot és agresszív rákokat okozva.
A citoplazmában lévő ecDNS szerkezetileg eltér a magitól. Kevésbé metilálódik a sejtmagban talált DNS-nél. Ezenkívül a citoplazmatikus DNS szekvenciái eltérnek a magitól egy élőlényen belül, így a citoplazmatikus DNS nem egyszerűen a maginak töredékeiből áll.[9] Ráksejtekben az ecDNS elsősorban a magba izolálódik.[2]
A magon kívüli DNS-en kívül a vírusgenom-fertőzések is ecDNS-t adnak.
Prokariótákban
[szerkesztés]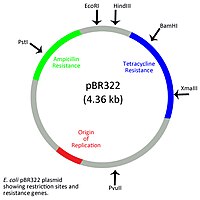
Bár a prokarióták nem rendelkeznek membránnal körülvett sejtmaggal, van maganyaguk, ahol a fő kromoszóma van. Az ecDNS az ezen kívüli részben található körkörös vagy lineáris plazmidokként. A bakteriális plazmidok általában rövidek, 1000 és néhány százezer bázis közt, és rendelkeznek replikációs origóval, mely lehetővé teszi a plazmid kromoszómától független replikációját.[10] Az adott plazmidok száma egy sejtben a másolatszám, és 2-től több száz másolatig terjedhet sejtenként.[11] A körkörös baktériumplazmidok osztályzása a benne kódolt gének speciális funkcióinak megfelelően történik. A termékenységi (f) plazmidok lehetővé teszi a konjugációt, az ellenállási (r) plazmidok ellenállást tesznek lehetővé számos antibiotikummal, például tetraciklinnel vagy ampicillinnel szemben. A virulenciaplazmidok a baktérium patogenicitását lehetővé tevő anyagokat tartalmaznak. A degeneratív plazmidok számos anyag, például aromás vegyületek és xenobiotikumok bontását teszik lehetővé.[12] A baktériumplazmidok pigmenttermelést, nitrogénkötést és nehézfémeknek való ellenállást is okozhatnak.[13]
A természetes körkörös plazmidok módosíthatók több rezisztenciagénre és egyedi resztrikciós helyekre, így értékes klónozási vektorok.[10] A körkörös baktériumplazmidok lehetővé tesznek DNS-vakcinák létrehozását. A plazmid-DNS-vakcinák patogén vírus, baktérium vagy más parazita által termelt antigént vagy fehérjét kódoló géneket tartalmaznak.[14] A gazdába jutva a plazmidgének termékei stimulálják a nem specifikus és a specifikus immunválaszt is. A plazmidok gyakran adjuvánssal vannak borítva az immunválasz javításához.[15]
Lineáris baktériumplazmidok találhatók egyes Spirochaete-fajokban, például a Borrelia nemzetségben, melynek tagja a Lyme-kórért felelős patogén; egyes Streptomyces-fajok, és a kénoxidáló Thiobacillus versutusban. A prokarióták lineáris plazmidjai vagy hajtűt, vagy a telomervéghez kovalensen kötődő fehérjét tartalmaznak. A Borrelia-baktériumok AT-gazdag hajtűi 5000-200 000 bázisból állnak,[16] és egy antigéncsoportért felelős géneket tartalmaznak, melyek lehetővé teszik az immunválasz elkerülését.[17] Az 5’-véghez csatlakozó fehérjét tartalmazó lineáris plazmidok az invertronok, méretük 9-től 600 kb közt lehet, és fordított terminális ismétlődésekből állnak.[16] A lineáris plazmák kovalensen kapcsolódó fehérjékkel segítheti a bakteriális konjugációt és a plazmid integrációját. E lineáris plazmák a legnagyobb ecDNS-t alkotják, mivel nemcsak egyes baktériumokban vannak jelen, hanem az eukariótákban lévő sejtekben is invertronszerkezetet vesznek fel, ahol a fehérje az 5’-véghez csatlakozik.[16][17]
A hosszú, lineáris „borg” egyes archeákban – melyek tartalmazhatják, és számos közös gént tartalmazhatnak – ismeretlen ecDNS-szerkezettel rendelkezhet.[18][19][20]
Eukariótákban
[szerkesztés]Mitokondriális
[szerkesztés]
A mitokondriumok az eukariótákban több mitokondriális DNS-t (mtDNS) tartalmaznak a mitokondriális mátrixban.[21] Többsejtű állatokban a körkörös mtDNS-kromoszóma 13 gént tartalmaz, melyek az elektrontranszportlánc fehérjéi kódolja, és 24 gént, melyek mitokondrális RNS-t; ezek 2 rRNS- és 22 tRNS-gént kódolnak.[22] Az állati mtDNS-plazmid mintegy 16,6 kb-os, és bár tRNS- és mRNS-szintézishez tartalmaz géneket, a magi gének kellenek az mtDNS-replikációhoz vagy a mitokondriális fehérjék transzlációjához.[23] A mitokondriális kromoszóma csak 1 kódoló szakasz nélküli részt tartalmaz, ez a D-ciklus, melyhez a magi szabályzó fehérjék kapcsolódnak.[22] Az mtDNS-molekulák mitokondriumonkénti száma fajonként és eltérő energiaigényű sejtenként eltér. Például az izom- és májsejtek több mtDNS-t tartalmaznak mitokondriumonként, mint a vér- és bőrsejtek.[23] Az elektrontranszportlánc közelsége a mitokondriális belső membránhoz és a reaktívoxigénszármazék-termeléshez, valamint az mtDNS hisztonokhoz kapcsolódásának és általuk való védelmének hiánya miatt az mtDNS érzékenyebb a DNS-sérülésre a magi DNS-nél.[24] Az mtDNS-sérülés esetén a DNS javítható báziskivágásos javítással, vagy a sérült mtDNS megszűnik (a mitokondrium károsodása nélkül, mivel mitokondriumonként több mtDNS-példány van jelen).[25]
A géntranszlációhoz tartozó szabályos genetikai kód állandó – minden 3 bázisos szekvencia azonos aminosavat kódol, függetlenül a fajtól, amelyé a DNS. Azonban ez a kód kissé eltér gombák, állatok, protiszták és növények mtDNS-ében.[21] Bár a 3 bázisos szekvenciák (kodonok) legtöbbje azonos aminosavat kódol a magi genetikai kóddal, egyesek ettől eltérőt kódolnak.
| Genetikai kód | Transzláció | DNS-kodon | RNS-kodon | Transzláció e kóddal | Összehasonlítás az univerzális kóddal |
|---|---|---|---|---|---|
| Gerincesek | 2 | AGA
|
AGA
|
Ter (*)
|
Arg (R)
|
AGG
|
AGG
|
Ter (*)
|
Arg (R)
| ||
ATA
|
AUA
|
Met (M)
|
Ile (I)
| ||
TGA
|
UGA
|
Trp (W)
|
Ter (*)
| ||
| Élesztőgombák | 3 | ATA
|
AUA
|
Met (M)
|
Ile (I)
|
CTT
|
CUU
|
Thr (T)
|
Leu (L)
| ||
CTC
|
CUC
|
Thr (T)
|
Leu (L)
| ||
CTA
|
CUA
|
Thr (T)
|
Leu (L)
| ||
CTG
|
CUG
|
Thr (T)
|
Leu (L)
| ||
TGA
|
UGA
|
Trp (W)
|
Ter (*)
| ||
CGA
|
CGA
|
nincs jelen | Arg (R)
| ||
CGC
|
CGC
|
nincs jelen | Arg (R)
| ||
| Penész-, protozoon- és cölentrelát-mitokondriáliskód és mycoplasma/spiroplasma-kód | 4–7 | TGA
|
UGA
|
Trp (W)
|
Ter (*)
|
| Gerinctelenek mitokondriális kódja | 5 | AGA
|
AGA
|
Ser (S)
|
Arg (R)
|
AGG
|
AGG
|
Ser (S)
|
Arg (R)
| ||
ATA
|
AUA
|
Met (M)
|
Ile (I)
| ||
TGA
|
UGA
|
Trp (W)
|
Ter (*)
| ||
| Tüskésbőrű- és laposféreg-mitokondriáliskód | 9 | AAA
|
AAA
|
Asn (N)
|
Lys (K)
|
AGA
|
AGA
|
Ser (S)
|
Arg (R)
| ||
AGG
|
AGG
|
Ser (S)
|
Arg (R)
| ||
TGA
|
UGA
|
Trp (W)
|
Ter (*)
| ||
| Aszcídia-mitokondráliskód | 13 | AGA
|
AGA
|
Gly (G)
|
Arg (R)
|
AGG
|
AGG
|
Gly (G)
|
Arg (R)
| ||
ATA
|
AUA
|
Met (M)
|
Ile (I)
| ||
TGA
|
UGA
|
Trp (W)
|
Ter (*)
| ||
| Alternatív laposféreg-mitokondriáliskód | 14 | AAA
|
AAA
|
Asn (N)
|
Lys (K)
|
AGA
|
AGA
|
Ser (S)
|
Arg (R)
| ||
AGG
|
AGG
|
Ser (S)
|
Arg (R)
| ||
TAA
|
UAA
|
Tyr (Y)
|
Ter (*)
| ||
TGA
|
UGA
|
Trp (W)
|
Ter (*)
| ||
| Chlorophycea-mitokondráliskód | 16 | TAG
|
UAG
|
Leu (L)
|
Ter (*)
|
| Métely-mitokondriáliskód | 21 | TGA
|
UGA
|
Trp (W)
|
Ter (*)
|
ATA
|
AUA
|
Met (M)
|
Ile (I)
| ||
AGA
|
AGA
|
Ser (S)
|
Arg (R)
| ||
AGG
|
AGG
|
Ser (S)
|
Arg (R)
| ||
AAA
|
AAA
|
Asn (N)
|
Lys (K)
| ||
| Scenedesmus obliquus-mitokondráliskód | 22 | TCA
|
UCA
|
Ter (*)
|
Ser (S)
|
TAG
|
UAG
|
Leu (L)
|
Ter (*)
| ||
| Thraustochytrium-mitokondráliskód | 23 | TTA
|
UUA
|
Ter (*)
|
Leu (L)
|
| Pterobranchia-mitokondriáliskód | 24 | AGA
|
AGA
|
Ser (S)
|
Arg (R)
|
AGG
|
AGG
|
Lys (K)
|
Arg (R)
| ||
TGA
|
UGA
|
Trp (W)
|
Ter (*)
|
| Aminosavak | apoláris | poláris | bázikus | savas | stop kodon |
A kódolási különbségek okai lehetnek a transzfer RNS-ek kémiai eltérései, melyek az mtDNS-transzkripcióval keletkező messenger RNS-ekkel kölcsönhatnak.[26]
Kloroplasztisz
[szerkesztés]Az eukarióta kloroplasztiszok és más növényi plasztiszok is tartalmaznak ecDNS-t. A legtöbb kloroplasztisz egy gyűrűs kromoszómában tartalmazza örökítőanyagát, azonban több kisebb gyűrűs plazmidra is ismert bizonyíték.[27][28][29] Egy 2004-es elmélet megkérdőjelezi a kloroplasztisz-DNS (cpDNS) gyűrű alakját, szerinte a cpDNS gyakrabban lineáris.[30] Egy cpDNS 100-200 génből állhat,[31] mérete fajonként eltérhet. A cpDNS szövetes növényekben 120-160 kb-os. [21] A cpDNS génjei a fotoszintézishez szükséges mRNS-eket, továbbá tRNS-eket, rRNS-eket, RNS-polimeráz- és riboszomális fehérjealegységeket kódolnak.[32] Az mtDNS-hez hasonlóan a cpDNS nem teljesen autonóm, és magi géneket használ a replikációhoz és fehérjéi termeléséhez. A kloroplasztiszok több cpDNS-másolatot tartalmaz, ezek száma nemcsak fajonként vagy sejttípusonként, hanem annak korától és fejlettségétől függően egy sejten belül is. Például a cpDNS-tartalom a fejlődés korai szakaszában, ahol a kloroplasztiszok proplasztiszként vannak jelen, sokkal nagyobb, mint a kitágult, érett sejtben, ahol a plasztiszok teljesen érettek.[33]
Körkörös
[szerkesztés]Az eccDNS minden eukarióta sejtben jelen van, általában genomi DNS-ből származik, és ismétlődő DNS-szekvenciákból áll a kódoló és nem kódoló kromoszómaszakaszokban. Az eccDNS mérete kevesebb mint 2000-től több mint 20 000 bázispárig terjedhet.[34] A növényi eccDNS a kromoszómák centromerjéhez és a repetitív szatellit-DNS-hez hasonló ismétlődő szekvenciákat tartalmaz.[35] Állatokban az eccDNS a szatellit-, 5S-riboszomális és telomer-DNS-hez hasonlít.[34] Egyes élőlények, például az élesztő eccDNS-termelése a DNS-replikáción alapul,[35] míg másokban, például az emlősökben a replikációtól függetlenül keletkezhet eccDNS.[36] Az eccDNS funkciója nincs széles körben kutatva, de feltehetően az eccDNS keletkezése genombeli DNS-ből növeli az eukarióta genom plaszticitását, és befolyásolhatja a genomstabilitást, a sejtöregedést és a kromoszómafejlődést.[37]
Extrakromoszomális DNS figyelhető meg gyakran a humán ráksejtekben.[2][3][4] A ráksejtek ecDNS-e egy vagy több, szelektív előnyt adó gént tartalmaz. Az ecDNS hosszabb az eccDNS-nél, fénymikroszkóppal megfigyelhető. A ráksejtek ecDNS-e általában 1-3 Mb-osak vagy hosszabbak.[2] A nagy ecDNS megtalálható humán ráksejtek magjában, és több, a tumorsejtben átírt onkogént tartalmaz. Ezért feltehetően az ecDNS összefügghet a rákkal.
Speciális eszközök vannak az ecDNS azonosítására, például:
- Paul Mischel és Vineet Bafna által fejlesztett szoftver, mely lehetővé teszi az ecDNS azonosítását mikroszkópos képeken
- a fizikai ecDNS-izolációt lehetővé tevő Circle-Seq, mely enzimmel távolítja el a lineáris DNS-t, és az eccDNS-t izolálja. Ezt Brigitte Regenberg és csapata fejlesztette ki a Koppenhágai Egyetemen.[38]
Virális
[szerkesztés]A virális DNS-ek gyakori extrakromoszomális DNS-ek. A vírusgenomok megismerése fontos a vírus evolúciójának és mutációinak ismeretéhez.[39] Egyes vírusok, például a HIV és az onkogén vírusok, saját DNS-üket a gazda genomjába illesztik.[40] A vírusgenomok állhatnak egy- (ssDNS) vagy kétszálú DNS-ből (dsDNS), és megtalálhatók lineáris és körkörös változatban.[41]
Extrakromoszomális DNS-t tartalmazó vírus például a humán papillómavírus (HPV). DNS-genomja 3 különböző replikációs fázison megy keresztül: bekerülés, fenntartás, erősödés. Az anogenitalis rendszert és a szájat fertőzi meg. Általában a HPV-t az immunrendszer észleli és eltávolítja. A virális DNS felismerése fontos az immunválaszban. A vírus fennmaradásához a genomnak replikálódni és öröklődni kell sejtosztódáskor.[42]
Gazdasejt általi felismerés
[szerkesztés]A sejtek felismerhetik az idegen citoplazmatikus DNS-t. A felismerési utak megértése betegségek megelőzésében és kezelésében is fontos lehet.[43] A sejtek specifikusan felismerhetik a vírus-DNS-t például a Toll-típusú (TLR) útvonal révén.[44]
A Toll-útvonalról először rovarokban ismerték fel, hogy képes a sejteket érzékelőként működtetni számos baktérium-, vírusgenom vagy PAMP (patogén-asszociált molekuláris minta) észleléséhez. A PAMP-ek a nem spcifikus immunválasz jelentős aktivátorai. Nagyjából 10 humán TLR ismert. A különböző TLR-ek különböző PAMP-eket észlelnek: a TLR4 lipopoliszacharidokat, a vírus-dsRNS-t a TLR3, a vírus-ssRNS-t a TLR7/TLR8, a vírus-/bakteriális metilálatlan DNS-t a TLR9. Ez utóbbi észleli a CpG DNS-t, mely baktériumokban és vírusokban gyakori, és elindítja az IFN (I-es típusú interferonok) és más citokinek készítését.[44]
Öröklődés
[szerkesztés]
Az ecDNS öröklődése eltér a kromoszómákban lévő magi DNS-étől. Szembena a kromoszómákkal, az ecDNS nem tartalmaz centromereket, így nem mendeli öröklődési mintát mutat, mely heterogén sejtpopulációkat ad. Emberben a citoplazma nagy része az anya petesejtje révén öröklődik.[45] Ezért a sejtalkotók, például a mitokondrium DNS-e anyai ágon öröklődik. Az mtDNS vagy más citoplazmatikus DNS mutációi szintén anyai ágon öröklődnek. Ez az egyszülős öröklődés példa a nem mendeli öröklődésre. A növények is egyszülős öröklődést mutatnak. A legtöbb növény anyai ágon örököl mtDNS-t, fontos kivétel a Sequoia sempervirens, mely apai ágon örökli.[46]
Két elmélet van, miért örököl ritkán az utód apai mtDNS-t. Az egyik szerint az apai mtDNS egyszerűen alacsonyabb koncentrációban van jelen az anyainál, így nem észlelhető az utódban. A másik, összetettebb elmélet az apai mtDNS öröklődését megakadályozó bontásáról szól. Feltehetően az egyszülős mtDNS-öröklődés a citoplazmatikus DNS homoplazmiáját tartja fenn.[46]
Klinikai jelentősége
[szerkesztés]Néha EE-knek (extrakromoszomális elem) is nevezik, és az eukarióták genomikai instabilitásával is összefügghet. A kis polidiszpergált DNS-ek gyakoriak a genominstabilitással együtt. Az spcDNS-ek ismétlődő szekvenciákból, például szatellit-DNS-ből, retrovírusszerű DNS-elemekből és genomon belüli transzponálható elemekből származnak. Feltehetően génátrendeződési termékek.
Az ecDNS-t a rákban korábban nevezték kettős kicsi kromoszómáknak (DM), melyek páros kromatintesteknek tűnnek fénymikroszkóp alatt. Ezek a rákokozó ecDNS mintegy 30%-át adják, beleértve az egyszeri testeket, és kiderült, hogy azokkal azonos tartalmúak.[3] Az ecDNS név minden nagy, onkogéntartalmú extrakromoszomális DNS-t magába foglal. Ez gyakori bizonyos hisztológiájú ráksejtekben, de normál sejtekben nem fordul elő.[3] Az ecDNS feltehetően kettős száltöréssel a kromoszómában vagy egy élőlényben való DNS-túlreplikációval keletkezik. Ezenkívül rák és más genomikai instabilitás esetén nagyobb az EE-szint.[5]
Az mtDNS számos módon szerepet játszhat a betegségekben. A pontmutációk vagy alternatív génelrendeződések számos, a szívet, a központi idegrendszert, az endokrin rendszert, az emésztőrendszert és a vesét érintő betegséghez kapcsolódnak.[22] A mitokondriumokban lévő mtDNS elvesztése több betegséghez, mitokondriális depléciós szindrómákhoz (MDD) vezethet melyek a májat, a központi és perifériás idegrendszert, a simaizmot és a hallást érintik.[23] Vegyes, néha egymásnak ellentmondó eredmények is vannak tanulmányokban, melyek az mtDNS-példányszámot egyes rákok fejlődésével kapcsolják össze. Tanulmányokat végeztek a nagyobb és a kisebb mtDNS-szintek és a mellrák közti összefüggés kiderítésére. Pozitív asszociációt észleltek a nagyobb mtDNS-szint és a vesetumor kialakulása közt, de nincs kapcsolat az mtDNS-szintek és a gyomorrák közt.[47]
Az Apicomplexa protozooncsoportban jelen van ecDNS. A maláriaparazita (Plasmodium), az AIDS-hez kapcsolódó patogének (Taxoplasma, Cryptosporidium) szintén e csoport tagjai. A Plasmodiumban található mtDNS.[48] Kétféle ecDNS-e van: az egyik 6 kb-os lineáris, a másik 35 kb-os körkörös DNS. Ezek antibiotikumok lehetséges nukleotidcélpontjai.[49]
Szerepe a rákban
[szerkesztés]A génerősítés az egyik leggyakoribb onkogénaktivációs módszer. Ez a rákban gyakran extrakromoszomális körkörös elemeken történik.[50][4] Az ecDNS egyik fő funkciója a tumor gyors terjedése nagy sejtközi genetikai homogenitással.[3][8] A leggyakrabban erősített onkogének az ecDNS-en vannak, és erősen dinamikusak, nem natív kromoszómákba integrálódnak vissza homogén festési régióként (HSR),[51][3] és megváltoztatva a másolatszámokat és összetételeket bizonyos kezelések után.[52][7][53]
Az ecDNS a legsúlyosabb és legelőrehaladottabb rákokért és a rák elleni szerekkel szembeni ellenállásért is felel.[54]
Az ecDNS körkörös szerkezete úgy tér el a kromoszomális lineáris alakjától, hogy ez befolyásolhatja a rák patogenezisét.[55] Az ecDNS-en kódolt onkogének transzkripciója jelentős. Szemben a bakteriális plazmirdokkal vagy az mtDNS-sel, az ecDNS kromatinizált, sok aktív hisztont tartalmaz, de kevés represszív hisztont. Az ecDNS kromatinszerkezetében nincs jelen a kromoszomálisra jellemző nagyobb rendű tömörülés, így ez a rákgenom legelérhetőbbjei közt van.
Az ecDNS-ek csoportosíthatók a mag köré (ecDNS-hubok).[56] Ezek onkogéntúlexpressziót okozó géninterakciókat okozhatnak.
Jegyzetek
[szerkesztés]- ↑ a b c Rush MG, Misra R (1985. november 1.). „Extrachromosomal DNA in eucaryotes”. Plasmid 14 (3), 177–191. o. DOI:10.1016/0147-619X(85)90001-0. PMID 3912782.
- ↑ a b c d Verhaak RG, Bafna V, Mischel PS (2019. május 1.). „Extrachromosomal oncogene amplification in tumour pathogenesis and evolution”. Nature Reviews. Cancer 19 (5), 283–288. o. DOI:10.1038/s41568-019-0128-6. PMID 30872802. PMC 7168519.
- ↑ a b c d e f g Turner KM, Deshpande V, Beyter D, Koga T, Rusert J, Lee C, Li B, Arden K, Ren B, Nathanson DA, Kornblum HI, Taylor MD, Kaushal S, Cavenee WK, Wechsler-Reya R, Furnari FB, Vandenberg SR, Rao PN, Wahl GM, Bafna V, Mischel PS (2017. március 1.). „Extrachromosomal oncogene amplification drives tumour evolution and genetic heterogeneity”. Nature 543 (7643), 122–125. o. DOI:10.1038/nature21356. PMID 28178237. PMC 5334176.
- ↑ a b c Cancer May Be Driven by DNA Outside of Chromosomes (angol nyelven). The Scientist Magazine® . (Hozzáférés: 2021. október 5.)
- ↑ a b Kuttler F, Mai S (2007. február 1.). „Formation of non-random extrachromosomal elements during development, differentiation and oncogenesis”. Seminars in Cancer Biology 17 (1), 56–64. o. DOI:10.1016/j.semcancer.2006.10.007. PMID 17116402.
- ↑ Alberts, Bruce. Essential Cell Biology, Fourth, New York, New York, USA: Garland Science, 449. o. (2014. november 4.). ISBN 978-0-8153-4454-4
- ↑ a b Nathanson DA, Gini B, Mottahedeh J, Visnyei K, Koga T, Gomez G, Eskin A, Hwang K, Wang J, Masui K, Paucar A, Yang H, Ohashi M, Zhu S, Wykosky J, Reed R, Nelson SF, Cloughesy TF, James CD, Rao PN, Kornblum HI, Heath JR, Cavenee WK, Furnari FB, Mischel PS (2014. január 1.). „Targeted therapy resistance mediated by dynamic regulation of extrachromosomal mutant EGFR DNA”. Science 343 (6166), 72–76. o. DOI:10.1126/science.1241328. PMID 24310612. PMC 4049335.
- ↑ a b deCarvalho AC, Kim H, Poisson LM, Winn ME, Mueller C, Cherba D, Koeman J, Seth S, Protopopov A, Felicella M, Zheng S, Multani A, Jiang Y, Zhang J, Nam DH, Petricoin EF, Chin L, Mikkelsen T, Verhaak RG (2018. május 1.). „Discordant inheritance of chromosomal and extrachromosomal DNA elements contributes to dynamic disease evolution in glioblastoma”. Nature Genetics 50 (5), 708–717. o. DOI:10.1038/s41588-018-0105-0. PMID 29686388. PMC 5934307.
- ↑ Koch J, Vogt G, Kissel W (1983. május 1.). „Cytoplasmic DNA is structurally different from nuclear DNA”. Die Naturwissenschaften 70 (5), 252–4. o. DOI:10.1007/BF00405447. PMID 6877387.
- ↑ a b Nelson, David. Lehninger Principles of Biochemistry. New York: W. H. Freeman and Company, 307–308. o. (2008). ISBN 978-0-7167-7108-1
- ↑ Watson, James. Recombinant RNA: Genes and Genomes- A Short Course. New York: W. H. Freeman and Company, 81. o. (2007). ISBN 978-0-7167-2866-5
- ↑ Dib JR, Liebl W, Wagenknecht M, Farías ME, Meinhardt F (2013. január 1.). „Extrachromosomal genetic elements in Micrococcus”. Applied Microbiology and Biotechnology 97 (1), 63–75. o. DOI:10.1007/s00253-012-4539-5. PMID 23138713.
- ↑ Barnum, Susan. Biotechnology- An Introduction. California: Brooks / Cole, 62–63. o. (2005). ISBN 978-0-495-11205-1
- ↑ Laddy DJ, Weiner DB (2006). „From plasmids to protection: a review of DNA vaccines against infectious diseases”. International Reviews of Immunology 25 (3–4), 99–123. o. DOI:10.1080/08830180600785827. PMID 16818367.
- ↑ Ongkudon CM, Ho J, Danquah MK (2011. március 1.). „Mitigating the looming vaccine crisis: production and delivery of plasmid-based vaccines”. Critical Reviews in Biotechnology 31 (1), 32–52. o. DOI:10.3109/07388551.2010.483460. PMID 20879832.
- ↑ a b c Hinnebusch J, Tilly K (1993. december 1.). „Linear plasmids and chromosomes in bacteria”. Molecular Microbiology 10 (5), 917–922. o. DOI:10.1111/j.1365-2958.1993.tb00963.x. PMID 7934868.
- ↑ a b Meinhardt F, Schaffrath R, Larsen M (1997. április 1.). „Microbial linear plasmids”. Applied Microbiology and Biotechnology 47 (4), 329–36. o. DOI:10.1007/s002530050936. PMID 9163946.
- ↑ Dance, Amber (2021. július 16.). „Massive DNA 'Borg' structures perplex scientists” (angol nyelven). Nature 595 (7869), 636. o. DOI:10.1038/d41586-021-01947-3.
- ↑ „Previously undiscovered DNA 'borgs' found on California wetlands”, The Independent, 2021. július 30. (Hozzáférés: 2021. augusztus 13.) (angol nyelvű)
- ↑ (2021. július 10.) „Borgs are giant extrachromosomal elements with the potential to augment methane oxidation” (angol nyelven). bioRxiv, 2021.07.10.451761. o. DOI:10.1101/2021.07.10.451761. (Hozzáférés: 2021. augusztus 13.)
- ↑ a b c Lodish, Harvey. Molecular Cell Biology, 7th, New York: W.H. Freeman and Company, 245–251. o. (2013). ISBN 978-1-4641-2398-6
- ↑ a b c Chinnery PF, Turnbull DM (1999. július 1.). „Mitochondrial DNA and disease”. Lancet 354 Suppl 1 (9176), SI17-21. o. DOI:10.1016/S0140-6736(99)90244-1. PMID 10437851.
- ↑ a b c Dimmock D, Tang LY, Schmitt ES, Wong LJ (2010. július 1.). „Quantitative evaluation of the mitochondrial DNA depletion syndrome”. Clinical Chemistry 56 (7), 1119–27. o. DOI:10.1373/clinchem.2009.141549. PMID 20448188.
- ↑ Bohr VA, Anson RM (1999. augusztus 1.). „Mitochondrial DNA repair pathways”. Journal of Bioenergetics and Biomembranes 31 (4), 391–8. o. DOI:10.1023/A:1005484004167. PMID 10665528.
- ↑ Bendich AJ (2010. június 1.). „Mitochondrial DNA, chloroplast DNA and the origins of development in eukaryotic organisms”. Biology Direct 5 (42), 42. o. DOI:10.1186/1745-6150-5-42. PMID 20587059. PMC 2907347.
- ↑ Bernt M, Braband A, Schierwater B, Stadler PF (2013. november 1.). „Genetic aspects of mitochondrial genome evolution”. Molecular Phylogenetics and Evolution 69 (2), 328–38. o. DOI:10.1016/j.ympev.2012.10.020. PMID 23142697.
- ↑ Clegg MT, Gaut BS, Learn GH, Morton BR (1994. július 1.). „Rates and patterns of chloroplast DNA evolution”. Proceedings of the National Academy of Sciences of the United States of America 91 (15), 6795–6801. o. DOI:10.1073/pnas.91.15.6795. PMID 8041699. PMC 44285.
- ↑ Lilly JW, Havey MJ, Jackson SA, Jiang J (2001. február 1.). „Cytogenomic analyses reveal the structural plasticity of the chloroplast genome in higher plants”. The Plant Cell 13 (2), 245–54. o. DOI:10.1105/tpc.13.2.245. PMID 11226183. PMC 102240.
- ↑ Aronsson, Henrik. The chloroplast interactions with the environment, Online-Ausg., Berlin: Springer, 18. o. (2009). ISBN 978-3540686965
- ↑ Bendich AJ (2004. július 1.). „Circular chloroplast chromosomes: the grand illusion”. The Plant Cell 16 (7), 1661–6. o. DOI:10.1105/tpc.160771. PMID 15235123. PMC 514151.
- ↑ Wang Y, Ding J, Daniell H, Hu H, Li X (2012. szeptember 1.). „Motif analysis unveils the possible co-regulation of chloroplast genes and nuclear genes encoding chloroplast proteins”. Plant Molecular Biology 80 (2), 177–87. o. DOI:10.1007/s11103-012-9938-6. PMID 22733202.
- ↑ Pfalz J, Pfannschmidt T (2013. április 1.). „Essential nucleoid proteins in early chloroplast development”. Trends in Plant Science 18 (4), 186–94. o. DOI:10.1016/j.tplants.2012.11.003. PMID 23246438.
- ↑ Rowan BA, Bendich AJ (2009). „The loss of DNA from chloroplasts as leaves mature: fact or artefact?”. Journal of Experimental Botany 60 (11), 3005–10. o. DOI:10.1093/jxb/erp158. PMID 19454766.
- ↑ a b Cohen S, Houben A, Segal D (2008. március 1.). „Extrachromosomal circular DNA derived from tandemly repeated genomic sequences in plants”. The Plant Journal 53 (6), 1027–34. o. DOI:10.1111/j.1365-313X.2007.03394.x. PMID 18088310.
- ↑ Cohen Z, Lavi S (2009. július 1.). „Replication independent formation of extrachromosomal circular DNA in mammalian cell-free system”. PLOS ONE 4 (7), e6126. o. DOI:10.1371/journal.pone.0006126. PMID 19568438. PMC 2699479.
- ↑ Cohen S, Agmon N, Sobol O, Segal D (2010. március 1.). „Extrachromosomal circles of satellite repeats and 5S ribosomal DNA in human cells”. Mobile DNA 1 (1), 11. o. DOI:10.1186/1759-8753-1-11. PMID 20226008. PMC 3225859.
- ↑ The curious DNA circles that make treating cancer so hard. cen.acs.org . (Hozzáférés: 2021. október 2.)
- ↑ Sanjuán R, Nebot MR, Chirico N, Mansky LM, Belshaw R (2010. október 1.). „Viral mutation rates”. Journal of Virology 84 (19), 9733–48. o. DOI:10.1128/JVI.00694-10. PMID 20660197. PMC 2937809.
- ↑ Silverthorn, Dee Unglaub. Human Physiology. Peason/Benjamin Cummings (2007)
- ↑ Viral Genomes
- ↑ Lorenz LD, Rivera Cardona J, Lambert PF (2013. október 1.). „Inactivation of p53 rescues the maintenance of high risk HPV DNA genomes deficient in expression of E6”. PLOS Pathogens 9 (10), e1003717. o. DOI:10.1371/journal.ppat.1003717. PMID 24204267. PMC 3812038.
- ↑ Barber GN (2011. szeptember 1.). „Cytoplasmic DNA innate immune pathways”. Immunological Reviews 243 (1), 99–108. o. DOI:10.1111/j.1600-065X.2011.01051.x. PMID 21884170.
- ↑ a b Barber GN (2011. február 1.). „Innate immune DNA sensing pathways: STING, AIMII and the regulation of interferon production and inflammatory responses”. Current Opinion in Immunology 23 (1), 10–20. o. DOI:10.1016/j.coi.2010.12.015. PMID 21239155. PMC 3881186.
- ↑ Griffiths AJ. An Introduction to Genetic Analysis. New York: W.H.Freeman (2000)
- ↑ a b Sato M, Sato K (2013. augusztus 1.). „Maternal inheritance of mitochondrial DNA by diverse mechanisms to eliminate paternal mitochondrial DNA”. Biochimica et Biophysica Acta (BBA) - Molecular Cell Research 1833 (8), 1979–84. o. DOI:10.1016/j.bbamcr.2013.03.010. PMID 23524114.
- ↑ Thyagarajan B, Wang R, Nelson H, Barcelo H, Koh WP, Yuan JM (2013. június 1.). „Mitochondrial DNA copy number is associated with breast cancer risk”. PLOS ONE 8 (6), e65968. o. DOI:10.1371/journal.pone.0065968. PMID 23776581. PMC 3680391.
- ↑ Wilson RJ, Williamson DH (1997. március 1.). „Extrachromosomal DNA in the Apicomplexa”. Microbiology and Molecular Biology Reviews 61 (1), 1–16. o. DOI:10.1128/mmbr.61.1.1-16.1997. PMID 9106361. PMC 232597.
- ↑ Creasey A, Mendis K, Carlton J, Williamson D, Wilson I, Carter R (1994. május 1.). „Maternal inheritance of extrachromosomal DNA in malaria parasites”. Molecular and Biochemical Parasitology 65 (1), 95–8. o. DOI:10.1016/0166-6851(94)90118-X. PMID 7935632.
- ↑ Kim H, Nguyen NP, Turner K, Wu S, Gujar AD, Luebeck J, Liu J, Deshpande V, Rajkumar U, Namburi S, Amin SB, Yi E, Menghi F, Schulte JH, Henssen AG, Chang HY, Beck CR, Mischel PS, Bafna V, Verhaak RG (2020. augusztus 1.). „Extrachromosomal DNA is associated with oncogene amplification and poor outcome across multiple cancers”. Nature Genetics 52 (8), 891–897. o. DOI:10.1038/s41588-020-0678-2. PMID 32807987. PMC 7484012.
- ↑ Storlazzi CT, Lonoce A, Guastadisegni MC, Trombetta D, D'Addabbo P, Daniele G, L'Abbate A, Macchia G, Surace C, Kok K, Ullmann R, Purgato S, Palumbo O, Carella M, Ambros PF, Rocchi M (2010. szeptember 1.). „Gene amplification as double minutes or homogeneously staining regions in solid tumors: origin and structure”. Genome Research 20 (9), 1198–206. o. DOI:10.1101/gr.106252.110. PMID 20631050. PMC 2928498.
- ↑ Von Hoff DD, McGill JR, Forseth BJ, Davidson KK, Bradley TP, Van Devanter DR, Wahl GM (1992. szeptember 1.). „Elimination of extrachromosomally amplified MYC genes from human tumor cells reduces their tumorigenicity”. Proceedings of the National Academy of Sciences of the United States of America 89 (17), 8165–9. o. DOI:10.1073/pnas.89.17.8165. PMID 1518843. PMC 49877.
- ↑ Xu K, Ding L, Chang TC, Shao Y, Chiang J, Mulder H, Wang S, Shaw TI, Wen J, Hover L, McLeod C, Wang YD, Easton J, Rusch M, Dalton J, Downing JR, Ellison DW, Zhang J, Baker SJ, Wu G (2019. január 1.). „Structure and evolution of double minutes in diagnosis and relapse brain tumors”. Acta Neuropathologica 137 (1), 123–137. o. DOI:10.1007/s00401-018-1912-1. PMID 30267146. PMC 6338707.
- ↑ (2023. február 18.) „'Bond villain' DNA could transform cancer treatment, scientists say” 54 (10), 1527–1533. o. DOI:10.1038/s41588-022-01177-x. PMID 36123406. PMC 9534767.
- ↑ Wu S, Turner KM, Nguyen N, Raviram R, Erb M, Santini J, Luebeck J, Rajkumar U, Diao Y, Li B, Zhang W, Jameson N, Corces MR, Granja JM, Chen X, Coruh C, Abnousi A, Houston J, Ye Z, Hu R, Yu M, Kim H, Law JA, Verhaak RG, Hu M, Furnari FB, Chang HY, Ren B, Bafna V, Mischel PS (2019. november 1.). „Circular ecDNA promotes accessible chromatin and high oncogene expression”. Nature 575 (7784), 699–703. o. DOI:10.1038/s41586-019-1763-5. PMID 31748743. PMC 7094777.
- ↑ Hung, King L. (2021. december 1.). „ecDNA hubs drive cooperative intermolecular oncogene expression” (angol nyelven). Nature 600 (7890), 731–736. o. DOI:10.1038/s41586-021-04116-8. ISSN 1476-4687. PMID 34819668. PMC 9126690.
Fordítás
[szerkesztés]Ez a szócikk részben vagy egészben az Extrachromosomal DNA című angol Wikipédia-szócikk ezen változatának fordításán alapul. Az eredeti cikk szerkesztőit annak laptörténete sorolja fel. Ez a jelzés csupán a megfogalmazás eredetét és a szerzői jogokat jelzi, nem szolgál a cikkben szereplő információk forrásmegjelöléseként.
Források
[szerkesztés]- Bogenhagen DF (2012). „Mitochondrial DNA nucleoid structure”. Biochimica et Biophysica Acta (BBA) - Gene Regulatory Mechanisms 1819 (9–10), 914–20. o. DOI:10.1016/j.bbagrm.2011.11.005. PMID 22142616.
- Cara A, Reitz MS (1997. szeptember 1.). „New insight on the role of extrachromosomal retroviral DNA”. Leukemia 11 (9), 1395–9. o. DOI:10.1038/sj.leu.2400776. PMID 9305590.
- Cohen S, Yacobi K, Segal D (2003. június 1.). „Extrachromosomal circular DNA of tandemly repeated genomic sequences in Drosophila”. Genome Research 13 (6A), 113–45. o. DOI:10.1101/gr.907603. PMID 12799349. PMC 403641.
- Cohen S, Méchali M (2002. december 1.). „Formation of extrachromosomal circles from telomeric DNA in Xenopus laevis”. EMBO Reports 3 (12), 1168–74. o. DOI:10.1093/embo-reports/kvf240. PMID 12446568. PMC 1308322.
- Colosimo A, Guida V, Palka G, Dallapiccola B (2002. június 1.). „Extrachromosomal genes: a powerful tool in gene targeting approaches”. Gene Therapy 9 (11), 679–82. o. DOI:10.1038/sj.gt.3301749. PMID 12032686.
- Cummings, Donald. Extrachromosomal DNA. New York: Academic Press Inc. (1979)
- Goebel W (1970. augusztus 1.). „Studies on extrachromosomal DNA elements. Replication of the colicinogenic factor Col E1 in two temperature sensitive mutants of Escherichia coli defective in DNA replication”. European Journal of Biochemistry 15 (2), 311–20. o. DOI:10.1111/j.1432-1033.1970.tb01009.x. PMID 4926129.
- Jabaji-Hare SH, Burger G, Forget L, Lang BF (1994. május 1.). „Extrachromosomal plasmids in the plant pathogenic fungus Rhizoctonia solani”. Current Genetics 25 (5), 423–31. o. DOI:10.1007/BF00351781. PMID 8082188.
- Preer JR (1971). „Extrachromosomal inheritance: hereditary symbionts, mitochondria, chiloroplasts”. Annual Review of Genetics 5, 361–406. o. DOI:10.1146/annurev.ge.05.120171.002045. PMID 16097660.
- Shibata Y, Kumar P, Layer R, Willcox S, Gagan JR, Griffith JD, Dutta A (2012. április 1.). „Extrachromosomal microDNAs and chromosomal microdeletions in normal tissues”. Science 336 (6077), 82–6. o. DOI:10.1126/science.1213307. PMID 22403181. PMC 3703515.
- Sloan DB (2013. december 1.). „One ring to rule them all? Genome sequencing provides new insights into the 'master circle' model of plant mitochondrial DNA structure”. The New Phytologist 200 (4), 978–85. o. DOI:10.1111/nph.12395. PMID 24712049.
- Watve MM, Dahanukar N, Watve MG (2010. február 1.). „Sociobiological control of plasmid copy number in bacteria”. PLOS ONE 5 (2), e9328. o. DOI:10.1371/journal.pone.0009328. PMID 20195362. PMC 2827543.
| Az itt található információk kizárólag tájékoztató jellegűek, nem minősülnek orvosi szakvéleménynek, nem pótolják az orvosi kivizsgálást és kezelést. A cikk tartalmát a Wikipédia önkéntes szerkesztői alakítják ki, és bármikor módosulhat. |
