Mikro-RNS
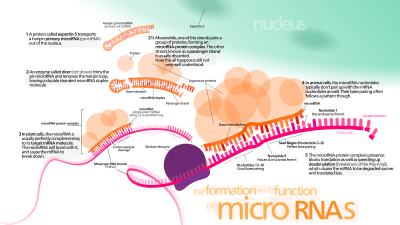

A mikro-RNS (miRNS) kis, egyszálú nemkódoló RNS 21–23 nukleotiddal.[1] Növényekben, állatokban, amőbákban és egyes vírusokban találhatók, és az RNS-csendesítésben és a poszttranszlációs génexpresszió-szabályzásban fontosak.[2][3] A miRNS-ek a megfelelő mRNS-ek komplementer szakaszaihoz kötnek,[4] majd ezeket az alábbi folyamatok egyikével csendesítik:[1][5]
- felbontás két részre,
- destabilizáció a poli(A)-farok rövidítésével vagy
- fehérjékké való transzláció csökkentése.
Állati sejtekben a miRNS-ek elsősorban az mRNS destabilizációjával hatnak.[5][6]
A miRNS-ek a kis interferáló RNS-ekhez (siRNS) hasonlítanak az RNS-interferencia (RNSi) útjában, azonban a miRNS-ek önmagukra visszahajló rövid hajtűket alkotó RNS-transzkriptum-részekből származnak, míg a siRNS-ek hosszabb kétszálú RNS-részekből.[2] A humán genom több mint 1900 miRNS-t kódolhat,[7][8] de csak mintegy 500 humán miRNS jelent bona fide miRNS-t a kézzel kezelt MirGeneDB miRNS-gén-adatbázisban.[9]
Számos emlőssejttípusban sok miRNS található:[10][11] ezek a humán és más emlősgének mintegy 60%-át célozzák.[12][13] Sok miRNS evolúciósan állandósult, vagyis fontos biológiai funkcióik lehetnek.[14][1] Például 90 miRNS-család legalább az emlősök és a halak közös őséig visszamenőleg állandósult, és ezeknek fontos funkcióik vannak, melyeket ezek egy vagy több tagjának eltávolításával végzett egérkísérletek mutattak ki.[1]
Történet[szerkesztés]
Az első miRNS-t 1993-ban fedezték fel.[15] Azonban csak 2000-ben ismerték el önálló biológiaiszabályzó-osztályként.[16][17][18][19][20] a miRNS-kutatás számos eltérő miRNS-csoportot mutatott ki eltérő sejttípusokban és szövetekben,[11][21] valamint számos szerepet a miRNS-eknek a növényi és állati fejlődésben és sok más biológiai folyamatban.[22][23][24][25][26][27][28] A nem megfelelő miRNS-expresszió számos betegségben megjelenik. Vizsgálnak miRNS-alapú terápiákat.[29][30][31][32]
Az első miRNS-t 1993-ban fedezte fel Victor Ambros, Lee és Feinbaum. Azonban további vizsgálatához Gary Ruvkun, Wightman és Ha munkája kellett.[15][33] E csoportok egymás után közöltek tanulmányokat a lin-4 génről, mely a Caenorhabditis elegans-lárva fejlődését irányítja a lin-14 gén repressziójával. Miután Lee et al. izolálták a lin-4 miRNS-t, észrevették, hogy nem fehérjekódoló mRNS-t kódol, hanem rövid nem kódoló RNS-t, melyek egyike a lin-14-mRNS 3'-transzlálatlanrégiójának több szakaszával részben komplementer.[15] Feltételezésük szerint a lin-4 a lin-14 mRNS LIN-14 fehérjévé való transzlációját gátolta, és úgy gondolták, hogy a lin-4 csak gyűrűsférgekre jellemző.
2000-ben fedezték fel a második kis RNS-t, a let-7-RNS-t, mely a lin-41-et represszálja a C. elegans későbbi fejlődési átmenetéhez.[16] Ezt sok fajban állandósultnak találták, ahhoz a feltételezéshez vezetve, hogy a let-7-RNS és más „kis temporális RNS-ek” számos állat – beleértve az embert – fejlődésének időzítését szabályozhatják.[17]
2001-ben a lin-4 és let-7 RNS-ekről kiderítették, hogy egy, a C. elegans, a Drosophila és az ember sejtjeiben jelenlévő nagy kis-RNS-osztály tagja.[18][19][20] Ennek sok RNS-e hasonlított a lin-4 és let-7-RNS-ekre, eltekintve a fejlődésidőzítés szabályzásának nem megfelelő expressziós mintával. Ez alapján feltételezték, hogy a legtöbb más szabályzó utakban vesz részt. Ekkor kezdték el a kis szabályzó RNS-eket mikro-RNS-nek nevezni.[18][19][20]
Az első miRNS-deregulációval összefüggő humán betegség a B-sejtes krónikus limfocitás leukémia volt. E betegségben a miRNS-ek egyszerre működnek tumorszupresszorként és onkogénként.[34]
Nevezéktan[szerkesztés]
A szabványos nevezéktanban a kísérletileg igazolt miRNS-ek nevüket közlés előtt kapják.[35][36] A „miR” prefixumot kötőjel, majd szám követi, ez utóbbi gyakran a megnevezés sorrenjét jelenti. Például a miR-124 korábban kapta nevét, és korábban fedezték fel a miR-456-nál. A nagybetűs „miR-” érett miRNS-t jelent, a nagybetű nélküli „mir-” pre- és pri-miRNS-t.[37] A miRNS-kódoló gének ugyane 3 betűs prefixumot kapják génnevezéktanuknak megfelelően – példák miRNS-gén-nevekre a mir-1 C. elegans és Drosophila esetén, Mir1 Rattus norvegicus esetén és MIR25 emberben.
Az egy-két nukleotid kivételével teljesen eltérő szekvenciák további kisbetűt kapnak. Például a miR-124a a miR-124b közeli rokona. Például:
- hsa-miR-181a: 5’-aacauucaACgcugucggugAgu-3’
- hsa-miR-181b: 5’-aacauucaUUgcugucggugGgu-3’
A pre-, pri-miRNS-ek és az azonos érett miRNS-hez vezető különböző helyű gének további kötőjelet és számot kapnak. Például a hsa-mir-194-1 és hsa-mir-194-2 azonos érett miRNS-t (hsa-miR-194) hoz létre, de eltérő genombeli helyről származnak.
A faj hárombetűs prefixummal van jelölve – például a hsa-miR-124 humán, az oar-miR-123 bárány-miRNS. További prefixumok például a vírusokat jelölő v vagy a Drosophilát jelölő d.
Ha két érett miRNS azonos pre-miRNS ellentétes karjairól származik és nagyjából ugyanannyi van belőlük, -3p vagy -5p utótagot kapnak (korábban s (szenz) vagy as (antiszenz) utótaggal is jelölték). Azonban a hajtű egyik karjáról származó miRNS általában sokkal nagyobb mennyiségben van jelen, mint a másikról származó,[2] ez esetben a nevet követő csillag a kisebb mennyiségben a másik karról keletkező miRNS-t jelenti. Például a miR-124 és a miR-124* azonos pre-miRNS hajtűvel rendelkeznek, de sokkal több a miR-124.
Célpontok[szerkesztés]
A növényi miRNS-ek közel tökéletesen párosodnak mRNS célpontjaikkal, a génrepressziót a célpont bontásával elérve.[22][38] Ezzel szemben az állati miRNS-ek cél-mRNS-üket csupán 6–8 nukleotiddal (magrégió) ismerik fel a kezdetükön,[12][39][40] ami kevés a cél-mRNS-bontás elindításához.[4] A kombinációszabályzás fontos az állatok miRNS-szabályzásában.[4][41] Egy adott miRNS több száz mRNS-célponttal rendelkezhet, és egy célpontot több miRNS szabályozhatja.[13][42]
Adott miRNS által célzott egyedi mRNS-ek átlagos száma becsléstől függően eltér,[43] de több megközelítés szerint az emlős-miRNS-ek számos egyedi célponttal rendelkezhetnek. Például a gerincesekben állandósult miRNS-eknek átlagosan mintegy 400 állandó célpontjuk van.[13] Ugyanígy kísérletek szerint egy miRNS több száz egyedi mRNS stabilitását csökkentheti.[44] Más kísérletek szerint egy miRNS több száz fehérje termelését represszálhatja, de gyakran ez enyhe – kevesebb mint 2-szeres.[45][46]
Szintézis[szerkesztés]

A miRNS-gének akár 40%-a más gének intronjaiban vagy akár exonjaiban lehet.[47] Ezek gyakran szenz elrendezésben vannak,[48][49] így szabályzásuk a gazdagénekével együtt történik.[47][50][51]
A DNS-templáttal nem ér véget a miRNS-érés: a humán miRNS-ek 6%-a helyspecifikus RNS-szerkesztést mutat (izomiR) a DNS által kódolttól eltérő termékek létrehozásához. Ez a genom által kódolthoz képest növeli a miRNS-hatás diverzitását.
Transzkripció[szerkesztés]
A miRNS-géneket általában az RNS-polimeráz II (Pol II) írja át.[52][53] Gyakran köt a szekvenciához közeli promotert, mely a pre-miRNS hajtűhurka. Az így létrejött átirat 5'-végén speciálisan módosult nukleotid jön létre, poli(A)-farkat kap,[52][48] és leválik. Az állati miRNS-ek kezdetben egy mintegy 80 nukleotidos RNS-törzskörben keletkeznek, mely egy néhány száz nukleotidos miRNS-prekurzor, a pri-miRNS része.[52][48] Ha egy törzskörprekurzor van a 3'-UTR-ben, az átirat pri-miRNS és mRNS is lehet.[48] Az RNS-polimeráz III (Pol III) néhány miRNS-t átír, különösen a korai Alu-szekvenciákkal rendelkező, a transzfer- és az emlős-szélesszórtismétlődés (MWIR) promotereket.[54]
Magi feldolgozás[szerkesztés]

Egy pri-miRNS 1–6 miRNS-prekurzort tartalmazhat. Ezek mintegy 70 nukleotidból állnak, és a hatékony feldolgozáshoz szükséges szekvenciák veszik körül.
A kétszálú RNS-szerkezetet (dsRNS) a pri-miRNS-ek hajtűiben a DiGeorge-szindróma, 8. fontos régió (DGCR8, gerinctelenekben Pasha) veszi körül, mely nevét a DiGeorge-szindrómával való kapcsolatáról kapta. Ez az RNS-vágó Drosha enzimmel asszociál mikroprocesszor-komplexet alkotva.[55][56] E komplexben a DGCR8 a katalitikus RNáz III-domént hajtűk pri-miRNS-ekről való leválasztására használja mintegy 11 nukleotiddal a hajtűalap előtt (egy helikális dsRNS-fordulattal a törzsön belül).[57][58] A termék kétnukleotidos többlettel rendelkezik a 3'-végen, 3'-hidroxil- és 5'-foszfátcsoportjai vannak. Gyakran ezt pre-miRNS-nek nevezik. Ismertek a pre-miRNS utáni fontos szekvenciamotívumok.[59][60][61]
Az intronokból közvetlenül a mikroprocesszor-komplex megkerülésével leválasztott pre-miRNS-ek a mirtronok.[62] Ismertek mirtronok a Drosophilában, a C. elegansban és emlősökben.[63][64]
A pre-miRNS-ek 16%-át módosíthatja a magi RNS-szerkesztés.[65][66][67] A leggyakoribb az RNS-re ható adenozin-deaminázok (ADAR) általi módosítás, melyek az adenozint inozinná (A→I) alakítják. Az RNS-szerkesztés leállíthatja a magi folyamatot (például a pri-miR-142-ben a Tudor-SN ribonukleáz általi bomláshoz vezet) és a későbbi folyamatokat, például a citoplazmatikus miRNS-feldolgozást és a célspecificitást (például a miR-376 magrégiójának változtatásával a központi idegrendszerben).[65]
Magi kivitel[szerkesztés]

A pre-miRNS-hajtűk a magból a nukleocitoplazmatikus szállító exportin-5-öt tartalmazó folyamatban távoznak. E fehérje a karioferinek családjának tagja, és felismeri a Drosha által megmaradt kétnukleotidos többletet a per-miRNS-hajtű 3'-végén. Az exportin-5-mediált transzport energiafüggő, a Ran fehérjéhez kötött guanozin-trifoszfátot használ.[68]
Citoplazmatikus feldolgozás[szerkesztés]
A citoplazmában a pre-miRNS hajtűt a Dicer RNáz III bontja.[69] Ez az endoribonukleáz a hajtű 5'- és 3'-végeivel kölcsönhat,[70] és elvágja a 3'- és 5'-karokat összekötő kapcsolatokat, mintegy 22 nukleotid hosszú nem tökéletes miRNS:miRNS* duplexet adva.[69] A teljes hajtűhossz és a körméret befolyásolják a Dicer hatékonyságát. Hogy a miRNS:miRNS* pár nem tökéletes, befolyásolja az elválást is.[69][71] Egyes G-gazdag pre-miRNS-ek G-kvadruplex szerkezetet is alkothatnak a törzskörszerkezet miatt – például a humán pre-miRNS 92b a Dicer-mediált bontásnak ellenálló G-kvadruplexet alkot a citoplazmában.[72] Bár a duplex bármely szála funkcionális miRNS-ként működhet, csak egy szál kerül az RNS-indukált csendesítőkomplexbe, ahol a miRNS és mRNS célja kölcsönhatnak.
Míg a legtöbb miRNS a sejtben van, a keringő vagy extracelluláris miRNS-ek sejten kívül is megtalálhatók, beleértve számos biológiai folyadékot és sejtkultúraközeget.[73][74]
Szintézise növényekben[szerkesztés]
A miRNS-szintézis növényekben főleg a magi feldolgozásban és kivitelben tér el az állatokétól. Nem két eltérő enzim bontja a miRNS-eket egyszer a magban és egyszer azon kívül, hanem mindkét bontást a Dicer-like1 (DL1) enzim végzi. Ez csak a növényi sejtek magjában expresszálódik, így feltehetően mindkét reakció a magban történik. A növényi miRNS:miRNS* duplexek magból való kivitele előtt a 3'-végeket az RNS-metiltranszferáz Hua-erősítő 1 (HEN1) metilálja. Ezt az exportin-5-homológ Hasty (HST) szállítja a magból a citoplazmába, ahol kettéválnak, az érett miRNS a RISC-be kerül.[75]
RNS-indukált csendesítőkomplex[szerkesztés]
Az érett miRNS aktív RNS-indukált csendesítőkomplex (RISC) része, ahol a Dicer és sok kapcsolódó fehérje van.[76] A RISC-et nevezik mikro-RNS-ribonukleoprotein-komplexnek (miRNP) is.[77] A miRNS-sel rendelkező RISC-et nevezik miRISC-nek is.[78]
A pre-miRNS Dicer általi feldolgozása feltehetően a duplex kibontásával együtt történik. Általában egy szál van a miRISC-ben, melyet termodinamikai instabilitás és a másik szálnál gyengébb 5'-végi bázispárosodás alapján választ ki.[79][80][81] A törzskör helye is befolyásolhatja a szálválasztást.[82] A másik, kisebb koncentrációja miatt „utasszálnak” nevezett szálat *-gal jelölik, és általában bomlik. Egyes esetekben a duplex mindkét tagja életképes, és eltérő mRNS-eket célzó miRNS.[83]
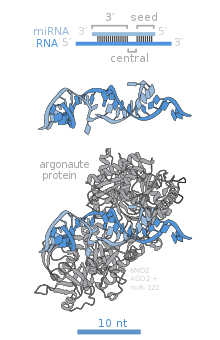
Az Argonaute (Ago) fehérjecsalád tagjai fontosak a RISC-működéshez. Ezek a miRNS-indukált csendesítéshez szükségesek, és két állandósult RNS-kötő helyük van: egy egyszálú 3'-véghez kötő PAZ és egy PIWI domén, mely szerkezetileg a ribonukleáz H-ra hasonlít, és az irányítószál 5'-végével kölcsönhat. Ezek az érett miRNS-hez kötnek és a cél-mRNS-sel való kölcsönhatáshoz fordítják. Egyes tagjai, például az AGO2 a céltranszkriptumokat közvetlenül bontják, és más fehérjéket is aktiválhatnak a transzlációs represszióhoz.[84] A humán genomban 8 tagja van jelen, melyek szekvenciahasonlóság alapján 2 családba tartoznak, ezek az AGO (4 minden emlőssejtben jelenlévő taggal, nevük emberben E1F2C/hAgo) és a PIWI (csíravonal- és vérképző őssejtekben).[77][84]
További RISC-komponensek például a TRBP (humán immunhiányvírus (HIV)-transzaktiváló válasz-RNS (TAR)-kötő fehérje),[85] a PACT (interferonindukált fehérjekináz aktivátora), az SMN-komplex, a törékeny X-mentálisretardáció-fehérje (FMRP), a Tudor staphylococcalis nukleázdomén-tartalmazó fehérje (Tudor-SN), a feltételezett DNS-helikáz MOV10 és az RNS-felismerőmotívum-tartalmazó fehérje TNRC6B.[68][86][87]
Csendesítés módja és szabályzókörök[szerkesztés]
A géncsendesítés történhet mRNS-bontással vagy mRNS-transzláció megakadályozásával. Például a miR16 számos instabil mRNS, például a TNF α vagy a GM-CSF 3'UTR-jában lévő AU-gazdag elemre komplementer szekvenciát tartalmaz.[88] Teljes miRNS:cél-mRNS komplementaritás esetén az Ago2 lebonthatja az mRNS-t, közvetlen mRNS-bomlást okozva. Komplementaritás nélkül a csendesítés transzlációakadályozással történik.[44] A miRNS és cél-mRNS-e közti kapcsolat egyszerű negatív cél-mRNS-szabályzáson alapulhat, de gyakori a „koherens előrecsatolási ciklus”, a „mutuális negatív visszacsatolási ciklus” és a „pozitív vissza-/előrecsatolási ciklus”. Egyes miRNS-ek a transzkripció, a transzláció és a fehérjestabilitás sztochasztikus eseményei miatti véletlen génexpresszió-változások pufferei. Ezt általában negatív vissza- vagy inkoherens előrecsatolással érik el a fehérje mRNS-transzkripciótól való leválasztásával.
Átfordulás[szerkesztés]
Az érett miRNS átfordulása szükséges a miRNS-expressziós profilok gyors változásához. A miRNS-érés során az Argonaute általi felvétel szabilizálhatja az irányítószálat, míg az ellentétes (*) szál általában megszűnik. A „használd vagy veszítsd” stratégiában az Argonaute általában a sokcélú miRNS-eket tartja meg a kevéscélúakkal vagy cél nélküliakkal szemben, lebontva a nem célzó molekulákat.[89]
Az érett miRNS-ek lebontása a C. elegansban az XRN2 5'–3' exoribonukleáz, más néven Rat1p által történik.[90] Növényekben az SDN (kis-RNS-bontó nukleáz) család tagjai ellentétes (3'–5') irányba bontják. Hasonló állati enzimek is vannak, de szerepük nem tisztázott.[89]
Számos miRNS-módosulás befolyásolja a miRNS-stabilitást. Az Arabidopsis thalianán végzett munka alapján az érett növényi miRNS-eket a 3'-vég metilálása stabilizálja. A 2'-O-konjugált metilcsoportok megakadályozzák az uracil uridiltranszferázok általi hozzáadását, mely összefügghet a miRNS-bomlással. Azonban az uridiláció meg is védhet egyes miRNS-eket, ennek következményei nem teljesen tisztázottak. Egyes állati miRNS-ek uridilációjáról beszámoltak. A növényi és állati miRNS-ek adenin 3'-véghez való adásával is befolyásolhatók. Egy A-többlet a hepatitis C-ben fontos emlős-miR-122-n stabilizálja a molekulát, az A-végű növényi miRNS-ek lassabban bomlanak.[89]
Sejtfunkciók[szerkesztés]

A miRNS-ek funkciója a génszabályzásban fontos. A miRNS komplementer egy vagy több mRNS részeihez. Az állati miRNS-ek általában a 3'-nemtranszláltrégióval, a növényiek általában a kódoló részekkel komplementerek.[92] A közel tökéletes bázispárosodás segíti az RNS-lebontást.[93] Ez a növényi miRNS-ek elsődleges módja.[94] Állatokban az egyezés nem tökéletes.
A részben komplementer miRNS-ek a magrégiókat (2–7. nukleotid) használják fel a célpont felismerésére,[12][39] ennek teljesen komplementernek kell lennie.[95] Az állati miRNS-ek gátolják a cél-mRNS transzlációját[96] (ez növényekben jelen van, de ritkább).[94] A részben komplementer miRNS-ek felgyorsíthatják a dezadenilációt, korábbi mRNS-bomlást okozva.[97] Bár a miRNS-célzott mRNS bontása ismert, hogy a transzlációs repressziót mRNS-bomlás, transzlációgátlás vagy ezek együtt okozzák-e, nem. 2012-benz zebradániókban miR-430-cal és bantam-miRNS-sel és miR-9-cel Drosophila-sejttenyészetben végzett kutatás szerint a transzlációs repressziót az iniciáció zavarása okozza dezadenilációtól függetlenül.[98][99]
A miRNS-ek okozhatnak továbbá hisztonmódosulást és a promoterek metilációját, melyek a célgének expresszióját befolyásolják.[100][101]
9 miRNS-hatásmechanizmus ismert és szerepel egyesített matematikai modellben:[91]
- Cap-40S-iniciációgátlás;
- 60S-csatlakozás-gátlás;
- elongációgátlás;
- riboszómaleválasztás (korai leállítás);
- kotranszlációs naszcensfehérje-bontás;
- P-testekbe való szekvesztráció;
- mRNS-bontás (destabilizáció);
- mRNS-leválasztás
- transzkripciós gátlás miRNS-mediált kromatinátrendezéssel és géncsendesítéssel.
Gyakran nem megkülönböztethetők e mechanizmusok kísérleti adatok alapján állandó reakciósebességekről, de dinamikájuk és kinetikai lenyomatuk eltér.[91]
A növényi miRNS-ekkel szemben az állatiak eltérő géneket céloznak.[39] Azonban a minden sejtre jellemző funkciókat érintő géneknek kevesebb miRNS-célhelyük van, és a miRNS-ek általi célzás elkerülésére választódnak ki.[102] Erős a korreláció az ITPR gén szabályzása, a mir-92 és a mir-19 közt.[103]
A dsRNS aktiválhatja is a génexpressziót (RNSa). A promotereket célzó dsRNS-ek erősen aktiválhatják a gének transzkripcióját. Ezt humán sejtekben szintetikus dsRNS-ekkel, kis aktiváló RNS-ekkel (saRNS) mutatták ki,[104] de endogén miRNS-nél is igazolták.[105]
A miRNS-ek és a komplementer gén- és homológ pszeudogénszakaszok közti kölcsönhatások a paralóg gének expressziós szintje közti csatorna lehetnek. E miRNS-ek, a „versenyző endogén RNS-ek” (ceRNS) „miRNS-válaszelemekhez” kötnek a génekben és a pszeudogénekben, és a nem kódoló DNS állandóságát is indokolhatják.[106]
Vannak miRNS-ek a sejten kívül (keringő miRNS),[107] és ezeket befolyásolják a sejten belüli miRNS-ek.[108] A keringő miRNS-ek testnedvekbe, például a vérbe és az agy-gerincvelői folyadékba áramolnak, és számos betegség biomarkerei lehetnek.[107][109] Több kutatás szerint az exoszómákban lévő mRNS-ek szerepet játszhatnak a beágyazódásban, adhéziót vonhatnak el a trofoblaszt és az endometrium közt vagy erősíthetik azt az adhézió/invázió génjeinek erősítésével vagy gyengítésével.[110]
Ezenkívül a miR-183/96/182 fontos lehet a cirkadián ritmusban Zhou et al. szerint.[111]
A miRNS-eket kódoló szakaszok gyakran gének intronjaiban vannak, de ismertek gének közt kódoltak is.[112]:13
Evolúció[szerkesztés]
A miRNS-ek nagymértékben állandók növényekben és állatokban, és feltehetően a génszabályzás fontos, ősi részei.[113][114][115][116][117] Bár a miRNS-út központi elemei állandók a növények és állatok körében, a miRNS-ek egymástól nagyrészt függetlenül jelentek meg eltérő elsődleges hatásmóddal.[118][119]
A miRNS-ek hasznos filogenetikai markerek alacsony evolúciós sebességük miatt.[120] Szabályzó mechanizmuskénti eredetük korábbi RNSi-ből származik, mely korábban exogén örökítőanyag, például vírusok elleni védelem volt.[121] Eredetük tehette lehetővé a morfológiai újításokat, és a génexpresszió specifikusabbá és finomhangolhatóbbá tételével lehetővé tette az összetett szervek[122] és Peterson et al. szerint a komplex élet megjelenését.[117] Rapid bursts of morphological innovation are generally associated with a high rate of microRNA accumulation.[120][122]
Az új miRNS-ek több módon jöhetnek létre. Létrejöhetnek nem kódoló DNS-szakaszokból véletlenszerű hajtűk kialakulásával vagy meglévő miRNS-ek duplikációjával vagy módosulásával.[123] Létrejöhetnek továbbá fehérjekódoló szakaszok invertált duplikációjával, lehetővé téve a visszahajtott hajtűszerkezetet.[124] Az új miRNS-ek evolúciós sebessége hasonló a nem kódoló DNS más részeihez, vagyis semleges sodródással történik, azonban a régebbiek ritkábban változnak – gyakran kevesebb mint 1-szer százmillió évente –,[117] vagyis a miRNS funkciószerzése után tisztító szelekció jellemzi.[123] A miRNS-gén egyes szakaszait eltérő evolúciós nyomás jellemzi, ahol a funkcióhoz és feldolgozáshoz fontos szakaszok jobban megőrződnek.[125] Ekkor ritkán tűnik el egy állat genomjából a miRNS,[117] de az újabb, így feltehetően nem működő miRNS-ek gyakran tűnnek el.[123] Az Arabidopsis thaliana esetén a miRNS-gének változása 1,2–3,3 gén millió évenként.[126] Ez értékes filogenetikai markerré teszi, és különböző filogenetikai problémák, például az ízeltlábúak kapcsolatai megoldására használható.[127] Azonban a miRNS-ek gyakran kevéssé korrelálnak a filogenetikával, és lehetséges, hogy filogenetikai egyezésük csekély mintát tükröz.[128]
A miRNS-ek a legtöbb eukarióta genomjában jelen vannak a barnamoszatoktól[129] az állatokig. Azonban ezek működésében és feldolgozásuk módjában való különbségek alapján az állatokban és a növényekben függetlenül jelentek meg a miRNS-ek.[130]
A Mnemiopsis leidyi genomja[131] feltehetően nem tartalmaz észlelhető miRNS-eket, Droshát vagy Pashát, melyek a miRNS-szintézishez fontosak. Ez az egyetlen ismert állat Drosha nélkül. A miRNS-ek fontosak a génexpresszió-szabályzásban a bordásmedúzák és a korongállatkák kivételével minden állatban.[132]
Az összes fajban több mint 5000 különböző miRNS-t azonosítottak 2010 márciusáig.[133] Bár széles funkciójú rövid (50 bázispártól több százig) RNS-ek megtalálhatók baktériumokban, bennük nincs valódi miRNS.[134]
Az amőbákban lévő miRNS-ek Edelbroek et al. 2024-es tanulmánya szerint a többsejtűség eredetét is megmagyarázhatják.[135]
Kísérleti észlelés és befolyásolás[szerkesztés]
Míg a kutatók a fiziológiai és patológiai folyamatokban jelenlévő miRNS-expresszióra fókuszáltak, számos miRNS-izolációhoz kapcsolódó technikai jellemző jelent meg. A tárolt miRNS-ek stabilitása kérdéses.[74] A miRNS-ek az mRNS-nél részben hosszuk, részben a mindenhol jelenlévő ribonukleázok miatt könnyebben bomlanak. Ez a minták jégen való hűtését és ribonukleázmentes felszerelés használatát igényli.[136]
A miRNS-expresszió kétlépéses polimeráz-láncreakcióban határozható meg módossított RT-PCR-rel és kvantitatív PCR-rel. Ennek változatai abszolút vagy relatív meghatározást érnek el.[137] A miRNS-ek hibridizálhatók mikroarray-kre, filmekre vagy chipekre több száz vagy ezer miRNS-célponttal, így relatív miRNS-szintek meghatározhatók eltérő mintákban.[138] A miRNS-ek felfedezhetők és jellemezhetők nagy átvitelű szekvenálási módszerekkel (miRNS-szekvenálás).[139] A miRNS-ek aktivitása kísérletileg gátolható zártnukleinsav- (LNS) vagy morfolino-[140][141] vagy 2’-O-metil-RNS-oligonukleotiddal.[142] Adott miRNS komplementer antagomirrel csendesíthető. A miRNS-érést több helyen gátolhatja sztérikusan akadályozó oligomer.[143][144] Az mRNS-átirat miRNS-célpontját is akadályozhatja sztérikusan blokkoló oligonukleotid.[145] A miRNS in situ észleléséhez LNS-[146] vagy morfolinopróbák használhatók.[147] Az LNS zárt konformációja nagyobb hibridizációt, szenzitivitást és szelektivitást okoz, ideálissá téve rövid miRNS-ek észlelésére.[148]
A miRNS-ek nagy átvitelű meghatározása nagy hibahatárú az mRNS-ekkel szemben a metodológiai problémák miatt nagyobb variancia miatt. Az mRNS-expressziót így gyakran miRNS-hatásokkal vizsgálják.[149] Az adatbázisok mRNS- és miRNS-adatok párosítására használhatók, melyek a miRNS-célokat alapszekvencia alapján meghatározhatják.[150][151] Bár ezt általában a fontos miRNS-ek észlelése után végzik például nagymértékű expresszió miatt, az mRNS- és miRNS-expressziós információt integráló eszközöket javasoltak.[152][153]
Humán és állatbetegségek[szerkesztés]
Ahogy a miRNS az eukarióta sejtek normál működésében fontos, úgy diszregulációja betegségekkel függ össze. Egy kézzel vezetett nyilvános adatbázis, a miR2Disease a miRNS-diszregulációja és humán betegségek kapcsolatait írja le.[154]
Örökletes betegségek[szerkesztés]
A miR-96 magrégiójának mutációja örökletes fokozatos hallásvesztést okoz.[155]
A miR-184 magrégiójának mutációja örökletes keratoconust okoz anterior poláris kataraktával.[156]
A miR17–92 csoport deléciója csontváz- és növekedési hibákat okoz.[157]
Rák[szerkesztés]
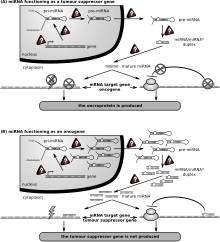
Az első miRNS-deregulációval összefüggésbe hozott betegség a krónikus limfocitás leukémia.[158] Sok más miRNS is összefügg a rákkal, ennek megfelelően „onkomirnak” nevezik.[159] Malignus B-sejtekben a miRNS-ek a B-sejt-fejlődéshez fontos utakban, például a B-sejt-receptor- (BCR) jelzésben, a B-sejt-vándorlásban/adhézióban, a sejtközi kölcsönhatásokban és az immunglobulin-termelésben és -osztályváltásban vesznek részt. A miRNS-ek a B-sejtek érését, pre-, marginális, follikuláris, B1-, plazma és memória B-sejtek létrehozását befolyásolja.[160]
A miRNS-ek expressziós szintje használható prognózisra. Nem kissejtes tüdőrákban a miR-324a alacsony szintje alacsony túlélési arányt jelezhet előre.[161] A magas miR-185- vagy alacsony miR-133b-szint összefügghet colorectalis rákban az áttéttel és az alacsony túlélési aránnyal.[162]
Ezenkívül specifikus miRNS-ek összefügghetnek a colorectalis rák bizonyos hisztológiai altípusaival. Például a miR-205 és a miR-373 expressziója mucinos és mucintermelő ulceratívcolitis-asszociált vastagbélrákban nagyobb, de nem a sporadikus mucinmentes vastagbél-adenokarcinómában.[163] In vitro tanulmányok szerint a miR-205 és -373 funkcionálisan eltérő mucinasszociált neoplasztikus előrehaladást mutathat bélepitélsejtekben.[163]
A májráksejt-proliferációt a miR-21 és a MAP2K3 tumorszupresszor kölcsönhatása okozhatja.[164] Az optimális rákkezelés a kockázatelemzésnek megfelelő terápiát igénylő betegek azonosítását igényli. A kezdeti kezelésre való gyors válasz esetén előnyös lehet a nem teljes kezelési rend. A sejtmentes keringő miRNS-ek (cimiRNS) nagyon stabilak a vérben, rákban túlexpresszálódnak, és a diagnosztikai laboratóriumban mérhetők. Klasszikus Hodgkin-limfóma esetén a plazma-miR-21, -miR-494 és -miR-1973 válaszbiomarkerek.[165] A keringő miRNS-ek segíthetik a döntéshozatalt és a pozitronemissziós tomográfia és a számítógépes tomográfia értelmezését. Ezek a betegségre való válasznál és a relapszusészlelésnél használhatók.
A miRNS-ek használhatók az eltérő rákok kezelésében eszközként vagy célként.[166] A miR-506 több tanulmány szerint tumorantagonista. Számos méhnyakrákminta kisebb miR-506-expressziót mutatnak. Ezenkívül a miR-506 a méhnyakráksejtek apoptózisát segíti közvetlen hedgehog-úti transzkripciós faktora, a Gli3 révén.[167][168]
DNS-javítás és rák[szerkesztés]
Sok miRNS közvetlenül célozhat és gátolhat sejtciklusgéneket a sejtproliferáció szabályzásához. A tumorkezelés új stratégiája a tumorsejt-proliferáció gátlása a hibás miRNS-út megjavításával a tumorokban.[169] A rákot a mutációk számának növekedése okozza DNS-károsodás vagy kijavítatlan DNS-replikációs hibák miatt.[170] A DNS-javítási hibák mutációk felhalmozódását okozza, amely rákhoz vezethet.[171] Számos DNS-javító gént miRNS-ek szabályoznak.[172]
A DNS-javító gének csíravonal-mutációi csak a vastagbélrákesetek 2–5%-át okozzák.[173] Azonban a DNS-javítási hibákat okozó megváltozott miRNS-expressziót gyakran hozzák összefüggésbe rákokkal, és fontos kauzális tényező lehet. Az MLH1 DNS-javító fehérje csökkent expressziójával rendelkező 68 sporadikus vastagbélrákból a legtöbb a CpG sziget epigenetikai metilációja miatt jelent meg.[174] Azonban a sporadikus vastagbélrákokban előforduló MLH1-hiányosságok 15%-ának oka az MLH1-represszor miR-155 túlexpressziója.[175]
A glioblastomák 29–66%-ában[176][177] a DNS-javítás az MGMT gén epigenetikai metilációja miatt csökkent, amely az MGMT-expressziót csökkenti. Azonban a glioblastomák 28%-ában az MGMT hibás, de az MGMT-promoter nem metilált.[176] A metilált MGMT-promoter nélküli glioblastomákban a miR-181d-szint inverz korrelációt mutat az MGMT-expresszióval, és a miR-181d közvetlen célpontja az MGMT-mRNS 3'UTR.[176] Így a glioblastomák 28%-ában a megnövekedett miR-181d-és kisebb MGMT-expresszió ok lehet.
A HMGA1a, HMGA1b és HMGA2 HMGA fehérjék fontosak a rákban, és e három fehérje expresszióját miRNS-ek szabályozzák. A HMGA-expresszió differenciált felnőtt szövetben csekély, de sok rákban emelkedett. E fehérjék mintegy 100 aminosavas moduláris ffehérjék, három erősen pozitív töltésű régiójuk (AT-horog) van, melyek az AT-gazdag DNS kis bemélyedéséhez kötnek. A humán tumorok, beleértve a pajzsmirigy-, a prosztata-, a méhnyak-, a colorectalis, a hasnyálmirigy- és a petefészekrákot erős HMGA1a- és HMGA1b-növekedést mutatnak.[178] A HMGA1-et limfoid sejtekben tartalmazó transzgenikus egerek agresszív limfómát mutatnak, vagyis a magas HMGA1-expresszió összefügg a rákokkal, és a HMGA1 onkogén.[179] A HMGA2 specifikusan célozza az ERCC1 promoterét, csökkentve e DNS-javító gén expresszióját.[180] Egy kutatásban mind a 47 vastagbélrákban alacsony volt az ERCC1-expresszió (de a HMGA2 szerepe ebben ismeretlen).[181]
Az egypontos nukleotid-polimorfizmusok (SNP) befolyásolhatják a 3'UTR-ek miRNS-kötését például a hsa-mir181a és a hsa-mir181b és a CDON tumorszupresszor esetén.[182]
Szívbetegség[szerkesztés]
A miRNS-funkció szívben játszott szerepét a miRNS-ek érésének feltételes gátlásával vizsgálták egérszívben. Ez alapján a szívfejlődésben fontosak a miRNS-ek.[183][184] A miRNS-expresszióprofil-tanulmányok alapján az egyes miRNS-ek expressziója megváltozik a beteg humán szívekben, így szerepet játszhatnak a kardiomiopátiában.[185][186][187] Továbbá egyes miRNS-ekkel kapcsolatos állattanulmányok az egyes miRNS-ek szerepét is kimutatták szívfejlődés közben és patológiás esetekben, beleértve a kardiogenezishez, a hipertrófiás válaszhoz és a szívvezetéshez szükséges tényezőket.[184][188][189][190][191][192] A cardiovascularis betegségekben a miRNS expressziója felhasználható diagnózisra, prognózisra vagy kockázatelemzésre.[193] Az állatmodellek miRNS-ei összefüggnek a koleszterinmetabolizmussal és -szabályzással.
miR-712[szerkesztés]
Az egér-miR-712 az atherosclerosis potenciális biomarkere.[194] A nem lamináris véráramlás összefügg az atherosclerosis kialakulásával, mivel az endotél sejtek mechanoreceptorai érzékelik a d-áramlás szakítóerejét.[195] Számos proaterogén gén, beleértve a mátrix-metalloproteinázokat, a d-áramlás erősíti,[195] proinflammatiós és proangiogén jeleket küldve. Ezeket ligált egérartériákban is megfigyelték a d-áramlás utánzására. 24 órán belül a meglévő éretlen mir-712 érett miR-712-t hozott létre, vagyis a miR-712 áramlásérzékeny.[195] Ezzel együtt a miR-712 erősödött a természetes d-áramlásnak kitett endotél sejtekben a nagyobb görbületű aortaívben.[195]
Eredet[szerkesztés]
A miR-712 pre-mRNS-szekvenciáját a riboszomális RN45s gén hozza létre az ITS2-nél.[195] Az XRN1 az ITS2-t az RN45S feldolgozása során bontó exonukleáz.[195] Az XRN1 d-áramlás alatti redukciója megnöveli így a miR-712-szintet.[195]
Mechanizmus[szerkesztés]
A miR-712 a szöveti metalloproteináz-gátló 3-at (TIMP3) célozza.[195] A TIMP-ek az extracelluláris mátrixot bontó mátrix-metalloproteinázok (MMP) aktivitását befolyásolják. Az artériás ECM főleg kollagénből és elasztinból áll, támasztva az artériákat és lehetővé téve visszaállásukat.[196] Ezek fontosak a vaszkuláris inflammatio és permeabilitás szabályzásában, melyek fontosak az atherosclerosis kialakulásában.[197] Az endotél sejtek expresszálta TIMP3 az egyetlen ECM-kötött TIMP.[196] Expressziójának csökkenése növeli az ECM-bomlást d-áramlás esetén. Ennek megfelelően a pre-miR712 gátlása növeli a TIMP3-expressziót turbulens áramláskor is.[195]
A TIMP3 csökkenti ezenkívül a proinflammatiós TNFα expresszióját turbulens áramláskor is.[195] Ennek aktivitását TACE-expresszióval mérték a vérben. A TNFα csökkent a miR-712 gátlásakor vagy TIMP3-túlexpressziókor,[195] tehát a miR-712 és a TIMP3 szabályozzák a TACE-aktivitás turbulens áramláskor.
Az anti-miR-712 megakadályozza a d-áramlás-indukált miR-712-expressziót és növeli a TIMP3-expressziót.[195] Az anti-miR-712 gátolja emellett a vaszkuláris hiperpermeabilitást, csökkentve az atherosclerotikus léziófejlődést és az immunsejt-infiltrációt.[195]
Humán homológ mikro-RNS-205[szerkesztés]
A humán miR-712-homológot az RN45s homológ génben találták, mely egerekhez hasonló miRNS-eket tartalmaz.[195] A humán miR-205 of hasonlít az egér-miR-712-höz, és sok gerincesben jelen van.[195] A miR-205 és a miR-712 sejtkommunikációs céljai több mint 50%-a, beleértve a TIMP3-at, állandósult.[195]
A d-áramlás csökkenti az XRN1-expressziót emberben az egérendotélsejtekhez hasonlóan, így gyakori lehet az XRN1 szerepe.[195]
Vesebetegség[szerkesztés]
A FoxD1-eredetű egérvese-progenitorsejtek célzott Dicer-deléciója összetett vesefenotípust okozott tágult nefronprogenitorokkal, kevesebb reninsejttel, simaizom-artériával, progresszív mezangiumvesztéssel és glomeruláris aneurizmákkal.[198] Teljes transzkriptomos elemzés megnövekedett proapoptotikus Bcl2L11- (Bim) expressziót és a p53-út diszregulációját mutatta nagyobb p53-effektorgénnel, beleértve a Baxot, a Tp53inp1-et, a Junt, az Mmp2-t és az Arid3a-t. A p53-szint változatlan, vagyis a FoxD1 stromalis miRNS-ei közvetlenül represszálják a p53-effektorokat. Vonalkövetéssel és fluoreszcenciaaktivált sejtrendezéssel a miRNS-kimutatás nemcsak meghatározta a vaszkuláris fejlődéshez szükséges miRNS-ek transzkripcióját, hanem a vesefenotípust valószínűleg hiányukban moduláló fontos miRNS-eket is kimutatott. Ilyen miRNS-ek például a Bcl2L11-szabályzó miR-10a, -18a, -24, -30c, -92a, -106a, -130a, -152, -181a, -214, -222, -302a, -370 és -381, valamint a p53-effektor-szabályzó miR-15b, -18a, -21, -30c, -92a, 106a, -125b-5p, -145, -214, -222, -296-5p és -302a. Ennek megfelelően ektópiás apoptózist találtak a FoxD1-eredetű progenitorvonalban, ami a vese stromális miRNS-ei fontosságát mutatja meg ismét a sejthomeosztázisban.[198]
Idegrendszer[szerkesztés]
A miRNS-ek szabályozzák az idegrendszer működését és fejlődését.[199] A szinapszisfejlődés eltérő szakaszaiban eltérő miRNS-ek fontosak: a dendritogenezisben és szinapszisképzésben a miR-132, a miR-134 és a miR-124[200] a szinapszisérésben a miR-134 és a miR-138 érintettek.[201] A miRNS-képzés megszüntetése egerekben a Dicer csendesítésével patológiás eredeményeket, például csökkent neuronméretet és motoros rendellenességeket okozott a striatalis[202] és neurodegenerációt az előagyi neuronokban való csendesítéskor.[203] Egyes tanulmányok megváltozott miRNS-expressziót találtak Alzheimer-kór,[204] szkizofrénia, bipoláris zavar, major depresszió és szorongás esetén.[205][206][207]
Stroke[szerkesztés]
A Center for Disease Control and Prevention szerint a stroke az egyik vezető halál- és hosszútávúfogyaték-ok Amerikában. Az esetek 87%-a ischaemiás, mely az agyi artéria okklúziójából ered. Így az agy nem jut elég tápanyaghoz, például oxigénhez és glükózhoz, és nem távoznak onnan a fölös anyagok, például a szén-dioxid.[208][209] A miRNS-ek a cerebralis ischaemia patogenezisében lévő, például a gyulladásban, az angiogenezisben és az apoptózisban fontos gének poszttranszlációs csendesítésével működnek.[210]
Függőség[szerkesztés]
A miRNS-ek génexpresszióban játszott fontos szerepe jelentős a függőségek, különösen az alkoholizmus esetén.[211] A krónikus alkoholfüggőség állandó agyi funkcióváltozásokat okoz, melyet részben a génexpresszió változásai okoznak.[211] Számos későbbi gén globális miRNS-szabályzása fontos lehet az átrendeződésben, a szinapszisokban vagy a hosszútávú idegi adaptációkban, ahol az alkoholfogyasztásról az elvonási tünetek vagy a függőségre való viselkedésváltozás jelenik meg.[212] Eddig mintegy 35 különböző miRNS szintjéről bizonyosodott be, hogy az alkoholhoz szokott agyban megváltozik, melyek közt vannak a sejtciklust, az apoptózist, a sejtadhéziót, az idegi fejlődést és a sejtkommunikációt befolyásoló géneket célzók is.[211] Megváltozott miRNS-szinteket találtak alkoholfüggő egerek mediális prefrontális kérgében, így a miRNS okozhatja feltehetően a transzlációs eltéréseket és a komplex kognitív viselkedés és a döntéshozatal agyi részeiben.[213]
A miRNS-ek erősödhetnek vagy gyengülhetnek krónikus alkoholhasználat esetén. A miR-206-expresszió alkoholfüggő patkányok prefrontális kérgében nő, az agyi eredetű neurotróf faktort (BDNF) célozva és annak expresszióját csökkentve. A BDNF fontos az új neuronok és szinapszisok kialakulásában és érésében, lehetséges negatív következményeket okozva a szinapszisnövekedésben és -plaszticitásban az alkoholfüggőknél.[214] Az alkoholindukált neuroinflammatiós válaszok szabályzásában fontos miR-155 erősödik, ami a mikroglia és az inflammatiós citokinek alkohol-patofiziológiában való fontos szerepét alátámasztja.[215] A miR-382 kisebb expressziója van jelen a nucleus accumbensben, a jutalomérzetben fontos bazális előagyi térrészben. A miR-382 a D1 dopaminreceptor célja, túlexpressziója növeli a DRD1 és a δ-fosB expresszióját, mely a nucleus accumbensben lévő függőségokozó transzkripciós eseményeket okoz.[216] A miR-382-túlexpresszió csökkenti az ivást és a DRD1-inhibíciót és a δ-fosB-erősödést patkány-alkoholizmusmodellekben, a kezelésben használható miRNS-célú gyógyszereket lehetővé téve.[216]
Obesitas[szerkesztés]
A miRNS-ek fontosak az őssejtprogenitorok zsírsejtté való differenciálódásának szabályzásában.[217] A pluripotens őssejtek adipogenezisben játszott szerepét a hMSC-Tert20 halhatatlanná tett csontvelő-eredetű stromalis sejtvonalban vizsgálták.[218] Decreased expression of miR-155, miR-221, and miR-222, have been found during the adipogenic programming of both immortalized and primary hMSCs, suggesting that they act as negative regulators of differentiation. Conversely, ectopic expression of the miRNAs 155, 221, and 222 significantly inhibited adipogenesis and repressed induction of the master regulators PPARγ and CCAAT/enhancer-binding protein alpha (CEBPA).[219] This paves the way for possible genetic obesity treatments.
További inzulinrezisztenciát, obesitast és diabéteszt csoportosító miRNS-osztály a let-7 család. Mennyisége az öregedés során nő.[220] A let-7 gyorsult öregedés mintájára történt ektópiás túlexpressziója egerekben inzulinrezisztenciát, így az obesitasnak és a diabétesznek való nagyobb kitettséget okozott.[221] Ezzel szemben a let-7 let-7-specifikus antagomirek injekciójával való inhibeálása nagyobb inzulinszenzitivitást és ellenállást okozott a nagyobb zsírtartalom miatti obesitasra és diabéteszre. Nemcsak gátolta az obesitas és a diabétesz kialakulását, hanem a meglévőt gyógyította.[222] Ezek alapján a let-7-gátlás új terápia lehet az obesitas és a 2-es típusú cukorbetegség ellen.
Preeklampszia[szerkesztés]
A méhlepényben expresszált legtöbb miRNS a C19MC (19. kromoszómabeli miRNS-klaszter, angolul: Chromosome 19 miRNA cluster) tagja. Ezek fontosak a terhesség alatt, kóros expressziójuk több betegségben patogenetikai tényező lehet.[112]:16 A preeklampszia kialakulásával több miRNS expresszióváltozása is összefügghet, ezekre Biró 2018-as értekezésében példákat is említett.[112]:17
Több miRNS expressziója is több helyen befolyásolja a kialakulást: a hsa-miR-210 emelkedése például az angiogenezist többek közt az EFNA3, a trofoblasztinváziót a KCMF-1, míg a gyulladást a STAT6/interleukin 4-út befolyásolásával módosítja.[112]:17
A hsa-miR-517 család tagjainak túlexpressziója az antiangiogén sFlt-1 szintjének növekedését okozza, ami a preeklampsziában fontos szerepet játszik, ezenkívül gátolja a trofoblasztinváziót.[112]:18
A sejten kívüli miRNS-ek 90–99%-a Ago-kötött, de többféleképp felszabadulhatnak – egy részük vezikulumokkal, más részük fehérjékhez kötve válik ki a sejtekből. Felszabadulhatnak sejthalálkor, ekkor a sejten kívüli DNS-ek és AGO-kötött miRNS-ek jelennek meg, aktív szekréció során miRNS-t, fehérjét és mRNS-t tartalmazó exoszómák vannak jelen.[112]:19
A méhlepényleválás után a hsa-miR-517c szintje jelentősen megnő feltehetően a fokozott trofoblaszttöredék-felszabadulás miatt.[112]:21
Homeosztázis[szerkesztés]
A miRNS-ek fontosak lehetnek a komplex enzimkaszkádok, például a hemosztatikus vérkoagulációs rendszer szabályzásában.[223] Funkcionális miRNS-eken végzett kísérletek 2018-ban a hemosztatikus rendszer terápiás céljait mutatták ki.[224][225] Ezek összefüggnek az endoplazmatikus retikulum kalciumhomeosztázisával, mely fontos a sejtdifferenciációhoz.[226]
Növényekben[szerkesztés]
A miRNS-ek számos fejlődési, homeosztatikus és immunfolyamat fontos szabályzói növényekben.[227] Céljuk a növényfejlődésben például a hajtások merisztémafejlődése, a levélnövekedés, a virág-, a magképzés vagy a gyökérnövekedés.[228][229][230][231] Továbbá összetett a szerepük számos hő-, alacsony hőmérsékleti, szárazság-, fénystresszel vagy γ-sugárzásnak való kitettséggel kapcsolatos abiotikus folyamatban.[227]
Vírusokban[szerkesztés]
A vírus-miRNS-ek fontosak a virális vagy gazdagének expressziójának a vírus javára való szabályzásában. Így a miRNS-ek fontosak a gazda-vírus kölcsönhatásokban és a vírusbetegségek patogenezisében.[232][233] A humán herpeszvírus-6 DNS-ének transzkripcióaktivátorainak expresszióját feltehetően vírus-miRNS-ek szabályozzák.[234]
Horizontális transzfer[szerkesztés]
Lukasik et al. 2014-ben növényi miRNS-ekről számoltak be humán és disznótej-exoszómákban.[235]
Célelőrejelzés[szerkesztés]
A miRNS-ek fehérjekódoló gének cél-mRNS-eihez köthetnek, csökkentve transzlációjukat vagy lebontva azokat. Fontos pontos azonosításuk.[236] 18 in silico algoritmus összehasonlítása elérhető.[237] Funkcionális miRNS-célzó algoritmusok vizsgálata szerint sok funkcionális miRNS-t a célelőrejelző algoritmusok eltéveszthetnek.[224]
Adatbázisok[szerkesztés]
A 2010-ben megjelent miRTarBase kísérletileg igazolt miRNS–cél kölcsönhatásokat,[238] a miRBase a mikro-RNS-ek jelzését és szekvenálási adatait tartalmazza.[239]
Jegyzetek[szerkesztés]
- ↑ a b c d Bartel DP (2018. március 1.). „Metazoan MicroRNAs”. Cell 173 (1), 20–51. o. DOI:10.1016/j.cell.2018.03.006. PMID 29570994.
- ↑ a b c Bartel DP (2004. január 1.). „MicroRNAs: genomics, biogenesis, mechanism, and function”. Cell 116 (2), 281–297. o. DOI:10.1016/S0092-8674(04)00045-5. PMID 14744438.
- ↑ Qureshi A, Thakur N, Monga I, Thakur A, Kumar M (2014. január 1.). „VIRmiRNA: a comprehensive resource for experimentally validated viral miRNAs and their targets”. Database 2014, bau103. o. DOI:10.1093/database/bau103. PMID 25380780.
- ↑ a b c Bartel DP (2009. január 1.). „MicroRNAs: target recognition and regulatory functions”. Cell 136 (2), 215–33. o. DOI:10.1016/j.cell.2009.01.002. PMID 19167326.
- ↑ a b Jonas, S (2015. július 1.). „Towards a molecular understanding of microRNA-mediated gene silencing”. Nature Reviews. Genetics 16 (7), 421–33. o. DOI:10.1038/nrg3965. PMID 26077373.
- ↑ (2010. augusztus 12.) „Mammalian microRNAs predominantly act to decrease target mRNA levels”. Nature 466 (7308), 835–40. o. DOI:10.1038/nature09267. PMID 20703300.
- ↑ Homo sapiens miRNAs in the miRBase. Manchesteri Egyetem. [2014. március 27-i dátummal az eredetiből archiválva]. (Hozzáférés: 2024. február 22.)
- ↑ Alles J, Fehlmann T, Fischer U, Backes C, Galata V, Minet M, Hart M, Abu-Halima M, Grässer FA, Lenhof HP, Keller A, Meese E (2019. április 1.). „An estimate of the total number of true human miRNAs”. Nucleic Acids Research 47 (7), 3353–3364. o. DOI:10.1093/nar/gkz097. PMID 30820533.
- ↑ Fromm B, Domanska D, Høye E, Ovchinnikov V, Kang W, Aparicio-Puerta E, Johansen M, Flatmark K, Mathelier A, Hovig E, Hackenberg M, Friedländer MR, Peterson KJ (2020. január 1.). „MirGeneDB 2.0: the metazoan microRNA complement”. Nucleic Acids Research 48 (D1), D132–D141. o. DOI:10.1093/nar/gkz885. PMID 31598695.
- ↑ Lim LP, Lau NC, Weinstein EG, Abdelhakim A, Yekta S, Rhoades MW, Burge CB, Bartel DP (2003. április 1.). „The microRNAs of Caenorhabditis elegans”. Genes & Development 17 (8), 991–1008. o. DOI:10.1101/gad.1074403. PMID 12672692.
- ↑ a b Lagos-Quintana M, Rauhut R, Yalcin A, Meyer J, Lendeckel W, Tuschl T (2002. április 1.). „Identification of tissue-specific microRNAs from mouse”. Current Biology 12 (9), 735–9. o. DOI:10.1016/S0960-9822(02)00809-6. PMID 12007417.
- ↑ a b c Lewis BP, Burge CB, Bartel DP (2005. január 1.). „Conserved seed pairing, often flanked by adenosines, indicates that thousands of human genes are microRNA targets”. Cell 120 (1), 15–20. o. DOI:10.1016/j.cell.2004.12.035. PMID 15652477.
- ↑ a b c Friedman RC, Farh KK, Burge CB, Bartel DP (2009. január 1.). „Most mammalian mRNAs are conserved targets of microRNAs”. Genome Research 19 (1), 92–105. o. DOI:10.1101/gr.082701.108. PMID 18955434.
- ↑ Fromm B, Billipp T, Peck LE, Johansen M, Tarver JE, King BL, Newcomb JM, Sempere LF, Flatmark K, Hovig E, Peterson KJ (2015). „A Uniform System for the Annotation of Vertebrate microRNA Genes and the Evolution of the Human microRNAome”. Annual Review of Genetics 49, 213–42. o. DOI:10.1146/annurev-genet-120213-092023. PMID 26473382.
- ↑ a b c Lee RC, Feinbaum RL, Ambros V (1993. december 1.). „The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14”. Cell 75 (5), 843–54. o. DOI:10.1016/0092-8674(93)90529-Y. PMID 8252621.
- ↑ a b Reinhart BJ, Slack FJ, Basson M, Pasquinelli AE, Bettinger JC, Rougvie AE, Horvitz HR, Ruvkun G (2000. február 1.). „The 21-nucleotide let-7 RNA regulates developmental timing in Caenorhabditis elegans”. Nature 403 (6772), 901–6. o. DOI:10.1038/35002607. PMID 10706289.
- ↑ a b Pasquinelli AE, Reinhart BJ, Slack F, Martindale MQ, Kuroda MI, Maller B, Hayward DC, Ball EE, Degnan B, Müller P, Spring J, Srinivasan A, Fishman M, Finnerty J, Corbo J, Levine M, Leahy P, Davidson E, Ruvkun G (2000. november 1.). „Conservation of the sequence and temporal expression of let-7 heterochronic regulatory RNA”. Nature 408 (6808), 86–9. o. DOI:10.1038/35040556. PMID 11081512.
- ↑ a b c Lagos-Quintana M, Rauhut R, Lendeckel W, Tuschl T (2001. október 1.). „Identification of novel genes coding for small expressed RNAs”. Science 294 (5543), 853–8. o. DOI:10.1126/science.1064921. PMID 11679670.
- ↑ a b c Lau NC, Lim LP, Weinstein EG, Bartel DP (2001. október 1.). „An abundant class of tiny RNAs with probable regulatory roles in Caenorhabditis elegans”. Science 294 (5543), 858–62. o. DOI:10.1126/science.1065062. PMID 11679671.
- ↑ a b c Lee RC, Ambros V (2001. október 1.). „An extensive class of small RNAs in Caenorhabditis elegans”. Science 294 (5543), 862–4. o. DOI:10.1126/science.1065329. PMID 11679672.
- ↑ Wienholds E, Kloosterman WP, Miska E, Alvarez-Saavedra E, Berezikov E, de Bruijn E, Horvitz HR, Kauppinen S, Plasterk RH (2005. július 1.). „MicroRNA expression in zebrafish embryonic development”. Science 309 (5732), 310–1. o. DOI:10.1126/science.1114519. PMID 15919954.
- ↑ a b Jones-Rhoades MW, Bartel DP, Bartel B (2006. április 25.). „MicroRNAS and their regulatory roles in plants”. Annual Review of Plant Biology 57, 19–53. o. DOI:10.1146/annurev.arplant.57.032905.105218. PMID 16669754.
- ↑ Brennecke J, Hipfner DR, Stark A, Russell RB, Cohen SM (2003. április 1.). „bantam encodes a developmentally regulated microRNA that controls cell proliferation and regulates the proapoptotic gene hid in Drosophila”. Cell 113 (1), 25–36. o. DOI:10.1016/S0092-8674(03)00231-9. PMID 12679032.
- ↑ Cuellar TL, McManus MT (2005. december 1.). „MicroRNAs and endocrine biology”. The Journal of Endocrinology 187 (3), 327–32. o. DOI:10.1677/joe.1.06426. PMID 16423811.
- ↑ Poy MN, Eliasson L, Krutzfeldt J, Kuwajima S, Ma X, Macdonald PE, Pfeffer S, Tuschl T, Rajewsky N, Rorsman P, Stoffel M (2004. november 1.). „A pancreatic islet-specific microRNA regulates insulin secretion”. Nature 432 (7014), 226–30. o. DOI:10.1038/nature03076. PMID 15538371.
- ↑ Chen CZ, Li L, Lodish HF, Bartel DP (2004. január 1.). „MicroRNAs modulate hematopoietic lineage differentiation”. Science 303 (5654), 83–6. o. DOI:10.1126/science.1091903. PMID 14657504.
- ↑ Wilfred BR, Wang WX, Nelson PT (2007. július 1.). „Energizing miRNA research: a review of the role of miRNAs in lipid metabolism, with a prediction that miR-103/107 regulates human metabolic pathways”. Molecular Genetics and Metabolism 91 (3), 209–17. o. DOI:10.1016/j.ymgme.2007.03.011. PMID 17521938.
- ↑ Harfe BD, McManus MT, Mansfield JH, Hornstein E, Tabin CJ (2005. augusztus 1.). „The RNaseIII enzyme Dicer is required for morphogenesis but not patterning of the vertebrate limb”. Proceedings of the National Academy of Sciences of the United States of America 102 (31), 10898–903. o. DOI:10.1073/pnas.0504834102. PMID 16040801.
- ↑ Trang P, Weidhaas JB, Slack FJ (2008. december 1.). „MicroRNAs as potential cancer therapeutics”. Oncogene 27 (Suppl 2), S52–7. o. DOI:10.1038/onc.2009.353. PMID 19956180.
- ↑ Li C, Feng Y, Coukos G, Zhang L (2009. december 1.). „Therapeutic microRNA strategies in human cancer”. The AAPS Journal 11 (4), 747–57. o. DOI:10.1208/s12248-009-9145-9. PMID 19876744.
- ↑ Fasanaro P, Greco S, Ivan M, Capogrossi MC, Martelli F (2010. január 1.). „microRNA: emerging therapeutic targets in acute ischemic diseases”. Pharmacology & Therapeutics 125 (1), 92–104. o. DOI:10.1016/j.pharmthera.2009.10.003. PMID 19896977.
- ↑ Hydbring P, Badalian-Very G (2013. augusztus 1.). „Clinical applications of microRNAs”. F1000Research 2, 136. o. DOI:10.12688/f1000research.2-136.v2. PMID 24627783.
- ↑ Wightman B, Ha I, Ruvkun G (1993. december 1.). „Posttranscriptional regulation of the heterochronic gene lin-14 by lin-4 mediates temporal pattern formation in C. elegans”. Cell 75 (5), 855–62. o. DOI:10.1016/0092-8674(93)90530-4. PMID 8252622.
- ↑ Giza DE, Calin GA. MicroRNA and Chronic Lymphocytic Leukemia, MicroRNA: Cancer, Advances in Experimental Medicine and Biology, 23–40. o.. DOI: 10.1007/978-3-319-23730-5_2 (2015). ISBN 978-3-319-23729-9
- ↑ Ambros V, Bartel B, Bartel DP, Burge CB, Carrington JC, Chen X, Dreyfuss G, Eddy SR, Griffiths-Jones S, Marshall M, Matzke M, Ruvkun G, Tuschl T (2003. március 1.). „A uniform system for microRNA annotation”. RNA 9 (3), 277–9. o. DOI:10.1261/rna.2183803. PMID 12592000.
- ↑ Griffiths-Jones S, Grocock RJ, van Dongen S, Bateman A, Enright AJ (2006. január 1.). „miRBase: microRNA sequences, targets and gene nomenclature”. Nucleic Acids Research 34 (Database issue), D140–4. o. DOI:10.1093/nar/gkj112. PMID 16381832.
- ↑ Wright MW, Bruford EA (2011. január 1.). „Naming 'junk': human non-protein coding RNA (ncRNA) gene nomenclature”. Human Genomics 5 (2), 90–8. o. DOI:10.1186/1479-7364-5-2-90. PMID 21296742.
- ↑ Hunt M, Banerjee S, Surana P, Liu M, Fuerst G, Mathioni S, Meyers BC, Nettleton D, Wise RP (2019. április 25.). „Small RNA discovery in the interaction between barley and the powdery mildew pathogen”. BMC Genomics 20 (1), 19–53. o. DOI:10.1186/s12864-019-5947-z. PMID 31345162.
- ↑ a b c Lewis BP, Shih IH, Jones-Rhoades MW, Bartel DP, Burge CB (2003. december 1.). „Prediction of mammalian microRNA targets”. Cell 115 (7), 787–98. o. DOI:10.1016/S0092-8674(03)01018-3. PMID 14697198.
- ↑ Ellwanger DC, Büttner FA, Mewes HW, Stümpflen V (2011. május 1.). „The sufficient minimal set of miRNA seed types”. Bioinformatics 27 (10), 1346–50. o. DOI:10.1093/bioinformatics/btr149. PMID 21441577.
- ↑ Rajewsky N (2006. június 1.). „microRNA target predictions in animals”. Nature Genetics 38 (6s), S8–13. o. DOI:10.1038/ng1798. PMID 16736023.
- ↑ Krek A, Grün D, Poy MN, Wolf R, Rosenberg L, Epstein EJ, MacMenamin P, da Piedade I, Gunsalus KC, Stoffel M, Rajewsky N (2005. május 1.). „Combinatorial microRNA target predictions”. Nature Genetics 37 (5), 495–500. o. DOI:10.1038/ng1536. PMID 15806104.
- ↑ Thomson DW, Bracken CP, Goodall GJ (2011. szeptember 1.). „Experimental strategies for microRNA target identification”. Nucleic Acids Research 39 (16), 6845–53. o. DOI:10.1093/nar/gkr330. PMID 21652644.
- ↑ a b Lim LP, Lau NC, Garrett-Engele P, Grimson A, Schelter JM, Castle J, Bartel DP, Linsley PS, Johnson JM (2005. február 1.). „Microarray analysis shows that some microRNAs downregulate large numbers of target mRNAs”. Nature 433 (7027), 769–73. o. DOI:10.1038/nature03315. PMID 15685193.
- ↑ Selbach M, Schwanhäusser B, Thierfelder N, Fang Z, Khanin R, Rajewsky N (2008. szeptember 1.). „Widespread changes in protein synthesis induced by microRNAs”. Nature 455 (7209), 58–63. o. DOI:10.1038/nature07228. PMID 18668040.
- ↑ Baek D, Villén J, Shin C, Camargo FD, Gygi SP, Bartel DP (2008. szeptember 1.). „The impact of microRNAs on protein output”. Nature 455 (7209), 64–71. o. DOI:10.1038/nature07242. PMID 18668037.
- ↑ a b Rodriguez A, Griffiths-Jones S, Ashurst JL, Bradley A (2004. október 1.). „Identification of mammalian microRNA host genes and transcription units”. Genome Research 14 (10A), 1902–10. o. DOI:10.1101/gr.2722704. PMID 15364901.
- ↑ a b c d Cai X, Hagedorn CH, Cullen BR (2004. december 1.). „Human microRNAs are processed from capped, polyadenylated transcripts that can also function as mRNAs”. RNA 10 (12), 1957–66. o. DOI:10.1261/rna.7135204. PMID 15525708.
- ↑ Weber MJ (2005. január 1.). „New human and mouse microRNA genes found by homology search”. The FEBS Journal 272 (1), 59–73. o. DOI:10.1111/j.1432-1033.2004.04389.x. PMID 15634332.
- ↑ Kim YK, Kim VN (2007. február 1.). „Processing of intronic microRNAs”. The EMBO Journal 26 (3), 775–83. o. DOI:10.1038/sj.emboj.7601512. PMID 17255951.
- ↑ Baskerville S, Bartel DP (2005. március 1.). „Microarray profiling of microRNAs reveals frequent coexpression with neighboring miRNAs and host genes”. RNA 11 (3), 241–7. o. DOI:10.1261/rna.7240905. PMID 15701730.
- ↑ a b c Lee Y, Kim M, Han J, Yeom KH, Lee S, Baek SH, Kim VN (2004. október 1.). „MicroRNA genes are transcribed by RNA polymerase II”. The EMBO Journal 23 (20), 4051–60. o. DOI:10.1038/sj.emboj.7600385. PMID 15372072.
- ↑ Zhou X, Ruan J, Wang G, Zhang W (2007. március 1.). „Characterization and identification of microRNA core promoters in four model species”. PLOS Computational Biology 3 (3), e37. o. DOI:10.1371/journal.pcbi.0030037. PMID 17352530.
- ↑ Faller M, Guo F (2008. november 1.). „MicroRNA biogenesis: there's more than one way to skin a cat”. Biochimica et Biophysica Acta (BBA) - Gene Regulatory Mechanisms 1779 (11), 663–7. o. DOI:10.1016/j.bbagrm.2008.08.005. PMID 18778799.
- ↑ Lee Y, Ahn C, Han J, Choi H, Kim J, Yim J, Lee J, Provost P, Rådmark O, Kim S, Kim VN (2003. szeptember 1.). „The nuclear RNase III Drosha initiates microRNA processing”. Nature 425 (6956), 415–9. o. DOI:10.1038/nature01957. PMID 14508493.
- ↑ Gregory RI, Chendrimada TP, Shiekhattar R. MicroRNA biogenesis: isolation and characterization of the microprocessor complex, MicroRNA Protocols, Methods in Molecular Biology, 33–47. o.. DOI: 10.1385/1-59745-123-1:33 (2006). ISBN 978-1-59745-123-9
- ↑ Han J, Lee Y, Yeom KH, Kim YK, Jin H, Kim VN (2004. december 1.). „The Drosha-DGCR8 complex in primary microRNA processing”. Genes & Development 18 (24), 3016–27. o. DOI:10.1101/gad.1262504. PMID 15574589.
- ↑ Han J, Lee Y, Yeom KH, Nam JW, Heo I, Rhee JK, Sohn SY, Cho Y, Zhang BT, Kim VN (2006. június 1.). „Molecular basis for the recognition of primary microRNAs by the Drosha-DGCR8 complex”. Cell 125 (5), 887–901. o. DOI:10.1016/j.cell.2006.03.043. PMID 16751099.
- ↑ Conrad T, Marsico A, Gehre M, Orom UA (2014. október 1.). „Microprocessor activity controls differential miRNA biogenesisin Vivo”. Cell Reports 9 (2), 542–54. o. DOI:10.1016/j.celrep.2014.09.007. PMID 25310978.
- ↑ Auyeung VC, Ulitsky I, McGeary SE, Bartel DP (2013. február 1.). „Beyond secondary structure: primary-sequence determinants license pri-miRNA hairpins for processing”. Cell 152 (4), 844–58. o. DOI:10.1016/j.cell.2013.01.031. PMID 23415231.
- ↑ Ali PS, Ghoshdastider U, Hoffmann J, Brutschy B, Filipek S (2012. november 1.). „Recognition of the let-7g miRNA precursor by human Lin28B”. FEBS Letters 586 (22), 3986–90. o. DOI:10.1016/j.febslet.2012.09.034. PMID 23063642.
- ↑ Ruby, JG (2007. július 5.). „Intronic microRNA precursors that bypass Drosha processing”. Nature 448 (7149), 83–6. o. DOI:10.1038/nature05983. PMID 17589500.
- ↑ (2007. július 5.) „Intronic microRNA precursors that bypass Drosha processing.”. Nature 448 (7149), 83–6. o. DOI:10.1038/nature05983. PMID 17589500.
- ↑ Berezikov E, Chung WJ, Willis J, Cuppen E, Lai EC (2007. október 1.). „Mammalian mirtron genes”. Molecular Cell 28 (2), 328–36. o. DOI:10.1016/j.molcel.2007.09.028. PMID 17964270.
- ↑ a b Kawahara Y, Megraw M, Kreider E, Iizasa H, Valente L, Hatzigeorgiou AG, Nishikura K (2008. szeptember 1.). „Frequency and fate of microRNA editing in human brain”. Nucleic Acids Research 36 (16), 5270–80. o. DOI:10.1093/nar/gkn479. PMID 18684997.
- ↑ Winter J, Jung S, Keller S, Gregory RI, Diederichs S (2009. március 1.). „Many roads to maturity: microRNA biogenesis pathways and their regulation”. Nature Cell Biology 11 (3), 228–34. o. DOI:10.1038/ncb0309-228. PMID 19255566.
- ↑ Ohman M (2007. október 1.). „A-to-I editing challenger or ally to the microRNA process”. Biochimie 89 (10), 1171–6. o. DOI:10.1016/j.biochi.2007.06.002. PMID 17628290.
- ↑ a b Murchison EP, Hannon GJ (2004. június 1.). „miRNAs on the move: miRNA biogenesis and the RNAi machinery”. Current Opinion in Cell Biology 16 (3), 223–9. o. DOI:10.1016/j.ceb.2004.04.003. PMID 15145345.

- ↑ a b c Lund E, Dahlberg JE (2006). „Substrate selectivity of exportin 5 and Dicer in the biogenesis of microRNAs”. Cold Spring Harbor Symposia on Quantitative Biology 71, 59–66. o. DOI:10.1101/sqb.2006.71.050. PMID 17381281.
- ↑ Park JE, Heo I, Tian Y, Simanshu DK, Chang H, Jee D, Patel DJ, Kim VN (2011. július 1.). „Dicer recognizes the 5' end of RNA for efficient and accurate processing”. Nature 475 (7355), 201–5. o. DOI:10.1038/nature10198. PMID 21753850.
- ↑ Ji X. The Mechanism of RNase III Action: How Dicer Dices, RNA Interference, Current Topics in Microbiology and Immunology, 99–116. o.. DOI: 10.1007/978-3-540-75157-1_5 (2008). ISBN 978-3-540-75156-4
- ↑ Mirihana Arachchilage G, Dassanayake AC, Basu S (2015. február 1.). „A potassium ion-dependent RNA structural switch regulates human pre-miRNA 92b maturation”. Chemistry & Biology 22 (2), 262–72. o. DOI:10.1016/j.chembiol.2014.12.013. PMID 25641166.
- ↑ Sohel MH (2016). „Extracellular/Circulating MicroRNAs: Release Mechanisms, Functions and Challenges”. Achievements in the Life Sciences 10 (2), 175–186. o. DOI:10.1016/j.als.2016.11.007.
- ↑ a b Boeckel JN, Reis SM, Leistner D, Thomé CE, Zeiher AM, Fichtlscherer S, Keller T (2014. április 1.). „From heart to toe: heart's contribution on peripheral microRNA levels”. International Journal of Cardiology 172 (3), 616–7. o. DOI:10.1016/j.ijcard.2014.01.082. PMID 24508494.
- ↑ Lelandais-Brière C, Sorin C, Declerck M, Benslimane A, Crespi M, Hartmann C (2010. március 1.). „Small RNA diversity in plants and its impact in development”. Current Genomics 11 (1), 14–23. o. DOI:10.2174/138920210790217918. PMID 20808519.
- ↑ Rana TM (2007. január 1.). „Illuminating the silence: understanding the structure and function of small RNAs”. Nature Reviews Molecular Cell Biology 8 (1), 23–36. o. DOI:10.1038/nrm2085. PMID 17183358.
- ↑ a b Schwarz DS, Zamore PD (2002. május 1.). „Why do miRNAs live in the miRNP?”. Genes & Development 16 (9), 1025–31. o. DOI:10.1101/gad.992502. PMID 12000786.
- ↑ Kundu P, Fabian MR, Sonenberg N, Bhattacharyya SN, Filipowicz W (2012. június 1.). „HuR protein attenuates miRNA-mediated repression by promoting miRISC dissociation from the target RNA”. Nucleic Acids Research 40 (11), 5088–5100. o. DOI:10.1093/nar/gks148. PMID 22362743.
- ↑ Krol J, Sobczak K, Wilczynska U, Drath M, Jasinska A, Kaczynska D, Krzyzosiak WJ (2004. október 1.). „Structural features of microRNA (miRNA) precursors and their relevance to miRNA biogenesis and small interfering RNA/short hairpin RNA design”. The Journal of Biological Chemistry 279 (40), 42230–9. o. DOI:10.1074/jbc.M404931200. PMID 15292246.
- ↑ Khvorova A, Reynolds A, Jayasena SD (2003. október 1.). „Functional siRNAs and miRNAs exhibit strand bias”. Cell 115 (2), 209–16. o. DOI:10.1016/S0092-8674(03)00801-8. PMID 14567918.
- ↑ Schwarz DS, Hutvágner G, Du T, Xu Z, Aronin N, Zamore PD (2003. október 1.). „Asymmetry in the assembly of the RNAi enzyme complex”. Cell 115 (2), 199–208. o. DOI:10.1016/S0092-8674(03)00759-1. PMID 14567917.
- ↑ Lin SL, Chang D, Ying SY (2005. augusztus 1.). „Asymmetry of intronic pre-miRNA structures in functional RISC assembly”. Gene 356, 32–8. o. DOI:10.1016/j.gene.2005.04.036. PMID 16005165.
- ↑ Okamura K, Chung WJ, Lai EC (2008. szeptember 1.). „The long and short of inverted repeat genes in animals: microRNAs, mirtrons and hairpin RNAs”. Cell Cycle 7 (18), 2840–5. o. DOI:10.4161/cc.7.18.6734. PMID 18769156.
- ↑ a b Pratt AJ, MacRae IJ (2009. július 1.). „The RNA-induced silencing complex: a versatile gene-silencing machine”. The Journal of Biological Chemistry 284 (27), 17897–901. o. DOI:10.1074/jbc.R900012200. PMID 19342379.
- ↑ MacRae IJ, Ma E, Zhou M, Robinson CV, Doudna JA (2008. január 1.). „In vitro reconstitution of the human RISC-loading complex”. Proceedings of the National Academy of Sciences of the United States of America 105 (2), 512–7. o. DOI:10.1073/pnas.0710869105. PMID 18178619.
- ↑ Mourelatos Z, Dostie J, Paushkin S, Sharma A, Charroux B, Abel L, Rappsilber J, Mann M, Dreyfuss G (2002. március 1.). „miRNPs: a novel class of ribonucleoproteins containing numerous microRNAs”. Genes & Development 16 (6), 720–8. o. DOI:10.1101/gad.974702. PMID 11914277.
- ↑ Meister G, Landthaler M, Peters L, Chen PY, Urlaub H, Lührmann R, Tuschl T (2005. december 1.). „Identification of novel argonaute-associated proteins”. Current Biology 15 (23), 2149–55. o. DOI:10.1016/j.cub.2005.10.048. PMID 16289642.
- ↑ Jing Q, Huang S, Guth S, Zarubin T, Motoyama A, Chen J, Di Padova F, Lin SC, Gram H, Han J (2005. március 1.). „Involvement of microRNA in AU-rich element-mediated mRNA instability”. Cell 120 (5), 623–34. o. DOI:10.1016/j.cell.2004.12.038. PMID 15766526.
- ↑ a b c Kai ZS, Pasquinelli AE (2010. január 1.). „MicroRNA assassins: factors that regulate the disappearance of miRNAs”. Nature Structural & Molecular Biology 17 (1), 5–10. o. DOI:10.1038/nsmb.1762. PMID 20051982.
- ↑ Chatterjee S, Grosshans H (2009. szeptember 1.). „Active turnover modulates mature microRNA activity in Caenorhabditis elegans”. Nature 461 (7263), 546–9. o. DOI:10.1038/nature08349. PMID 19734881.
- ↑ a b c Morozova N, Zinovyev A, Nonne N, Pritchard LL, Gorban AN, Harel-Bellan A (2012. szeptember 1.). „Kinetic signatures of microRNA modes of action”. RNA 18 (9), 1635–55. o. DOI:10.1261/rna.032284.112. PMID 22850425.
- ↑ Wang XJ, Reyes JL, Chua NH, Gaasterland T (2004). „Prediction and identification of Arabidopsis thaliana microRNAs and their mRNA targets”. Genome Biology 5 (9), R65. o. DOI:10.1186/gb-2004-5-9-r65. PMID 15345049.
- ↑ Kawasaki H, Taira K (2004). „MicroRNA-196 inhibits HOXB8 expression in myeloid differentiation of HL60 cells”. Nucleic Acids Symposium Series 48 (1), 211–2. o. DOI:10.1093/nass/48.1.211. PMID 17150553.
- ↑ a b Moxon S, Jing R, Szittya G, Schwach F, Rusholme Pilcher RL, Moulton V, Dalmay T (2008. október 1.). „Deep sequencing of tomato short RNAs identifies microRNAs targeting genes involved in fruit ripening”. Genome Research 18 (10), 1602–9. o. DOI:10.1101/gr.080127.108. PMID 18653800.
- ↑ Mazière P, Enright AJ (2007. június 1.). „Prediction of microRNA targets”. Drug Discovery Today 12 (11–12), 452–8. o. DOI:10.1016/j.drudis.2007.04.002. PMID 17532529.
- ↑ Williams AE (2008. február 1.). „Functional aspects of animal microRNAs”. Cellular and Molecular Life Sciences 65 (4), 545–62. o. DOI:10.1007/s00018-007-7355-9. PMID 17965831.
- ↑ Eulalio A, Huntzinger E, Nishihara T, Rehwinkel J, Fauser M, Izaurralde E (2009. január 1.). „Deadenylation is a widespread effect of miRNA regulation”. RNA 15 (1), 21–32. o. DOI:10.1261/rna.1399509. PMID 19029310.
- ↑ Bazzini AA, Lee MT, Giraldez AJ (2012. április 1.). „Ribosome profiling shows that miR-430 reduces translation before causing mRNA decay in zebrafish”. Science 336 (6078), 233–7. o. DOI:10.1126/science.1215704. PMID 22422859.
- ↑ Djuranovic S, Nahvi A, Green R (2012. április 1.). „miRNA-mediated gene silencing by translational repression followed by mRNA deadenylation and decay”. Science 336 (6078), 237–40. o. DOI:10.1126/science.1215691. PMID 22499947.
- ↑ Tan Y, Zhang B, Wu T, Skogerbø G, Zhu X, Guo X, He S, Chen R (2009. február 1.). „Transcriptional inhibiton of Hoxd4 expression by miRNA-10a in human breast cancer cells”. BMC Molecular Biology 10 (1), 12. o. DOI:10.1186/1471-2199-10-12. PMID 19232136.
- ↑ Hawkins PG, Morris KV (2008. március 1.). „RNA and transcriptional modulation of gene expression”. Cell Cycle 7 (5), 602–7. o. DOI:10.4161/cc.7.5.5522. PMID 18256543.
- ↑ Stark A, Brennecke J, Bushati N, Russell RB, Cohen SM (2005. december 1.). „Animal MicroRNAs confer robustness to gene expression and have a significant impact on 3'UTR evolution”. Cell 123 (6), 1133–46. o. DOI:10.1016/j.cell.2005.11.023. PMID 16337999.
- ↑ He L, Hannon GJ (2004. július 1.). „MicroRNAs: small RNAs with a big role in gene regulation”. Nature Reviews. Genetics 5 (7), 522–531. o. DOI:10.1038/nrg1379. PMID 15211354.
- ↑ Li LC.szerk.: Morris KV: Small RNA-Mediated Gene Activation, RNA and the Regulation of Gene Expression: A Hidden Layer of Complexity. Horizon Scientific Press (2008). ISBN 978-1-904455-25-7
- ↑ Place RF, Li LC, Pookot D, Noonan EJ, Dahiya R (2008. február 1.). „MicroRNA-373 induces expression of genes with complementary promoter sequences”. Proceedings of the National Academy of Sciences of the United States of America 105 (5), 1608–13. o. DOI:10.1073/pnas.0707594105. PMID 18227514.
- ↑ Salmena L, Poliseno L, Tay Y, Kats L, Pandolfi PP (2011. augusztus 1.). „A ceRNA hypothesis: the Rosetta Stone of a hidden RNA language?”. Cell 146 (3), 353–8. o. DOI:10.1016/j.cell.2011.07.014. PMID 21802130.
- ↑ a b Kumar S, Reddy PH (2016. szeptember 1.). „Are circulating microRNAs peripheral biomarkers for Alzheimer's disease?”. Biochim Biophys Acta 1862 (9), 1617–27. o. DOI:10.1016/j.bbadis.2016.06.001. PMID 27264337.
- ↑ Duttagupta R, Jiang R, Gollub J, Getts RC, Jones KW (2011. június 17.). „Impact of Cellular miRNAs on Circulating miRNA Biomarker Signatures”. PLoS ONE 6 (6), e20769. o. DOI:10.1371/journal.pone.0020769. (Hozzáférés: 2024. február 23.)
- ↑ van den Berg MM, Krauskopf J, Ramaekers JG et al. (2020. február 1.). „Circulating microRNAs as potential biomarkers for psychiatric and neurodegenerative disorders”. Prog Neurobiol 185, 101732. o. DOI:10.1016/j.pneurobio.2019.101732. PMID 31816349.
- ↑ Cuman C, Van Sinderen M, Gantier MP, Rainczuk K, Sorby K, Rombauts L, Osianlis T, Dimitriadis E (2015. október 1.). „Human Blastocyst Secreted microRNA Regulate Endometrial Epithelial Cell Adhesion”. eBioMedicine 2 (10), 1528–1535. o. DOI:10.1016/j.ebiom.2015.09.003. PMID 26629549.
- ↑ Zhou L, Miller C, Miraglia LJ, Romero A, Mure LS, Panda S, Kay SA (2021. január 1.). „A genome-wide microRNA screen identifies the microRNA-183/96/182 cluster as a modulator of circadian rhythms”. Proceedings of the National Academy of Sciences of the United States of America 118 (1), e2020454118. o. DOI:10.1073/pnas.2020454118. PMID 33443164.
- ↑ a b c d e f g Biró OB (2018), A mikroRNS-ek patogenetikai szerepe és expressziós mintázata praeeclampsiában, Budapest: Semmelweis Egyetem Klinikai orvostudományok Doktori Iskola, <http://old.semmelweis.hu/wp-content/phd/phd_live/vedes/export/biroorsolya.d.pdf>. Hozzáférés ideje: 2024-04-16
- ↑ Axtell MJ, Bartel DP (2005. június 1.). „Antiquity of microRNAs and their targets in land plants”. The Plant Cell 17 (6), 1658–73. o. DOI:10.1105/tpc.105.032185. PMID 15849273.
- ↑ Tanzer A, Stadler PF (2004. május 1.). „Molecular evolution of a microRNA cluster”. Journal of Molecular Biology 339 (2), 327–35. o. DOI:10.1016/j.jmb.2004.03.065. PMID 15136036.
- ↑ Chen K, Rajewsky N (2007. február 1.). „The evolution of gene regulation by transcription factors and microRNAs”. Nature Reviews Genetics 8 (2), 93–103. o. DOI:10.1038/nrg1990. PMID 17230196.
- ↑ Lee CT, Risom T, Strauss WM (2007. április 1.). „Evolutionary conservation of microRNA regulatory circuits: an examination of microRNA gene complexity and conserved microRNA-target interactions through metazoan phylogeny”. DNA and Cell Biology 26 (4), 209–18. o. DOI:10.1089/dna.2006.0545. PMID 17465887.
- ↑ a b c d Peterson KJ, Dietrich MR, McPeek MA (2009. július 1.). „MicroRNAs and metazoan macroevolution: insights into canalization, complexity, and the Cambrian explosion”. BioEssays 31 (7), 736–47. o. DOI:10.1002/bies.200900033. PMID 19472371.
- ↑ Shabalina SA, Koonin EV (2008. október 1.). „Origins and evolution of eukaryotic RNA interference”. Trends in Ecology & Evolution 23 (10), 578–87. o. DOI:10.1016/j.tree.2008.06.005. PMID 18715673.
- ↑ Axtell MJ, Westholm JO, Lai EC (2011). „Vive la différence: biogenesis and evolution of microRNAs in plants and animals”. Genome Biology 12 (4), 221. o. DOI:10.1186/gb-2011-12-4-221. PMID 21554756.
- ↑ a b Wheeler BM, Heimberg AM, Moy VN, Sperling EA, Holstein TW, Heber S, Peterson KJ (2009). „The deep evolution of metazoan microRNAs”. Evolution & Development 11 (1), 50–68. o. DOI:10.1111/j.1525-142X.2008.00302.x. PMID 19196333.
- ↑ Pashkovskiy PP, Ryazansky SS (2013. június 1.). „Biogenesis, evolution, and functions of plant microRNAs”. Biochemistry. Biokhimiia 78 (6), 627–37. o. DOI:10.1134/S0006297913060084. PMID 23980889.
- ↑ a b Heimberg AM, Sempere LF, Moy VN, Donoghue PC, Peterson KJ (2008. február 1.). „MicroRNAs and the advent of vertebrate morphological complexity”. Proceedings of the National Academy of Sciences of the United States of America 105 (8), 2946–50. o. DOI:10.1073/pnas.0712259105. PMID 18287013.
- ↑ a b c Nozawa M, Miura S, Nei M (2010. július 1.). „Origins and evolution of microRNA genes in Drosophila species”. Genome Biology and Evolution 2, 180–9. o. DOI:10.1093/gbe/evq009. PMID 20624724.
- ↑ Allen E, Xie Z, Gustafson AM, Sung GH, Spatafora JW, Carrington JC (2004. december 1.). „Evolution of microRNA genes by inverted duplication of target gene sequences in Arabidopsis thaliana”. Nature Genetics 36 (12), 1282–90. o. DOI:10.1038/ng1478. PMID 15565108.
- ↑ Warthmann N, Das S, Lanz C, Weigel D (2008. május 1.). „Comparative analysis of the MIR319a microRNA locus in Arabidopsis and related Brassicaceae”. Molecular Biology and Evolution 25 (5), 892–902. o. DOI:10.1093/molbev/msn029. PMID 18296705.
- ↑ Fahlgren N, Jogdeo S, Kasschau KD, Sullivan CM, Chapman EJ, Laubinger S, Smith LM, Dasenko M, Givan SA, Weigel D, Carrington JC (2010. április 1.). „MicroRNA gene evolution in Arabidopsis lyrata and Arabidopsis thaliana”. The Plant Cell 22 (4), 1074–89. o. DOI:10.1105/tpc.110.073999. PMID 20407027.
- ↑ Caravas J, Friedrich M (2010. június 1.). „Of mites and millipedes: recent progress in resolving the base of the arthropod tree”. BioEssays 32 (6), 488–95. o. DOI:10.1002/bies.201000005. PMID 20486135.
- ↑ Kenny NJ, Namigai EK, Marlétaz F, Hui JH, Shimeld SM (2015. december 1.). „Draft genome assemblies and predicted microRNA complements of the intertidal lophotrochozoans Patella vulgata (Mollusca, Patellogastropoda) and Spirobranchus (Pomatoceros) lamarcki (Annelida, Serpulida)”. Marine Genomics 24, 139–46. o. DOI:10.1016/j.margen.2015.07.004. PMID 26319627.
- ↑ Cock JM, Sterck L, Rouzé P, Scornet D, Allen AE, Amoutzias G, Anthouard V, Artiguenave F, Aury JM, Badger JH, Beszteri B, Billiau K, Bonnet E, Bothwell JH, Bowler C, Boyen C, Brownlee C, Carrano CJ, Charrier B, Cho GY, Coelho SM, Collén J, Corre E, Da Silva C, Delage L, Delaroque N, Dittami SM, Doulbeau S, Elias M, Farnham G, Gachon CM, Gschloessl B, Heesch S, Jabbari K, Jubin C, Kawai H, Kimura K, Kloareg B, Küpper FC, Lang D, Le Bail A, Leblanc C, Lerouge P, Lohr M, Lopez PJ, Martens C, Maumus F, Michel G, Miranda-Saavedra D, Morales J, Moreau H, Motomura T, Nagasato C, Napoli CA, Nelson DR, Nyvall-Collén P, Peters AF, Pommier C, Potin P, Poulain J, Quesneville H, Read B, Rensing SA, Ritter A, Rousvoal S, Samanta M, Samson G, Schroeder DC, Ségurens B, Strittmatter M, Tonon T, Tregear JW, Valentin K, von Dassow P, Yamagishi T, Van de Peer Y, Wincker P (2010. június 1.). „The Ectocarpus genome and the independent evolution of multicellularity in brown algae”. Nature 465 (7298), 617–21. o. DOI:10.1038/nature09016. PMID 20520714.
- ↑ Cuperus JT, Fahlgren N, Carrington JC (2011. február 1.). „Evolution and functional diversification of MIRNA genes”. The Plant Cell 23 (2), 431–42. o. DOI:10.1105/tpc.110.082784. PMID 21317375.
- ↑ Ryan JF, Pang K, Schnitzler CE, Nguyen AD, Moreland RT, Simmons DK, Koch BJ, Francis WR, Havlak P, Smith SA, Putnam NH, Haddock SH, Dunn CW, Wolfsberg TG, Mullikin JC, Martindale MQ, Baxevanis AD (2013. december 1.). „The genome of the ctenophore Mnemiopsis leidyi and its implications for cell type evolution”. Science 342 (6164), 1242592. o. DOI:10.1126/science.1242592. PMID 24337300.
- ↑ Maxwell EK, Ryan JF, Schnitzler CE, Browne WE, Baxevanis AD (2012. december 1.). „MicroRNAs and essential components of the microRNA processing machinery are not encoded in the genome of the ctenophore Mnemiopsis leidyi”. BMC Genomics 13 (1), 714. o. DOI:10.1186/1471-2164-13-714. PMID 23256903.
- ↑ Dimond PF (2010. március 15.). „miRNAs' Therapeutic Potential”. Genetic Engineering & Biotechnology News 30 (6), 1. o. (Hozzáférés: 2010. július 10.)
- ↑ Tjaden B, Goodwin SS, Opdyke JA, Guillier M, Fu DX, Gottesman S, Storz G (2006). „Target prediction for small, noncoding RNAs in bacteria”. Nucleic Acids Research 34 (9), 2791–802. o. DOI:10.1093/nar/gkl356. PMID 16717284.
- ↑ Edelbroek B, Kjellin J, Biryukova I, Liao Z, Lundberg T, Noegel AA, Eichinger L, Friedländer MR, Söderbrom F (2024. február 20.). „Evolution of microRNAs in Amoebozoa and implications for the origin of multicellularity”. Nucleic Acids Research 52 (4), gkae109. o. DOI:10.1093/nar/gkae109. PMID 38375870. (Hozzáférés: 2024. február 22.)
- ↑ Liu CG, Calin GA, Volinia S, Croce CM (2008). „MicroRNA expression profiling using microarrays”. Nature Protocols 3 (4), 563–78. o. DOI:10.1038/nprot.2008.14. PMID 18388938.
- ↑ Chen C, Ridzon DA, Broomer AJ, Zhou Z, Lee DH, Nguyen JT, Barbisin M, Xu NL, Mahuvakar VR, Andersen MR, Lao KQ, Livak KJ, Guegler KJ (2005. november 1.). „Real-time quantification of microRNAs by stem-loop RT-PCR”. Nucleic Acids Research 33 (20), e179. o. DOI:10.1093/nar/gni178. PMID 16314309.
- ↑ Shingara J, Keiger K, Shelton J, Laosinchai-Wolf W, Powers P, Conrad R, Brown D, Labourier E (2005. szeptember 1.). „An optimized isolation and labeling platform for accurate microRNA expression profiling”. RNA 11 (9), 1461–70. o. DOI:10.1261/rna.2610405. PMID 16043497.
- ↑ Buermans HP, Ariyurek Y, van Ommen G, den Dunnen JT, 't Hoen PA (2010. december 1.). „New methods for next generation sequencing based microRNA expression profiling”. BMC Genomics 11, 716. o. DOI:10.1186/1471-2164-11-716. PMID 21171994.
- ↑ Kloosterman WP, Wienholds E, Ketting RF, Plasterk RH (2004). „Substrate requirements for let-7 function in the developing zebrafish embryo”. Nucleic Acids Research 32 (21), 6284–91. o. DOI:10.1093/nar/gkh968. PMID 15585662.
- ↑ Flynt AS, Li N, Thatcher EJ, Solnica-Krezel L, Patton JG (2007. február 1.). „Zebrafish miR-214 modulates Hedgehog signaling to specify muscle cell fate”. Nature Genetics 39 (2), 259–63. o. DOI:10.1038/ng1953. PMID 17220889.
- ↑ Meister G, Landthaler M, Dorsett Y, Tuschl T (2004. március 1.). „Sequence-specific inhibition of microRNA- and siRNA-induced RNA silencing”. RNA 10 (3), 544–50. o. DOI:10.1261/rna.5235104. PMID 14970398.
- ↑ Kloosterman WP, Lagendijk AK, Ketting RF, Moulton JD, Plasterk RH (2007. augusztus 1.). „Targeted inhibition of miRNA maturation with morpholinos reveals a role for miR-375 in pancreatic islet development”. PLOS Biology 5 (8), e203. o. DOI:10.1371/journal.pbio.0050203. PMID 17676975.
- ↑ Gebert LF, Rebhan MA, Crivelli SE, Denzler R, Stoffel M, Hall J (2014. január 1.). „Miravirsen (SPC3649) can inhibit the biogenesis of miR-122”. Nucleic Acids Research 42 (1), 609–21. o. DOI:10.1093/nar/gkt852. PMID 24068553.
- ↑ Choi WY, Giraldez AJ, Schier AF (2007. október 1.). „Target protectors reveal dampening and balancing of Nodal agonist and antagonist by miR-430”. Science 318 (5848), 271–4. o. DOI:10.1126/science.1147535. PMID 17761850.
- ↑ You Y, Moreira BG, Behlke MA, Owczarzy R (2006. május 1.). „Design of LNA probes that improve mismatch discrimination”. Nucleic Acids Research 34 (8), e60. o. DOI:10.1093/nar/gkl175. PMID 16670427.
- ↑ Lagendijk AK, Moulton JD, Bakkers J (2012. június 1.). „Revealing details: whole mount microRNA in situ hybridization protocol for zebrafish embryos and adult tissues”. Biology Open 1 (6), 566–9. o. DOI:10.1242/bio.2012810. PMID 23213449.
- ↑ Kaur H, Arora A, Wengel J, Maiti S (2006. június 1.). „Thermodynamic, counterion, and hydration effects for the incorporation of locked nucleic acid nucleotides into DNA duplexes”. Biochemistry 45 (23), 7347–55. o. DOI:10.1021/bi060307w. PMID 16752924.
- ↑ Nielsen JA, Lau P, Maric D, Barker JL, Hudson LD (2009. augusztus 1.). „Integrating microRNA and mRNA expression profiles of neuronal progenitors to identify regulatory networks underlying the onset of cortical neurogenesis”. BMC Neuroscience 10, 98. o. DOI:10.1186/1471-2202-10-98. PMID 19689821.
- ↑ Grimson A, Farh KK, Johnston WK, Garrett-Engele P, Lim LP, Bartel DP (2007. július 1.). „MicroRNA targeting specificity in mammals: determinants beyond seed pairing”. Molecular Cell 27 (1), 91–105. o. DOI:10.1016/j.molcel.2007.06.017. PMID 17612493.
- ↑ Griffiths-Jones S, Saini HK, van Dongen S, Enright AJ (2008. január 1.). „miRBase: tools for microRNA genomics”. Nucleic Acids Research 36 (Database issue), D154–8. o. DOI:10.1093/nar/gkm952. PMID 17991681.
- ↑ Nam S, Li M, Choi K, Balch C, Kim S, Nephew KP (2009. július 1.). „MicroRNA and mRNA integrated analysis (MMIA): a web tool for examining biological functions of microRNA expression”. Nucleic Acids Research 37 (Web Server issue), W356–62. o. DOI:10.1093/nar/gkp294. PMID 19420067.
- ↑ Artmann S, Jung K, Bleckmann A, Beissbarth T (2012). „Detection of simultaneous group effects in microRNA expression and related target gene sets”. PLOS ONE 7 (6), e38365. o. DOI:10.1371/journal.pone.0038365. PMID 22723856.
- ↑ Jiang Q, Wang Y, Hao Y, Juan L, Teng M, Zhang X, Li M, Wang G, Liu Y (2009. január 1.). „miR2Disease: a manually curated database for microRNA deregulation in human disease”. Nucleic Acids Research 37 (Database issue), D98–104. o. DOI:10.1093/nar/gkn714. PMID 18927107.
- ↑ Mencía A, Modamio-Høybjør S, Redshaw N, Morín M, Mayo-Merino F, Olavarrieta L, Aguirre LA, del Castillo I, Steel KP, Dalmay T, Moreno F, Moreno-Pelayo MA (2009. május 1.). „Mutations in the seed region of human miR-96 are responsible for nonsyndromic progressive hearing loss”. Nature Genetics 41 (5), 609–13. o. DOI:10.1038/ng.355. PMID 19363479.
- ↑ Hughes AE, Bradley DT, Campbell M, Lechner J, Dash DP, Simpson DA, Willoughby CE (2011. november 1.). „Mutation altering the miR-184 seed region causes familial keratoconus with cataract”. American Journal of Human Genetics 89 (5), 628–33. o. DOI:10.1016/j.ajhg.2011.09.014. PMID 21996275.
- ↑ de Pontual L, Yao E, Callier P, Faivre L, Drouin V, Cariou S, Van Haeringen A, Geneviève D, Goldenberg A, Oufadem M, Manouvrier S, Munnich A, Vidigal JA, Vekemans M, Lyonnet S, Henrion-Caude A, Ventura A, Amiel J (2011. szeptember 1.). „Germline deletion of the miR-17~92 cluster causes skeletal and growth defects in humans”. Nature Genetics 43 (10), 1026–30. o. DOI:10.1038/ng.915. PMID 21892160.
- ↑ Calin GA, Liu CG, Sevignani C, Ferracin M, Felli N, Dumitru CD, Shimizu M, Cimmino A, Zupo S, Dono M, Dell'Aquila ML, Alder H, Rassenti L, Kipps TJ, Bullrich F, Negrini M, Croce CM (2004. augusztus 1.). „MicroRNA profiling reveals distinct signatures in B cell chronic lymphocytic leukemias”. Proc Natl Acad Sci U S A 101 (32), 11755–60. o. DOI:10.1073/pnas.0404432101. PMID 15284443.
- ↑ Velle, Angelo (2023. január 17.). „A comprehensive investigation of histotype-specific microRNA and their variants in Stage I epithelial ovarian cancers” (angol nyelven). International Journal of Cancer 152 (9), 1989–2001. o. DOI:10.1002/ijc.34408. ISSN 0020-7136. PMID 36541726.
- ↑ Kyriakidis I, Kyriakidis K, Tsezou A (2022. augusztus 1.). „MicroRNAs and the Diagnosis of Childhood Acute Lymphoblastic Leukemia: Systematic Review, Meta-Analysis and Re-Analysis with Novel Small RNA-Seq Tools”. Cancers 14 (16), 3976. o. DOI:10.3390/cancers14163976. PMID 36010971.
- ↑ Võsa U, Vooder T, Kolde R, Fischer K, Välk K, Tõnisson N, Roosipuu R, Vilo J, Metspalu A, Annilo T (2011. október 1.). „Identification of miR-374a as a prognostic marker for survival in patients with early-stage nonsmall cell lung cancer”. Genes, Chromosomes & Cancer 50 (10), 812–22. o. DOI:10.1002/gcc.20902. PMID 21748820.
- ↑ Akçakaya P, Ekelund S, Kolosenko I, Caramuta S, Ozata DM, Xie H, Lindforss U, Olivecrona H, Lui WO (2011. augusztus 1.). „miR-185 and miR-133b deregulation is associated with overall survival and metastasis in colorectal cancer”. International Journal of Oncology 39 (2), 311–8. o. DOI:10.3892/ijo.2011.1043. PMID 21573504.
- ↑ a b Eyking A, Reis H, Frank M, Gerken G, Schmid KW, Cario E (2016). „MiR-205 and MiR-373 Are Associated with Aggressive Human Mucinous Colorectal Cancer”. PLOS ONE 11 (6), e0156871. o. DOI:10.1371/journal.pone.0156871. PMID 27271572.
- ↑ Xu G et al. (2013). „NA-21 promotes hepatocellular carcinoma HepG2 cell proliferation through repression of mitogen-activated protein kinase-kinase 3”. PMID 24112539.
- ↑ Jones K, Nourse JP, Keane C, Bhatnagar A, Gandhi MK (2014. január 1.). „Plasma microRNA are disease response biomarkers in classical Hodgkin lymphoma”. Clinical Cancer Research 20 (1), 253–64. o. DOI:10.1158/1078-0432.CCR-13-1024. PMID 24222179.
- ↑ Hosseinahli N, Aghapour M, Duijf PH, Baradaran B (2018. augusztus 1.). „Treating cancer with microRNA replacement therapy: A literature review”. Journal of Cellular Physiology 233 (8), 5574–5588. o. DOI:10.1002/jcp.26514. PMID 29521426.
- ↑ Liu G, Sun Y, Ji P, Li X, Cogdell D, Yang D, Parker Kerrigan BC, Shmulevich I, Chen K, Sood AK, Xue F, Zhang W (2014. július 1.). „MiR-506 suppresses proliferation and induces senescence by directly targeting the CDK4/6-FOXM1 axis in ovarian cancer”. The Journal of Pathology 233 (3), 308–18. o. DOI:10.1002/path.4348. PMID 24604117.
- ↑ Wen SY, Lin Y, Yu YQ, Cao SJ, Zhang R, Yang XM, Li J, Zhang YL, Wang YH, Ma MZ, Sun WW, Lou XL, Wang JH, Teng YC, Zhang ZG (2015. február 1.). „miR-506 acts as a tumor suppressor by directly targeting the hedgehog pathway transcription factor Gli3 in human cervical cancer”. Oncogene 34 (6), 717–25. o. DOI:10.1038/onc.2014.9. PMID 24608427.
- ↑ Pekingi Egyetem (2022. szeptember 13.). „Scientists Develop a New, Powerful Cancer-Fighting Weapon”. Cell 185 (11), 1888–1904.e24. o, Kiadó: SciTech Daily. DOI:10.1016/j.cell.2022.04.030. PMID 35623329. (Hozzáférés: 2022. szeptember 15.)
- ↑ Loeb KR, Loeb LA (2000. március 1.). „Significance of multiple mutations in cancer”. Carcinogenesis 21 (3), 379–385. o. DOI:10.1093/carcin/21.3.379. PMID 10688858.
- ↑ Lodish H, Berk A, Kaiser CA, Krieger M, Bretscher A, Ploegh H, Amon A, Martin KC. Molecular Cell Biology, 8th, New York: W. H. Freeman and Company, 203. o. (2016). ISBN 978-1-4641-8339-3
- ↑ Hu H, Gatti RA (2011. június 1.). „MicroRNAs: new players in the DNA damage response”. Journal of Molecular Cell Biology 3 (3), 151–158. o. DOI:10.1093/jmcb/mjq042. PMID 21183529.
- ↑ Jasperson KW, Tuohy TM, Neklason DW, Burt RW (2010. június 1.). „Hereditary and familial colon cancer”. Gastroenterology 138 (6), 2044–58. o. DOI:10.1053/j.gastro.2010.01.054. PMID 20420945.
- ↑ Truninger K, Menigatti M, Luz J, Russell A, Haider R, Gebbers JO, Bannwart F, Yurtsever H, Neuweiler J, Riehle HM, Cattaruzza MS, Heinimann K, Schär P, Jiricny J, Marra G (2005. május 1.). „Immunohistochemical analysis reveals high frequency of PMS2 defects in colorectal cancer”. Gastroenterology 128 (5), 1160–71. o. DOI:10.1053/j.gastro.2005.01.056. PMID 15887099.
- ↑ Valeri N, Gasparini P, Fabbri M, Braconi C, Veronese A, Lovat F, Adair B, Vannini I, Fanini F, Bottoni A, Costinean S, Sandhu SK, Nuovo GJ, Alder H, Gafa R, Calore F, Ferracin M, Lanza G, Volinia S, Negrini M, McIlhatton MA, Amadori D, Fishel R, Croce CM (2010. április 1.). „Modulation of mismatch repair and genomic stability by miR-155”. Proceedings of the National Academy of Sciences of the United States of America 107 (15), 6982–7. o. DOI:10.1073/pnas.1002472107. PMID 20351277.
- ↑ a b c Zhang W, Zhang J, Hoadley K, Kushwaha D, Ramakrishnan V, Li S, Kang C, You Y, Jiang C, Song SW, Jiang T, Chen CC (2012. június 1.). „miR-181d: a predictive glioblastoma biomarker that downregulates MGMT expression”. Neuro-Oncology 14 (6), 712–9. o. DOI:10.1093/neuonc/nos089. PMID 22570426.
- ↑ Spiegl-Kreinecker S, Pirker C, Filipits M, Lötsch D, Buchroithner J, Pichler J, Silye R, Weis S, Micksche M, Fischer J, Berger W (2010. január 1.). „O6-Methylguanine DNA methyltransferase protein expression in tumor cells predicts outcome of temozolomide therapy in glioblastoma patients”. Neuro-Oncology 12 (1), 28–36. o. DOI:10.1093/neuonc/nop003. PMID 20150365.
- ↑ Sgarra R, Rustighi A, Tessari MA, Di Bernardo J, Altamura S, Fusco A, Manfioletti G, Giancotti V (2004. szeptember 1.). „Nuclear phosphoproteins HMGA and their relationship with chromatin structure and cancer”. FEBS Letters 574 (1–3), 1–8. o. DOI:10.1016/j.febslet.2004.08.013. PMID 15358530.
- ↑ Xu Y, Sumter TF, Bhattacharya R, Tesfaye A, Fuchs EJ, Wood LJ, Huso DL, Resar LM (2004. május 1.). „The HMG-I oncogene causes highly penetrant, aggressive lymphoid malignancy in transgenic mice and is overexpressed in human leukemia”. Cancer Research 64 (10), 3371–5. o. DOI:10.1158/0008-5472.CAN-04-0044. PMID 15150086.
- ↑ Borrmann L, Schwanbeck R, Heyduk T, Seebeck B, Rogalla P, Bullerdiek J, Wisniewski JR (2003. december 1.). „High mobility group A2 protein and its derivatives bind a specific region of the promoter of DNA repair gene ERCC1 and modulate its activity”. Nucleic Acids Research 31 (23), 6841–51. o. DOI:10.1093/nar/gkg884. PMID 14627817.
- ↑ Facista A, Nguyen H, Lewis C, Prasad AR, Ramsey L, Zaitlin B, Nfonsam V, Krouse RS, Bernstein H, Payne CM, Stern S, Oatman N, Banerjee B, Bernstein C (2012. április 1.). „Deficient expression of DNA repair enzymes in early progression to sporadic colon cancer”. Genome Integrity 3 (1), 3. o. DOI:10.1186/2041-9414-3-3. PMID 22494821.
- ↑ Gibert B, Delloye-Bourgeois C, Gattolliat CH, Meurette O, Le Guernevel S, Fombonne J, Ducarouge B, Lavial F, Bouhallier F, Creveaux M, Negulescu AM, Bénard J, Janoueix-Lerosey I, Harel-Bellan A, Delattre O, Mehlen P (2014. november 1.). „Regulation by miR181 family of the dependence receptor CDON tumor suppressive activity in neuroblastoma”. Journal of the National Cancer Institute 106 (11). DOI:10.1093/jnci/dju318. PMID 25313246.
- ↑ Chen JF, Murchison EP, Tang R, Callis TE, Tatsuguchi M, Deng Z, Rojas M, Hammond SM, Schneider MD, Selzman CH, Meissner G, Patterson C, Hannon GJ, Wang DZ (2008. február 1.). „Targeted deletion of Dicer in the heart leads to dilated cardiomyopathy and heart failure”. Proceedings of the National Academy of Sciences of the United States of America 105 (6), 2111–6. o. DOI:10.1073/pnas.0710228105. PMID 18256189.
- ↑ a b Zhao Y, Ransom JF, Li A, Vedantham V, von Drehle M, Muth AN, Tsuchihashi T, McManus MT, Schwartz RJ, Srivastava D (2007. április 1.). „Dysregulation of cardiogenesis, cardiac conduction, and cell cycle in mice lacking miRNA-1-2”. Cell 129 (2), 303–17. o. DOI:10.1016/j.cell.2007.03.030. PMID 17397913.
- ↑ Thum T, Galuppo P, Wolf C, Fiedler J, Kneitz S, van Laake LW, Doevendans PA, Mummery CL, Borlak J, Haverich A, Gross C, Engelhardt S, Ertl G, Bauersachs J (2007. július 1.). „MicroRNAs in the human heart: a clue to fetal gene reprogramming in heart failure”. Circulation 116 (3), 258–67. o. DOI:10.1161/CIRCULATIONAHA.107.687947. PMID 17606841.
- ↑ van Rooij E, Sutherland LB, Liu N, Williams AH, McAnally J, Gerard RD, Richardson JA, Olson EN (2006. november 1.). „A signature pattern of stress-responsive microRNAs that can evoke cardiac hypertrophy and heart failure”. Proceedings of the National Academy of Sciences of the United States of America 103 (48), 18255–60. o. DOI:10.1073/pnas.0608791103. PMID 17108080.
- ↑ Tatsuguchi M, Seok HY, Callis TE, Thomson JM, Chen JF, Newman M, Rojas M, Hammond SM, Wang DZ (2007. június 1.). „Expression of microRNAs is dynamically regulated during cardiomyocyte hypertrophy”. Journal of Molecular and Cellular Cardiology 42 (6), 1137–41. o. DOI:10.1016/j.yjmcc.2007.04.004. PMID 17498736.
- ↑ Zhao Y, Samal E, Srivastava D (2005. július 1.). „Serum response factor regulates a muscle-specific microRNA that targets Hand2 during cardiogenesis”. Nature 436 (7048), 214–20. o. DOI:10.1038/nature03817. PMID 15951802.
- ↑ Xiao J, Luo X, Lin H, Zhang Y, Lu Y, Wang N, Zhang Y, Yang B, Wang Z (2007. április 1.). „MicroRNA miR-133 represses HERG K+ channel expression contributing to QT prolongation in diabetic hearts”. The Journal of Biological Chemistry 282 (17), 12363–7. o. DOI:10.1074/jbc.C700015200. PMID 17344217.
- ↑ Yang B, Lin H, Xiao J, Lu Y, Luo X, Li B, Zhang Y, Xu C, Bai Y, Wang H, Chen G, Wang Z (2007. április 1.). „The muscle-specific microRNA miR-1 regulates cardiac arrhythmogenic potential by targeting GJA1 and KCNJ2”. Nature Medicine 13 (4), 486–91. o. DOI:10.1038/nm1569. PMID 17401374.
- ↑ Carè A, Catalucci D, Felicetti F, Bonci D, Addario A, Gallo P, Bang ML, Segnalini P, Gu Y, Dalton ND, Elia L, Latronico MV, Høydal M, Autore C, Russo MA, Dorn GW, Ellingsen O, Ruiz-Lozano P, Peterson KL, Croce CM, Peschle C, Condorelli G (2007. május 1.). „MicroRNA-133 controls cardiac hypertrophy”. Nature Medicine 13 (5), 613–8. o. DOI:10.1038/nm1582. PMID 17468766.
- ↑ van Rooij E, Sutherland LB, Qi X, Richardson JA, Hill J, Olson EN (2007. április 1.). „Control of stress-dependent cardiac growth and gene expression by a microRNA”. Science 316 (5824), 575–9. o. DOI:10.1126/science.1139089. PMID 17379774.
- ↑ Keller T, Boeckel JN, Groß S, Klotsche J, Palapies L, Leistner D, Pieper L, Stalla GK, Lehnert H, Silber S, Pittrow D, Maerz W, Dörr M, Wittchen HU, Baumeister SE, Völker U, Felix SB, Dimmeler S, Zeiher AM (2017. július 1.). „Improved risk stratification in prevention by use of a panel of selected circulating microRNAs”. Scientific Reports 7 (1), 4511. o. DOI:10.1038/s41598-017-04040-w. PMID 28674420.
- ↑ Insull W (2009. január 1.). „The pathology of atherosclerosis: plaque development and plaque responses to medical treatment”. The American Journal of Medicine 122 (1 Suppl), S3–S14. o. DOI:10.1016/j.amjmed.2008.10.013. PMID 19110086.
- ↑ a b c d e f g h i j k l m n o p q Son DJ, Kumar S, Takabe W, Kim CW, Ni CW, Alberts-Grill N, Jang IH, Kim S, Kim W, Won Kang S, Baker AH, Woong Seo J, Ferrara KW, Jo H (2013). „The atypical mechanosensitive microRNA-712 derived from pre-ribosomal RNA induces endothelial inflammation and atherosclerosis”. Nature Communications 4, 3000. o. DOI:10.1038/ncomms4000. PMID 24346612.
- ↑ a b Basu R, Fan D, Kandalam V, Lee J, Das SK, Wang X, Baldwin TA, Oudit GY, Kassiri Z (2012. december 1.). „Loss of Timp3 gene leads to abdominal aortic aneurysm formation in response to angiotensin II”. The Journal of Biological Chemistry 287 (53), 44083–96. o. DOI:10.1074/jbc.M112.425652. PMID 23144462.
- ↑ Libby P (2002). „Inflammation in atherosclerosis”. Nature 420 (6917), 868–74. o. DOI:10.1038/nature01323. PMID 12490960.
- ↑ a b Phua YL, Chu JY, Marrone AK, Bodnar AJ, Sims-Lucas S, Ho J (2015. október 1.). „Renal stromal miRNAs are required for normal nephrogenesis and glomerular mesangial survival”. Physiological Reports 3 (10), e12537. o. DOI:10.14814/phy2.12537. PMID 26438731.
- ↑ Maes OC, Chertkow HM, Wang E, Schipper HM (2009. május 1.). „MicroRNA: Implications for Alzheimer Disease and other Human CNS Disorders”. Current Genomics 10 (3), 154–68. o. DOI:10.2174/138920209788185252. PMID 19881909.
- ↑ Amin ND, Bai G, Klug JR, Bonanomi D, Pankratz MT, Gifford WD, Hinckley CA, Sternfeld MJ, Driscoll SP, Dominguez B, Lee KF, Jin X, Pfaff SL (2015. december 1.). „Loss of motoneuron-specific microRNA-218 causes systemic neuromuscular failure”. Science 350 (6267), 1525–9. o. DOI:10.1126/science.aad2509. PMID 26680198.
- ↑ Schratt G (2009. december 1.). „microRNAs at the synapse”. Nature Reviews. Neuroscience 10 (12), 842–9. o. DOI:10.1038/nrn2763. PMID 19888283.
- ↑ Cuellar TL, Davis TH, Nelson PT, Loeb GB, Harfe BD, Ullian E, McManus MT (2008. április 1.). „Dicer loss in striatal neurons produces behavioral and neuroanatomical phenotypes in the absence of neurodegeneration”. Proceedings of the National Academy of Sciences 105 (14), 5614–19. o. DOI:10.1073/pnas.0801689105. PMID 18385371.
- ↑ Hébert SS, Papadopoulou AS, Smith P, Galas M, Planel E, Silahtaroglu AN, Sergeant N, Buée L, De Strooper B (2010. október 1.). „Genetic ablation of Dicer in adult forebrain neurons results in abnormal tau hyperphosphorylation and neurodegeneration”. Human Molecular Genetics 19 (20), 3959–69. o. DOI:10.1093/hmg/ddq311. PMID 20660113.
- ↑ Hosseinian S, Arefian A, Rakhsh-Khorshid H (2020. április 25.). „A meta-analysis of gene expression data highlights synaptic dysfunction in the hippocampus of brains with Alzheimer's disease”. Scientific Reports 10 (1), 8384. o. DOI:10.1038/s41598-020-64452-z. PMID 32433480.
- ↑ Hommers LG, Domschke K, Deckert J (2015. január 1.). „Heterogeneity and individuality: microRNAs in mental disorders”. Journal of Neural Transmission 122 (1), 79–97. o. DOI:10.1007/s00702-014-1338-4. PMID 25395183.
- ↑ Feng J, Sun G, Yan J, Noltner K, Li W, Buzin CH, Longmate J, Heston LL, Rossi J, Sommer SS (2009. július 1.). „Evidence for X-chromosomal schizophrenia associated with microRNA alterations”. PLOS ONE 4 (7), e6121. o. DOI:10.1371/journal.pone.0006121. PMID 19568434.
- ↑ Beveridge NJ, Gardiner E, Carroll AP, Tooney PA, Cairns MJ (2010. december 1.). „Schizophrenia is associated with an increase in cortical microRNA biogenesis”. Molecular Psychiatry 15 (12), 1176–89. o. DOI:10.1038/mp.2009.84. PMID 19721432.
- ↑ Stroke Facts (amerikai angol nyelven). Centers for Disease Control and Prevention , 2019. március 15. (Hozzáférés: 2019. december 5.)
- ↑ Rink C, Khanna S (2011. május 1.). „MicroRNA in ischemic stroke etiology and pathology”. Physiological Genomics 43 (10), 521–528. o. DOI:10.1152/physiolgenomics.00158.2010. PMID 20841499.
- ↑ Ouyang YB, Stary CM, Yang GY, Giffard R (2013. január 1.). „microRNAs: innovative targets for cerebral ischemia and stroke”. Current Drug Targets 14 (1), 90–101. o. DOI:10.2174/138945013804806424. PMID 23170800.
- ↑ a b c Lewohl JM, Nunez YO, Dodd PR, Tiwari GR, Harris RA, Mayfield RD (2011. november 1.). „Up-regulation of microRNAs in brain of human alcoholics”. Alcoholism: Clinical and Experimental Research 35 (11), 1928–37. o. DOI:10.1111/j.1530-0277.2011.01544.x. PMID 21651580.
- ↑ Tapocik JD, Solomon M, Flanigan M, Meinhardt M, Barbier E, Schank JR, Schwandt M, Sommer WH, Heilig M (2013. június 1.). „Coordinated dysregulation of mRNAs and microRNAs in the rat medial prefrontal cortex following a history of alcohol dependence”. The Pharmacogenomics Journal 13 (3), 286–96. o. DOI:10.1038/tpj.2012.17. PMID 22614244.
- ↑ Gorini G, Nunez YO, Mayfield RD (2013). „Integration of miRNA and protein profiling reveals coordinated neuroadaptations in the alcohol-dependent mouse brain”. PLOS ONE 8 (12), e82565. o. DOI:10.1371/journal.pone.0082565. PMID 24358208.
- ↑ Tapocik JD, Barbier E, Flanigan M, Solomon M, Pincus A, Pilling A, Sun H, Schank JR, King C, Heilig M (2014. március 1.). „microRNA-206 in rat medial prefrontal cortex regulates BDNF expression and alcohol drinking”. The Journal of Neuroscience 34 (13), 4581–8. o. DOI:10.1523/JNEUROSCI.0445-14.2014. PMID 24672003.
- ↑ Lippai D, Bala S, Csak T, Kurt-Jones EA, Szabó G (2013). „Chronic alcohol-induced microRNA-155 contributes to neuroinflammation in a TLR4-dependent manner in mice”. PLOS ONE 8 (8), e70945. o. DOI:10.1371/journal.pone.0070945. PMID 23951048.
- ↑ a b Li J, Li J, Liu X, Qin S, Guan Y, Liu Y, Cheng Y, Chen X, Li W, Wang S, Xiong M, Kuzhikandathil EV, Ye JH, Zhang C (2013. szeptember 1.). „MicroRNA expression profile and functional analysis reveal that miR-382 is a critical novel gene of alcohol addiction”. EMBO Molecular Medicine 5 (9), 1402–14. o. DOI:10.1002/emmm.201201900. PMID 23873704.
- ↑ Romao JM, Jin W, Dodson MV, Hausman GJ, Moore SS, Guan LL (2011. szeptember 1.). „MicroRNA regulation in mammalian adipogenesis”. Experimental Biology and Medicine 236 (9), 997–1004. o. DOI:10.1258/ebm.2011.011101. PMID 21844119.
- ↑ Skårn M, Namløs HM, Noordhuis P, Wang MY, Meza-Zepeda LA, Myklebost O (2012. április 1.). „Adipocyte differentiation of human bone marrow-derived stromal cells is modulated by microRNA-155, microRNA-221, and microRNA-222”. Stem Cells and Development 21 (6), 873–83. o. DOI:10.1089/scd.2010.0503. PMID 21756067.
- ↑ Zuo Y, Qiang L, Farmer SR (2006. március 1.). „Activation of CCAAT/enhancer-binding protein (C/EBP) alpha expression by C/EBP beta during adipogenesis requires a peroxisome proliferator-activated receptor-gamma-associated repression of HDAC1 at the C/ebp alpha gene promoter”. The Journal of Biological Chemistry 281 (12), 7960–7. o. DOI:10.1074/jbc.M510682200. PMID 16431920.
- ↑ Jun-Hao ET, Gupta RR, Shyh-Chang N (2016. március 1.). „Lin28 and let-7 in the Metabolic Physiology of Aging”. Trends in Endocrinology and Metabolism 27 (3), 132–141. o. DOI:10.1016/j.tem.2015.12.006. PMID 26811207.
- ↑ Zhu H, Shyh-Chang N, Segrè AV, Shinoda G, Shah SP, Einhorn WS, Takeuchi A, Engreitz JM, Hagan JP, Kharas MG, Urbach A, Thornton JE, Triboulet R, Gregory RI, Altshuler D, Daley GQ (2011. szeptember 1.). „The Lin28/let-7 axis regulates glucose metabolism”. Cell 147 (1), 81–94. o. DOI:10.1016/j.cell.2011.08.033. PMID 21962509.
- ↑ Frost RJ, Olson EN (2011. december 1.). „Control of glucose homeostasis and insulin sensitivity by the Let-7 family of microRNAs”. Proceedings of the National Academy of Sciences of the United States of America 108 (52), 21075–80. o. DOI:10.1073/pnas.1118922109. PMID 22160727.
- ↑ Teruel-Montoya R, Rosendaal FR, Martínez C (2015. február 1.). „MicroRNAs in hemostasis”. Journal of Thrombosis and Haemostasis 13 (2), 170–181. o. DOI:10.1111/jth.12788. PMID 25400249.
- ↑ a b Nourse J, Braun J, Lackner K, Hüttelmaier S, Danckwardt S (2018. november 1.). „Large-scale identification of functional microRNA targeting reveals cooperative regulation of the hemostatic system”. Journal of Thrombosis and Haemostasis 16 (11), 2233–2245. o. DOI:10.1111/jth.14290. PMID 30207063.
- ↑ Nourse J, Danckwardt S (2021. február 1.). „A novel rationale for targeting FXI: Insights from the hemostatic microRNA targetome for emerging anticoagulant strategies”. Pharmacology & Therapeutics 218, 107676. o. DOI:10.1016/j.pharmthera.2020.107676. PMID 32898547.
- ↑ Berardi E, Pues M, Thorrez L, Sampaolesi M (2012. október 1.). „miRNAs in ESC differentiation”. American Journal of Physiology. Heart and Circulatory Physiology 303 (8), H931–H939. o. DOI:10.1152/ajpheart.00338.2012. PMID 22886416.
- ↑ a b Borchers A, Pieler T (2010. november 1.). „Programming pluripotent precursor cells derived from Xenopus embryos to generate specific tissues and organs”. Genes 1 (3), 413–426. o. DOI:10.3390/ijms232314755. PMID 36499082.
- ↑ Curaba J, Spriggs A, Taylor J, Li Z, Helliwell C (2012. július 1.). „miRNA regulation in the early development of barley seed”. BMC Plant Biology 12 (1), 120. o. DOI:10.1186/1471-2229-12-120. PMID 22838835.
- ↑ Choudhary A, Kumar A, Kaur H, Kaur N (2021. május 1.). „MiRNA: the taskmaster of plant world” (angol nyelven). Biologia 76 (5), 1551–1567. o. DOI:10.1007/s11756-021-00720-1. ISSN 1336-9563.
- ↑ Waheed S, Zeng L (2020. március 1.). „The Critical Role of miRNAs in Regulation of Flowering Time and Flower Development”. Genes 11 (3), 319. o. DOI:10.3390/genes11030319. PMID 32192095.
- ↑ Wong CE, Zhao YT, Wang XJ, Croft L, Wang ZH, Haerizadeh F, Mattick JS, Singh MB, Carroll BJ, Bhalla PL (2011. május 1.). „MicroRNAs in the shoot apical meristem of soybean”. Journal of Experimental Botany 62 (8), 2495–2506. o. DOI:10.1093/jxb/erq437. PMID 21504877.
- ↑ Qureshi A, Thakur N, Monga I, Thakur A, Kumar M (2014. január 1.). „VIRmiRNA: a comprehensive resource for experimentally validated viral miRNAs and their targets”. Database 2014, bau103. o. DOI:10.1093/database/bau103. PMID 25380780.
- ↑ Kumar M: VIRmiRNA. Resource for experimental viral miRNA and their targets . Bioinformatics center, CSIR-IMTECH
- ↑ Tuddenham L, Jung JS, Chane-Woon-Ming B, Dölken L, Pfeffer S (2012. február 1.). „Small RNA deep sequencing identifies microRNAs and other small noncoding RNAs from human herpesvirus 6B”. Journal of Virology 86 (3), 1638–49. o. DOI:10.1128/JVI.05911-11. PMID 22114334.
- ↑ Lukasik A, Zielenkiewicz P (2014. június 16.). „In Silico Identification of Plant miRNAs in Mammalian Breast Milk Exosomes – A Small Step Forward?”. PLoS ONE 9 (6), e99963. o. DOI:10.1371/journal.pone.0099963. (Hozzáférés: 2024. február 23.)
- ↑ Zheng H, Fu R, Wang JT, Liu Q, Chen H, Jiang SW (2013. április 1.). „Advances in the techniques for the prediction of microRNA targets”. International Journal of Molecular Sciences 14 (4), 8179–87. o. DOI:10.3390/ijms14048179. PMID 23591837.
- ↑ Agarwal V, Bell GW, Nam JW, Bartel DP (2015. augusztus 1.). „Predicting effective microRNA target sites in mammalian mRNAs”. eLife 4, e05005. o. DOI:10.7554/eLife.05005. PMID 26267216.
- ↑ Hsu SD, Lin FM, Wu WY, Liang C, Huang WC, Chan WL, Tsai WT, Chen GZ, Lee CJ, Chiu CM, Chien CH, Wu MC, Huang CY, Tsou AP, Huang HD (2011. január 1.). „miRTarBase: a database curates experimentally validated microRNA-target interactions”. Nucleic Acids Res 39 (Adatbázisszám), D163-D169. o. DOI:10.1093/nar/gkq1107. PMID 21071411.
- ↑ Kozomara A, Griffiths-Jones S (2011. január 1.). „miRBase: integrating microRNA annotation and deep-sequencing data”. Nucleic Acids Res 39 (Adatbázisszám), D152-D157. o. DOI:10.1093/nar/gkq1027. PMID 21037258.
Fordítás[szerkesztés]
Ez a szócikk részben vagy egészben a microRNA című angol Wikipédia-szócikk ezen változatának fordításán alapul. Az eredeti cikk szerkesztőit annak laptörténete sorolja fel. Ez a jelzés csupán a megfogalmazás eredetét és a szerzői jogokat jelzi, nem szolgál a cikkben szereplő információk forrásmegjelöléseként.
További információk[szerkesztés]
- miRNA definition and classification: Ambros V, Bartel B, Bartel DP, Burge CB, Carrington JC, Chen X, Dreyfuss G, Eddy SR, Griffiths-Jones S, Marshall M, Matzke M, Ruvkun G, Tuschl T (2003. március 1.). „A uniform system for microRNA annotation”. RNA 9 (3), 277–9. o. DOI:10.1261/rna.2183803. PMID 12592000.
- Science-összefoglaló: Baulcombe D (2002. szeptember 1.). „DNA events. An RNA microcosm”. Science 297 (5589), 2002–3. o. DOI:10.1126/science.1077906. PMID 12242426.
- miRBase adatbázis
- miRTarBase Archiválva 2011. március 24-i dátummal a Wayback Machine-ben, kísérletileg igazolt miRNS-cél kölcsönhatások adatbázisa
- semirna, növénygenomban miRNS-eket kereső webalkalmazás
- ONCO.IO: Integrative resource for microRNA and transcription factors analysis in cancer.
- MirOB Archiválva 2014. március 4-i dátummal a Wayback Machine-ben.: MicroRNA targets database and data analysis and dataviz tool.
- ChIPBase database: nyilvános adatbázis a miRNS-transzkripcióban érintett vagy azokat befolyásoló transzkripciós faktorokról ChIP-seq-adatok alapján.
- An animated video of the microRNA biogenesis process.
- miRNA modulation reagents to enable up-regulation or suppression of endogenous mature microRNA function
