Proteaszóma


A proteaszómák szükségtelen vagy hibás fehérjéket peptidkötést bontó proteolízissel bontó fehérjekomplexek. Az e reakciókat segítő enzimek a proteázok.
A proteaszómák a sejtek fehérjekoncentráció-szabályzó és hibásfehérje-bontó mechanizmusát alkotják. A lebontásra jelölt fehérjékre ubikvitin kerül. E reakciót az ubikvitin-ligázok katalizálják. A fehérjére kerülő első ubikvitin jelzi a többi ligáznak, hogy további ubikvitineket helyezzen el a fehérjére. Így poliubikvitin-lánc jön létre, melyet megköt a proteaszóma, lehetővé téve a jelölt fehérje lebontását.[1] Ez 7–8 aminosavas peptideket hoz létre, melyek tovább bonthatók rövidebb aminosav-sorozatokra, illetve felhasználhatók új fehérjék szintézisére.[1]
A proteaszómák minden eukariótában és archeában, valamint néhány baktériumban megtalálhatók. Az eukariótákban a sejtmagban és a citoplazmában is találhatók proteaszómák.[2]
A proteaszóma hengeres komplex 4 egymásra illesztett gyűrűből álló központi pórust tartalmazó maggal. Minden gyűrű 7 fehérjéből áll. A két belső gyűrű 7 β-alegységből áll, 3–7 proteáz aktív helyével. Ezek a gyűrűk belső felszínén találhatók, így a célfehérjének előbb a központi pórusba kell kerülnie lebontás előtt. A két külső gyűrű 7 α-alegységből áll, melyek kaput képeznek, melyen keresztül a hengerbe kerülnek a fehérjék. Ezen alegységeket a szabályzó elemekhez való kötés irányítja, melyek a poliubikvitin-jelet ismerik fel a fehérjeszubsztrátokban, és elindítják a bontást. Az ubikvitináció és proteaszomális bontás rendszere az ubikvitin-proteaszóma rendszer.[3]
A proteaszomális bomlás a sejtben sok folyamathoz szükséges, például a sejtciklushoz, a génexpresszió szabályzásához és az oxidatív stresszre adott válaszokhoz. Fontosságát 2004-ben kémiai Nobel-díjjal ismerték el, melyet Aaron Ciechanover, Avram Hersko és Irwin Rose kapott.[4]
Felfedezés[szerkesztés]
Az ubikvitin-proteaszóma rendszer felfedezése előtt a fehérjebontást főleg a membránnal határolt, savakkal és az exogén fehérjéket és az idős vagy sérült sejtszervecskéket lebontó és újrahasznosító proteázokkal rendelkező lizoszómáknak tulajdonították.[1] Azonban Joseph Etlinger és Alfred L. Goldberg lizoszómák nélküli retikulocitákban történő ATP-dependens fehérjebontásról szóló 1977-es munkája egy másik sejten belüli bontási mechanizmus jelenlétét feltételezte.[5] Erről 1978-ban kimutatták, hogy több különböző fehérjeláncból áll, mely újdonságnak számított az akkor ismert proteázok közt.[6] A hisztonok módosításáról szóló későbbi munkákban kovalens hisztonmódosításról számoltak be a hiszton lizinje és az akkor nem ismert funkciójú ubikvitin glicinje közt.[7] 2005-ben felfedezték, hogy egy korábban proteolízissel összefüggőnek tekintett fehérje, az ATP-dependens proteolízis-faktor 1 (APF-1) azonos az ubikvitinnel.[8] E rendszer proteolitikus aktivitását többfehérjés komplexként azonosították, melyet eredetileg Sherwin Wilk és Marion Orlowski multikatalitikus proteinázkomplexnek nevezett.[9] Később felfedezték az ATP-dependens proteolitikus komplexet, mely az ubikvitindependens fehérjebontásért felel, és elnevezték 26S proteaszómának.[10][11]
Az ubikvitin-proteaszóma rendszer felfedezését lehetővé tevő munka nagy része az 1970-es évek végén és az 1980-as évek elején történt a Technionban Avram Hersko laboratóriumában, ahol Aaron Ciechanover hallgatóként dolgozott. Több éves munkássága Irwin Rose laboratóriumában a Fox Chase Rákközpontban fontos információkat nyújtott, azonban Rose később a felfedezésében játszott szerepét kisebbnek gondolta.[12] Hármuk megosztva kapta a 2004-es kémiai Nobel-díjat a rendszer felfedezéséért.[4]
Bár elektron-mikroszkópiai adatok a proteaszómák halmozott gyűrűs szerkezetét az 1980-as évek közepén igazolták,[13] a proteaszómamag szerkezetét röntgenkrisztallográfiai úton csak 1994-ben határozták meg.[14] 2018-ban a humán 26S proteaszóma holoenzim és a poliubikvitinált fehérjeszubsztrát rendszerének szerkezetét krioelektron-mikroszkópia révén határozták meg, felfedve a szubsztrát felismerésének, dezubikvitilációjának és bontásának módját.[15]
Szerkezet és elrendezés[szerkesztés]
A proteaszóma részeit gyakran Svedberg-szedimentációsegyütthatója (S) alapján nevezik. A leggyakoribb proteaszóma emlősökben a citoszol-26S proteaszóma, melynek molekulatömege mintegy 2000 kDa, egy 20S fehérje- és két 19S szabályzóegységgel. Magja üreges, a fehérjebontáshoz zárt helyet biztosít, a mag két végén lévő nyílások lehetővé teszik a fehérje bekerülését. A mag részei egy több ATPáz-aktív- és ubikvitinkötő hellyel rendelkező 19S szabályzóegységgel asszociálnak, ez teszi lehetővé a poliubikvitinált fehérjék felismerését és katalitikus magba kerülését.[15] A szabályzóegység alternatív formája a 11S részecske, mely a 19S-hez hasonlóan asszociálhat a maggal, és idegen, például vírusfertőzéskor keletkező peptidek bontásában lehet fontos.[16]
20S-mag[szerkesztés]
A 20S-mag alegységeinek száma és diverzitása fajfüggő, többsejtű élőlényekben több különböző és specializált egység van, mint egysejtűekben, és több van eukariótákban, mint prokariótákban. Minden 20S-részecske 4 heptamer gyűrűből áll, melyek két alegységből állnak, az α-egységek szerkezetiek, a β-egységek elsősorban katalitikusak. Az α-egységek az β-egységekkel homológ pszeudoenzimek. A β-egységekkel szomszédosan N-terminálisan vannak összekötve.[17] A két külső gyűrű 7 α-alegységet tartalmaz, melyek a szabályzórészecskék kapcsolódoménjei, az α-egységek N-terminusai (Pfam PF10584) a szubsztrát szabályozatlan bekerülését akadályozó kaput alkotnak.[18] A két belső gyűrű 7 β-alegységből áll, és N-terminusaikban proteázok aktív helyei vannak, melyek a proteolízist végzik.[19] Három eltérő katalitikus aktivitást azonosítottak a tiszta komplexben: chimotripszin-, tripszinszerűt és peptidilglutamilpeptid-hidrolitikust.[20] A proteaszóma mérete viszonylag állandó, körülbelül 150 × 115 Å. A belső kamra legfeljebb 53 Å széles, de a bejárat akár 13 Å is lehet, így a szubsztrátok szerkezeteinek feltehetően legalább részben fel kell bomlaniuk a bemenethez.[21]
Az archeákban, például a Thermoplasma acidophilumban minden α- és minden β-alegység azonos egymással, míg az eukariótákéi, például az élesztőéi az egyes alegységekből 7 eltérő típust tartalmaz. Emlősökben a β1, a β2 és a β5 egységek katalitikusak, és bár mechanizmusuk azonos, szubsztrátspecificitásuk eltér – chimotripszin-, tripszinszerű és peptidilglutamilpeptid-hidrolitikus (PHGH).[22] A β1i, β2i és β5i alternatív β-formákat expresszálhatják vérképző sejtek proinflammatiós sejtjelzésekre, például citokinekre, különösen a γ-interferonra válaszul. Az ezen alternatív egységekkel rendelkező proteaszóma az immunproteaszóma, melynek szubsztrátspecificitása eltér a normálistól.[21] 2016-ban az α3 magrész nélküli alternatív proteaszómát azonosítottak humán sejtekben.[23] Ezek az α4–α4 proteaszómák a hiányzó α3 egység helyén α4 egységet tartalmaznak. Ezek létét korábban igazolták élesztőben.[24] Pontos működésük nagyrészt ismeretlen, de az ezeket tartalmazó sejtek jobban ellenállnak a mérgező fémeknek, például a kadmiumnak.[23][25]
19S-szabályzórészecske[szerkesztés]
A 19S részecske eukariótákban 19 különböző fehérjéből áll, és két részből áll, az egyik egy közvetlenül a 20S maghoz kötő 9 alegységes alap, a másik 10 alegységes fedő. 6 alapfehérje az AAA családba tartozó ATPázalegység, homológjuk, a proteaszómaaktiváló nukleotidáz (PAN) megtalálható archeákban.[26] A 19S és 20S részecskék asszociációja az ATP 19S-ATPázokhoz kötését igényli, az ATP-hidrolízis a szerkezettel rendelkező ubikvitinált fehérjék bontásához szükséges. Csak a szubsztrátszerkezet felbontása igényel ATP-hidrolízisből energiát, a többi lépéshez (például komplexképzés, kapunyitás, transzlokáció, proteolízis) elegendő kizárólag az ATP-kötés.[27][28] Az ATP kötése az ATPázokhoz önmagában a szerkezet nélküli fehérjék gyors lebontását segíti. Azonban míg az ATP-hidrolízis csak a szerkezetbontáshoz kell, nem ismert, hogy ezt az energiát felhasználja-e a proteaszóma e lépések bármelyikében.[28][29]
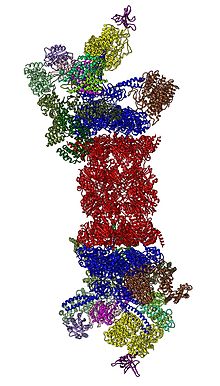
2012-ben két független kutatás a 26S proteaszóma szerkezetét egyrészecskés elektronmikroszkópiával határozta meg.[31][32] 2016-ban három független kutatás először határozta meg a humán 26S proteaszóma közel atomi felbontású szerkezetét szubsztrát nélkül krioelektron-mikroszkópiával.[33][34][35] 2018-ban egy kutatásban kiderítették a dezubikvitiláció, a transzlokáció-iniciáció és a processzív szerkezetbontás mechanizmusát 7 szubsztráttal rendelkező 26S proteaszóma szerkezete révén.[15] A 19S-ben a 20S-sel szomszédosan vannak az AAA-ATPázok, melyek Rpt1/Rpt2/Rpt6/Rpt3/Rpt4/Rpt5 sorrendben heterohexamert alkotnak. Ez az Rpt1/Rpt2, Rpt6/Rpt3 és Rpt4/Rpt5 dimerek trimerje, mely N-terminális szerkezeteik mentén jön létre. Ezek a hexamer gyűrűből kiemelkednek. A legnagyobb szabályzó nem ATPázok az Rpn1 és Rpn2, melyek az Rpt1/2, illetve az Rpt6/3 fehérjékhez kötnek. Az ubikvitinreceptor Rpn13 az Rpn2-höz köt, lezárva ezt az alkomplexet. A fedő az AAA-ATPáz-hexamer egyik felét (Rpt6/3/4) fedi le, és közvetlenül érintkezik a 20S-sel az Rpn6 és kisebb mértékben az Rpn5 révén. Az Rpn9, Rpn5, Rpn6, Rpn7, Rpn3 és Rpn12 szerkezetileg egymáshoz és a COP9 szignaloszóma és az eukarióta iniciációs faktor 3 egységeihez kötnek, melyek az Rpn8/11 heterodimert körülzáró patkószerkezetben állnak össze. Az Rpn11, a dezubikvitináló enzim az AAA-ATPáz hexamer nyílásán van, lehetővé téve a szubsztrátokról az ubikvitin eltávolítását bekerülésük előtt. Az Rpn10 a fedő perifériáján, az Rpn8 és Rpn9 mellett van.
A 19S konformációs változásai[szerkesztés]
A 19S szabályzórészecskét a 26S proteaszóma holoenzimjében szubsztrát nélkül 6 erősen eltérő konformációs állapotban azonosították.[36][37] Az alacsony energiájú AAA-ATPáz-konfigurációban a domének lépcsőzetesen vagy alátétszerűen találhatók.[30][31] ATP jelenlétében szubsztrát nélkül 3 alternatív, kevésbé gyakori konformáció jelenik meg, melyek elsősorban a fedél elhelyezkedésében térnek el az AAA-ATPáz-modulhoz viszonyítva.[33][37] ATP-γS vagy szubsztrát jelenlétében sokkal több konfiguráció ismert az AAA-ATPáz jelentős szerkezeti változásaival.[15][36][38][39] Egyes szubsztrátkötött változatok hasonlítanak a szubsztrátmentesekhez, de nem azonosak, különösen az AAA-ATPázban.[15][36] A 26S létrejötte előtt megfigyelték a szabad 19S szabályzórészecskét 7 konformációs állapotban.[40] Ezek kissé eltérnek, és jellemzőik is eltérnek. Így a 19S legalább 20 konformációs állapotot mutathat eltérő fiziológiai körülmények közt.
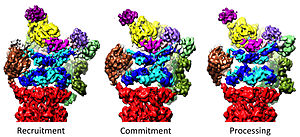
A 20S szabályzása a 19S által[szerkesztés]
A 19S szabályzórész felel a 20S fehérjebontásának stimulálásáért. A 19S szabályzó ATPázainak elsődleges funkciója a 20S kapujának nyitása, mely akadályozza a szubsztrátok bekerülését.[41] A proteaszóma-ATPáz kapunyitási mechanizmusát 2007-ben igazolták.[18] A 20S-kapunyitás, így a szubsztrátbontás is, a proteaszóma-ATPázok C-terminusait igényli, melyek seciális HbYX motívumot tartalmaznak. Ezek a 20S tetején lévő üregekbe kötnek, és az ATPázt a 20S proteolitikus komplexbe rögzítik, összekötve a szubsztrátszerkezet-bontó szerkezetet a 20S bontószerkezetével. Maga e kötés a kulcshoz hasonlóan nyitja a 20S kapuját.[18] Ennek pontos mechanizmusa a humán 26S proteaszómánál közel atomi felbontásban ismert, így feltehetően az Rpt1/2/3/5/6 ATPázalegység C-terminusainak a 20S felszínébe való kötése kell a teljes nyitáshoz.[36][15][33]
További szabályzórészecskék[szerkesztés]
A 20S proteaszómák asszociálhatnak a 11S szabályzórészecskével is, ez ATPáz nélküli heptamer, mely rövid peptidek bontását segítheti, de teljes fehérjékéit nem. Feltehetően ennek oka a nagyobb szubsztrátok szerkezetének bontására való képesség hiánya. Ezt nevezik PA28-nak, REG-nek vagy PA26-nak is.[17] Az alegységek C-terminusához való kötés és az α-gyűrű konformációs változása a 20S-kapu nyitásához hasonló a 19S-éhez.[42] A 11S expresszióját a γ-interferon indukálja, és a fő hisztokompatibilitási komplexhez kötni képes peptideket hoz létre.[16]
További nem ATPáz szabályzórészecske a Blm10 (élesztő) vagy a PA200/PSME4 (ember). A 20S-kapuban 1 α-egységet nyit, és kupolává hajlik kis pórussal felette.[17]
Keletkezés[szerkesztés]
A proteaszóma keletkezése összetett az aktív komplexhez szükséges alegységek száma miatt. A β-egységek N-terminális propeptidekből jönnek létre, melyek poszttranszlációsan módosulnak a 20S összeállásakor a proteolitikus aktív hely felszínre kerüléséhez. A 20S két félproteaszómából áll, melyekben 7 tagú pro-β gyűrű kapcsolódik 7 tagú α-gyűrűhöz. A félproteaszómák β-gyűrűinek összeállása a propeptidek treonindependens autolízisét okozza, megmutatva az aktív helyet. E β-kölcsönhatásokat szupramolekuláris sóhidak és az α-hélixek közti hidrofób kölcsönhatások mediálják, melyek mutációi csökkentik a proteaszóma összeállási képességét.[43] A félproteaszómák összeállását az α-alegységek heptamerizálódása indítja, mely a megfelelő pro-β-gyűrűhöz alkot sablont. Az α-alegységek összeállását nem jellemezték.[44]
2009-ig a 19S szabályzórészecske létrejöttének folyamatának nagy része ismert. A 19S két önálló alkomponensként (alap, fedő) jön létre. Az alap összeállását 4 chaperon (S5b, p27, PAAF1 és gankirin) könnyítik.[45] Ezek az AAA-ATPázokhoz kötnek, fő működésük feltehetően a heterohexamer AAA-ATPáz-gyűrű megfelelő összeállásának biztosítása. Még nem ismert, hogy az alap függetlenül jön-e létre, hogy ennek sablonja-e a 20S mag, és hogy van-e alternatív összeállási út. A 4 chaperon mellett a dezubikvitináló Usp14 is segíti az alap összeállását, de nem szükséges.[46] A fedő ettől függetlenül meghatározott sorrendben áll össze chaperon nélkül.[47]
Fehérjebontó folyamat[szerkesztés]

Ubikvitináció és célzás[szerkesztés]
A fehérjék proteaszóma általi bontása egy lizin kovalens módosításával történik, melyhez 3 enzim koordinált reakciója szükséges. Először egy ubikvitinaktiváló enzim (E1) hidrolizálja az ATP-t és ubikvitint adenilál. Ez ezután az E1 aktív helyen lévő ciszteinjére kerül egy második ubikvitin adenililációjával együtt.[48] Ezután ez az ubikvitinkonjugáló enzim (E2) ciszteinjére kerül. Végül egy erősen diverz enzimosztály, az ubikvitin-ligázok egy tagja (E3) felismeri az ubikvitinálandó fehérjét, és katalizálja az ubikvitin átvitelét az E2-ről a célfehérjére. A célfehérje legalább 4 ubikvitinnel jelölendő, hogy a proteaszóma felismerje.[49] It is therefore the E3 that confers substrate specificity to this system.[50] Az E1, E2 és E3 fehérjék száma fajtól és sejttípustól függ, de több különböző E3 enzim van emberben, ami azt jelenti, hogy az ubikvitin-proteaszóma rendszernek sok célpontja van.
A poliubikvitinált fehérje proteaszómához kerülésének mechanizmusa nem teljesen ismert. A poliubikvitinált fehérjéhez kötött proteaszómáról szóló néhány nagy felbontású pillanatkép alapján az ubikvitinreceptorok feltehetően az Rpn11 dezubikvitinázzal vannak koordinálva a kezdeti szubsztrátcélzáshoz és -bevitelhez.[15] Az ubikvitinreceptorokon N-terminális ubikvitinszerű (UBL) és egy vagy több ubikvitinasszociált (UBA) domén van. Az UBL doméneket a 19S szabályzóegységek felismerik, az UBA-k háromhélixes kötéssel kötnek ubikvitint. E receptorok segíthetik a poliubikvitinált fehérjéket a proteaszómához, de ennek módja nem ismert.[51]
Az ubikvitin 76 aminosavból áll, neve onnan ered, hogy mindenhol jelen van erősen állandósult szekvenciája miatt, és minden ismert eukariótában megtalálható.[52] Az ubikvitint kódoló gének az eukariótákban tandem ismétlődésekben vannak, feltehetően e gének erős transzkripciós igényei miatt a sejtnek az elegendő ubikvitin termeléséhez. Feltehetően az ubikvitin az egyik leglassabban változó fehérje.[53] Az ubikvitin 7 lizint tartalmaz, melyhez további ubikvitin csatlakozhat, különböző poliubikvitin-láncokat alkotva.[54] Az előző ubikvitin 48. lizinjéhez kötött ubikvitint tartalmazó fehérjék fontosak a proteaszómás bontásban, a többi lánctípusok más folyamatokban lehetnek fontosak.[55][56]

Dezubikvitiláció[szerkesztés]
A proteaszomális bontásra jelölt fehérje ubikvitinjeit általában a 3 proteaszómaasszociált dezubikvitáló enzim (DUB) egyike, az Rpn11, az USP14 vagy az UCH37 eltávolítja, újrahasznosítva az ubikvitint, és az ubikvitinszintet fenntartva.[56] Az Rpn11 a 19S belső, sztöchiometriai alegysége, és a 26S proteaszómához szükséges. DUB-aktivitása a monomer formájáénál nagyobb. Ubikvitineltávolító módszerét az EB konformációjú proteaszómában figyelték meg.[15] Ez megmutatja, hogy a DUB-aktivitás az AAA-ATPáz általi szubsztrátfelismeréssel összefügg. Az Rpn11-gyel szemben az USP14 és az UCH37 nem mindig asszociálnak a proteaszómával. A sejtekben a proteaszómák 10–40%-a tartalmaz USP14-et. Az USP14 és az UCH37 is nagyrészt a proteaszómával aktiválódik, és csekély a saját DUB-aktivitása. Aktiválása után az USP14 gátolja a proteaszóma működését DUB-aktivitásával és párhuzamos proteaszómakonformációs átmenetek indukálásával, melyek egyike megakadályozza a szubsztrát AAA-ATPázba kerülését, melyet krioelektron-mikroszkópiával figyeltek meg.[57] Az USP14 a proteaszómaműködést több ellenőrzőponton szabályozza, mivel katalitikusan verseng az Rpn11-gyel, és allosztérikusan módosítja az AAA-ATPáz állapotát.[57] Ezek alapján a proteaszóma szabályzása függhet konformációs állapotai dinamikus átmenetétől.
Kibontás és transzlokáció[szerkesztés]
Egy fehérje ubikvitinációja után a 19S ATP-dependens kötéssel felismeri.[15][28] Ezután a szubsztrát bekerül a 20S alegységbe, hogy érintkezzék a proteolitikus aktív helyekkel. Mivel a 20S részecske központi csatornája szűk, és az α-alegységek N-terminusai határolják, a szubsztrátoknak bekerülés előtt legalább részben fel kell bomlaniuk.[15] Ez a bekerülés a transzlokáció, és a dezubikvitináció után történik.[15][28] Azonban a dezubikvitináció és a felbontás sorrendje nem ismert.[58] Hogy melyik a sebességmeghatározó lépés, a szubsztráttól függ, egyes fehérjéknél a felbontás az, más fehérjéknél a dezubikvitináció.[27] A szubsztrátok felbontásának mértéke a transzlokáció előtt feltehetően 20 aminosav a szubsztráttal rendelkező 26S proteaszóma szerkezete alapján a dezubikvitilációkompatibilis állapotban,[15] de a jelentős harmadlagos szerkezet, és különösen a nem lokális kölcsönhatások, például a diszulfidkötések gátolják a lebontást.[59] A kellő méretű rendezetlen szakaszok hossza feltehetően segíti a lebontás elindítását.[60][61]
Az α-alegységek alkotta kapu megakadályozza a nagyjából 4 aminosavnál hosszabb peptidek bekerülését a 20S-be. A felismerés előtt kötött ATP-molekulák a transzlokáció előtt hidrolizálódnak. Míg a szerkezet felbontásához energia kell, a transzlokációhoz nem.[27][28] A 26S proteaszóma képes nem hidrolizálható ATP-analóggal a felbontott fehérjéket lebontani, de felbontatlanokat nem, így az ATP-hidrolízisből származó energia szükséges a szubsztrát felbontásáhozg.[27] A felbontott szubsztrát áthaladását a nyitott kapun a könnyített diffúzió teszi lehetővé, ha a 19S elemhez ATP köt.[62]
A globuláris fehérjék felbontásának mechanizmusa általános, de aminosavszekvencia-függő. A hosszú glicin-alanin szekvenciák gátolják a szubsztrát felbontását gátolják, csökkentve a proteaszómás bontás hatékonyságát, részben bontott melléktermékeket feltehetően az ATP-hidrolízis és a felbontás lépései közti kapcsolat hiánya miatt.[63] Ilyen glicin-alanin ismétlődések vannak a természetben, például a selyem fibroinjában, továbbá az Epstein–Barr-vírus egyes ilyen szakaszt tartalmazó géntermékei leállíthatják a proteaszómát, segítve a vírus terjedését az antigénprezentáció megakadályozásával a fő hisztokompatibilitási komplexen.[64]

Proteolízis[szerkesztés]
A proteaszóma endoproteázként működik.[65][66][67][68] A β-alegységek általi proteolízis mechanizmusa treonindependens nukleofil támadással történik. Ez asszociált víztől is függhet, mely deprotonálja a treonin hidroxilcsoportját. A bontás a központban történik, melyet a két β-gyűrű alkot, és nem bocsát ki részben lebontott termékeket, ehelyett rövid, 7–9 aminosavból álló polipeptidekre bontja a szubsztrátot, azonban ez élőlénytől és szubsztráttól függően 4–25 aminosav közt változhat. A termékhosszt meghatározó biokémiai mechanizmus nem teljesen ismert.[69] Bár a 3 katalitikus β-alegység mechanizmusa közös, szubsztrátspecificitásuk eltér, ezek chimotripszin-, tripszin- és peptidil-glutamilpeptid-hidroláz-szerű (PHGH). E specificitáseltérések az aminosavakkal való atomközi érintkezések eredményei az alegységek aktív helyei közelében. A katalitikus β-alegységek a proteolízishez szükséges állandósult lizint is tartalmaznak.[22]
Bár a proteaszóma normál esetben rövid peptidrészleteket tartalmaz, egyes esetekben ezek biológiailag aktívak és működőképesek. Egyes bizonyos gének expresszióját szabályzó transzkripciós faktorok, beleértve az NF-κB egy részét is, inaktív prekurzorként jönnek létre, melyeket az ubikvitináció, majd a proteaszomális bontás tesz aktívvá. Ez az aktivitás a szubsztrát belső proteaszomális bontását igényli a processzív helyett. A hosszú gyűrűk e fehérjéken feltehetően proteaszómaszubsztrátok, melyek középre kerülnek, míg a fehérje legnagyobb része kívül marad.[70] Hasonló hatást figyeltek meg élesztőben is, ez a szabályzott ubikvitin/proteaszóma-dependens feldolgozás (RUP).[71]
Ubikvitinfüggetlen bontás[szerkesztés]
Bár a legtöbb proteaszómaszubsztrát bontás előtt ubikvitinációt igényel, ez alól vannak kivételek, különösen ha a proteaszóma normál szerepet játszik a poszttranszlációs fehérjefeldolgozásban. Az NF-κB p105 p50-re való feldolgozása és belső proteolízis révén történő aktiválása fontos példa erre.[70] Egyes belső szerkezet nélküli régiók miatt feltehetően instabil fehérjéket[72] is ubikvitinfüggetlenül bont. Fontos példa ubikvitinfüggetlen proteaszómaszubsztrátra az ornitin-dekarboxiláz.[73] Ismertek ubikvitinfüggetlen sejtciklusszabályzó fehérjék is, példa erre a p53, azonban ez ubikvitindependensen is bomlik.[74] A hibás szerkezetű vagy erősen oxidált fehérjék ubikvitin- és 19S-independensen bomlanak sejtstressz esetén.[75]
Evolúció[szerkesztés]

A 20S proteaszóma mindenhol jelen van, és fontos az eukariótákban és az archeákban. A baktériumok Actinomycetales rendjében is vannak 20S-homológok, a legtöbb baktériumban jelen van továbbá a hslV és a hslU hősokkgén, melyek terméke kétrétegű gyűrűt és ATPázt tartalmazó multimer proteáz.[76] A hslV feltehetően a 20S proteaszóma ősére hasonlít.[77] Azonban a HslV nem szükséges a baktériumoknak, és nincs minden baktériumban, azonban egyes protiszták rendelkeznek a 20S és a hslV rendszerrel is.[76] Sok baktériumban más homológok és megfelelő ATPázok is jelen vannak, például a ClpP és ClpX. Ez magyarázhatja, miért nem szükséges a HslUV-rendszer.
Szekvenciaanalízis alapján a β-alegységke a főleg szerkezeti α-nál korábban váltak szét. A 20S proteaszómát expresszáló baktériumokban a β-alegységek szekvenciái az archeákéira és eukariótákéira nagyon hasonlítanak, míg az α-alegységekéi kevésbé. A 20S proteaszómák jelenléte a baktériumokban feltehetően horizontális géntranszfer eredménye, míg az alegységek eukariótákban lévő diverzifikációja több génduplikáció miatt történt.[76]
Sejtciklus-irányítás[szerkesztés]
A sejtciklus előrehaladását a ciklindependens kinázok (CDK) irányítják, melyeket specifikus ciklinek aktiválnak, melyek a sejtciklus szakaszait határolják. A csak néhány percig a sejtben lévő mitotikus ciklinek élettartama az egyik legrövidebb a sejtbeli fehérjék közt.[1] Miután egy CDK–ciklin komplex elvégezte működését, a megfelelő ciklin poliubikvitinálódik, és a proteaszóma lebontja, irányítva a sejtciklust. Például a mitózisból való kilépéshez a mitózissegítő faktorba tartozó ciklin B proteaszómadependens disszociációja kell.[78] Gerincessejtekben a mitotikus ellenőrzőponton való, az M-fázisból való idő előtti kilépést okozó „átcsúszás”, az orsó-ellenőrzőpont általi késleltetés ellenére is megtörténhet.[79]
A korábbi sejtciklus-ellenőrzőpontok, például a G1 és az S fázis közti posztrestrikciós ellenőrző pont ugyanígy a ciklin A bontását igényli, ennek ubikvitinációjához anafázissegítő komplex (APC) kell, mely E3-ubikvitin-ligáz.[80] Az APC és az Skp1/Cul1/F-box fehérjekomplex (SCF komplex) a ciklinbontás és ellenőrzőpont-irányítás két fontos szabályzója, az APC az adaptorprotein Skp2 ubikvitinációjával irányítja az SCF komplexet, megakadályozva annak aktivitását a G1–S átmenet előtt.[81]
A 19S részecske egyes tagjainak is saját szabályzószerepük van. Az onkoprotein gankirin egyike a 19S alkomponenseinek, mely erősen köti a CDK4-et, és fontos az ubikvitil-p53 felismerésében az MDM2 ubikvitinligázhoz való affinitása révén. A gankirin antiapoptotikus, és egyes tumorok, például a májsejtes karcinóma túlexpresszálják.[82]
Az eukariótákhoz hasonlóan egyes archeák is a proteaszómával irányítják sejtciklusukat az ESCRT-III-mediált sejtosztódás irányításával.[83]
Növénynövekedés-szabályzás[szerkesztés]
A növényekben az auxinok általi jelzés számos transzkripciósfaktor-represszor proteaszomális bontásra való jelölését indukálja. Ezeket az SCFTIR1 (az SCF és a TIR1 auxinreceptor komplexe) ubikvitinálják. Az Aux/IAA-fehérjék bomlása derepresszálja az auxinválasz-faktor (ARF) család transzkripciós faktorait, és indukálja az ARF-irányított génexpressziót.[84] Hatásai függnek a növény típusától és fejlődési szakaszától, de fontosak a gyökerek és a levélerek növekedésének irányításában. Az ARF-derepressziós választ feltehetően az ARF- és Aux/IAA-fehérjék kapcsolódási specificitása mediálja.[85]
Apoptózis[szerkesztés]
A belső és külső jelzések is apoptózisindukcióhoz vezethetnek. A sejtalkotók ily lebontását elsősorban speciális proteázok, a kaszpázok végzik, de a proteaszóma fontos az apoptózisban is. A proteaszóma részvételét e folyamatban a nagyobb fehérjeubikvitináció és E1-, E2- és E3-koncentráció mutatják, melyek az apoptózis előtt jóval láthatók.[86][87][88] Az apoptózis során a magi proteaszómák a külső membrán blebjeihez transzlokálnak.[89]
A proteaszómagátlás az apoptózisindukción különböző sejttípusokban eltérően hat. Általában nem szükséges a proteaszóma az apoptózishoz, de gátlása a legtöbb tanulmányozott sejttípusban proapoptotikus. Az apoptózist a növekedéssegítő sejtciklusfehérjék bomlásának zavara mediálja.[90] Azonban egyes sejtvonalakban – például kvieszcens és differenciált sejtek, például timociták és neuronok – primer kultúrájában a proteaszómagátlás akadályozza az apoptózist. Ennek mechanizmusa nem ismert, de feltehetően csak kvieszcens állapotú sejtekre jellemző, vagy a JNK proapoptotikus kináz eltérő aktivitása okozhatja.[91] A proteaszómagátlók gyorsan osztódó sejtekben történő apoptózisokozása néhány újabb kemoterápiás szerben, például a bortezomibban és a szalinosporamid A-ban használatos.
Sejtstresszre adott válasz[szerkesztés]
Sejtstresszekre – például fertőzésre, hősokkra vagy oxidatív stresszre – válaszolva a hibás szerkezetű fehérjéket azonosító és proteaszomális bontásra jelölő hősokkproteinek expresszálódnak. A Hsp27 és a Hsp90 chaperonok is összefüggnek az ubikvitin-proteaszóma rendszer aktivitásának növelésében, bár nem közvetlen résztvevők.[92] A Hsp70 ezzel szemben a kitett hidrofób részekhez köt a hibás fehérjék felszínén, és E3 ubikvitinligázokat, például CHIP-et aktivál a fehérjék proteaszomális bontásra való jelöléséhez.[93] A CHIP-et (Hsp70-kölcsönhatófehérje karboxilterminusa) arotein) az E3 enzim és az E2 kötőpartner közti kölcsönhatások szabályozzák.[94]
Hasonló mechanizmusok segítik az oxidatívan sérült fehérjék lebontását a proteaszóma-rendszer révén. A magi proteaszómákat a PARP szabályozza, és a nem megfelelően oxidált hisztonokat bontják le.[95] A gyakran nagy amorf aggregátumokat alkotó oxidált fehérjéket közvetlenül a 20S mag bonthatja le a 19S szabályzóelem nélkül, és nem igényelnek ATP-hidrolízist vagy ubikvitines jelölést.[75] Azonban a nagymértékű oxidatív károsodás növeli a fehérjerészek közti keresztkötések kialakulását, proteolízissel szemben ellenállóvá téve őket. Az ilyen oxidált aggregátumok nagyobb mennyisége összefügg az öregedéssel.[96]
Az ubikvitin-proteaszóma rendszer hibás szabályzása számos idegrendszeri betegségben közreműködhet. Agytumorokhoz (például asztrocitóma) vezethet.[97] Néhány késői megjelenésű neurodegeneratív betegség, melyek közös jellemzői a hibás fehérjék, például a Parkinson-kór és az Alzheimer-kór, nagy oldhatatlan hibásfehérje-aggregátumok jöhetnek létre, melyek neurotoxicitást okozhatnak ismeretlen mechanizmus miatt. A csökkent proteaszómaaktivitás lehet az aggregátumok és Lewy-testek keletkezésének oka a Parkinson-kórban.[98] Ezt támasztja alá, hogy a Parkinson-kór élesztőmodelljei érzékenyebbek az α-szinuklein, a Lewy-testek legfőbb összetevője toxicitására alacsony proteaszómaaktivitás esetén.[99] Szintén ez okozhatja az autizmus-spektrumzavart és bizonyos izom- és idegrendszeri betegségeket, például az inklúziós testes miopátiát.[97]
Az immunitásban[szerkesztés]
A proteaszóma szerepe egyszerű, de fontos az adaptív immunitásban. A peptidantigéneket a fő hisztokompatibilitási komplex I. osztályú fehérjéi mutatják az antigénprezentáló sejtekben. Ezek a patogén proteaszomális bontásának termékei. Bár a kis mennyiségben expresszált proteaszómák részt vehetnek e folyamatban, egy speciális fehérjekomplex, melynek expresszióját a γ-interferon indukálja, az MHC-kötéshez optimális méretű és összetételű peptidek elsődleges termelője. E fehérjék, melyek expressziója immunválasz során megnő, például a 11S szabályzófehérje, mely szabályozza az MHC-ligandum-termelést, valamint a β1i, a β2i és a β5i egységek. Ezenk az immunproteaszómát alkotják.[16] Egy másik β5i-variánst, a β5t-t a csecsemőmirigy expresszál (timoproteaszóma), de funkciója ismeretlen.[100]
Az MHC I-hez való kötés ereje függ a ligandum C-terminális összetételétől, mivel a peptidek hidrogénkötéssel és a B-üreggel való közeli érintkezésekkel kötnek az MHC felszínén. Sok MHC I-allél preferálja a hidrofób C-terminális aminosavakat, és az immunproteaszóma nagyobb valószínűséggel hoz létre hidrofób C-terminusokat.[101]
Az aktív NF-κB létrehozásában való fontossága miatt a proteaszómák aktivitása összefügg a gyulladásos és az autoimmun betegségekkel. A nagyobb proteaszómaaktivitás összefügg a betegségek aktivitásával és az autoimmun betegségekkel, például a szisztémás lupusszal és a reumatoid arthritisszel.[16]
A proteaszóma fontos az antitesthez kötött virionok sejten belüli antitestmediált proteolízisében. Ekkor a TRIM21 immunglobulin G-hez köt, a viriont a proteaszómához irányítva, ahol az lebomlik.[102]
Proteaszómagátlók[szerkesztés]


A proteaszómagátlók fontos tumorellenes aktivitással rendelkeznek sejttenyészetben a növekedést segítő sejtciklusfehérjék bontását megzavarva apoptózist okoznak bennük.[90] E szelektív apoptózisindukció hatékonynak bizonyult állatmodellben és humán kísérletekben is.
A laktacisztin, a Streptomyces baktériumok által szintetizált természetes termék volt az első ismert nem peptid proteaszómagátló,[103] és gyakran használják biokémiai és sejtbiológiai kutatásokban. A laktacisztint a Myogenics/Proscript szabadalmaztatta, melyet a Millennium Pharmaceuticals vásárolt fel, mely a Takeda Pharmaceuticals része. A laktacisztin a proteaszóma katalitikus β-alegységeinek, különösen a proteaszóma chimotripszinszerű aktivitásáért felelős β5 N-terminális treoninját módosítja. Ez alapján a proteaszóma treoninproteáz.
A bortezomib (boronált MG132), melyet a Millennium Pharmaceuticals fejlesztett ki, és Velcade néven forgalmaz, az első kemoterápiás anyagként klinikailag használt proteaszómagátló.[104] A mielóma multiplex kezelésére használják.[105] A mielóma multiplex nagyobb proteaszómaeredetűpeptid-szintet okoz a vérszérumban, mely sikeres kemoterápia esetén normál értékre csökken.[106] Állatkísérletek igazolták, hogy a bortezomib klinikailag jelentős hatással rendelkezik hasnyálmirigyrákban.[107][108] Pre- és korai klinikai kutatásokban vizsgálták a bortezomib hatásosságát más B-sejt-alapú rákokban,[109] különös tekintettel néhány nem Hodgkin-limfóma-típusra.[110] A klinikai eredmények megerősítik a proteaszómagátló kemoterápiával való együttes használatát B-sejtes akut limfoblasztos leukémiában.[111] A proteaszómagátlók képesek glükokortikoid-rezisztens leukémiasejt-tenyészeteket elölni.[112]
A ritonavir proteázgátló, melyet HIV-fertőzés ellen használtak. Azonban kimutatták, hogy a proteaszómák chimotripszinszerű aktivitását gátolja, míg a tripszinszerűt kissé erősíti.[113] Állatkísérletek szerint a ritonavir gátolhatja a gliomasejtek növekedését.[114]
A proteaszómagátlók ezenkívül autoimmun betegségek kezelésében is hasznosak lehetnek. Például humán bőrgraftokkal rendelkező egerekben kisebbek lettek a pszoriázisos léziók proteaszómagátlóval való kezelés után.[115] Ezenkívül az asztma rágcsálómodelljeiben is hasznosnak bizonyultak.[116]
A proteaszóma jelölése és gátlása fontos ezenkívül a proteaszómaaktivitás in vitro és in vivo tanulmányozásában is. A leggyakrabban használt gátlók a laktacisztin és a Goldberg laboratórium által fejlesztett MG132. Fluoreszcens gátlókat is létrehoztak a proteaszóma aktív helyeinek megjelölésére.[117]
Klinikai jelentőség[szerkesztés]
A proteaszóma és alegységei legalább 2 okból fontosak klinikailag:
- a hibás komplex vagy a rosszul működő proteaszóma bizonyos betegségekkel összefügg,
- felhasználhatók gyógyszercélpontként terápiás beavatkozásokban. 2009-től további kísérleteket végeztek a proteaszóma vizsgálatához új diagnosztikai markerek és stratégiák fejlesztésében. A proteaszómák patofiziológiájának jobb megértése klinikailag használható gyógyszerekhez vezethet.
A proteaszómák az ubikvitin-proteaszóma rendszer[118] és a megfelelő fehérjeminőség-ellenőrzés fontos részei. A fehérjeubikvitináció és a proteolízis fontosak a sejtciklus, a sejtnövekedés és -differenciáció, a géntranszkripció, a jeltranszdukció és az apoptózis terén.[119] A hibás proteaszómák csökkent proteolízishez és a hibás fehérjék növekedéséhez vezet, mely közreműködhet neurodegeneratív,[120][121] cardiovascularis,[122][123][124] gyulladásos és autoimmun betegségekben[125] szisztémás DNS-károsodási válaszokhoz és daganatokhoz.[126]
A hibás UPS összefügg számos neuro- és miodegeneratív betegséggel, például az Alzheimer-kórral,[127] a Parkinson-kórral,[128] a Pick-kórral,[129] az amiotrófiás laterálszklerózissal,[129] a Huntington-kórral,[128] a Creutzfeldt–Jakob-kórral[130] és más mozgatóideg-, poliglutamin-betegségekkel (poliQ), izomdisztrófiákkal[131] és néhány demenciával összefüggő ritka neurodegeneratív betegséggel.[132] Az UPS részeként a proteaszóma fenntartja a szív fehérje-homeosztázisát, így fontos a szív ischaemiás sérülése,[133] ventricularis hipertrófia[134] és szívelégtelenség esetén.[135] Ezenkívül az UPS fontos a malignus átalakulásban. Az UPS-proteolízis a ráksejtek rák kialakulásához szükséges stimulációs jeleiben fontos, melyek a rák kialakulásához fontosak. Ennek megfelelően a transzkripciós faktorok, például a p53, a c-jun, a c-Fos, az NF-κB, a MYC, a HIF-1α, a MATα2, a STAT3, a szterolszabályzottelem-kötő fehérjék és az androgénreceptorok bomlását is az UPS szabályozza, így érintettek bizonyos ráktípusokban.[136] Ezenkívül a tumorszupresszorgén-termékeket, például az adenomatosus polyposis colit (APC) colorectalis rák és a von Hippel–Lindau-tumorszupresszort (VHL) retinoblasztóma esetén, valamint számos protoonkogént (Raf, Myc, MYB, Rel, Src, MOS, ABL). Ezenkívül befolyásolja a gyulladásos válaszokat is. Ezt elsősorban az NF-κB-aktivációnak tulajdonítják, segítve a proinflammatiós citokinek, például a TNF-α, az IL-β, az IL-8, az adhéziós fehérjék (ICAM-1, VCAM-1, P-szelektin), a prosztaglandinok és a nitrogén-monoxid (NO) termelését.[125] Ezenkívül da leukocitaproliferációt is segíti, legfőképp a ciklinek és a CDK-gátlók bontása révén.[137] Végül a szisztémás lupusos, Sjögren-szindrómás és a reumatoid arthritises (RA) betegekben elsősorban a vérkeringésben lévő proteaszómák klinikai biomarkerek.[138]
Jegyzetek[szerkesztés]
- ↑ a b c d Lodish H, Berk A, Matsudaira P, Kaiser CA, Krieger M, Scott MP, Zipursky SL, Darnell J. 3, Molecular cell biology, 5th, New York: W.H. Freeman and CO, 66–72. o. (2004). ISBN 978-0-7167-4366-8
- ↑ Peters JM, Franke WW, Kleinschmidt JA (1994. március 1.). „Distinct 19 S and 20 S subcomplexes of the 26 S proteasome and their distribution in the nucleus and the cytoplasm”. The Journal of Biological Chemistry 269 (10), 7709–18. o. DOI:10.1016/S0021-9258(17)37345-3. PMID 8125997.
- ↑ Nassif, Nicholas D. (2014. május 1.). „Slipping up: Partial substrate degradation by ATP-dependent proteases”. IUBMB Life 66 (5), 309–317. o. DOI:10.1002/iub.1271. PMID 24823973.
- ↑ a b Nobel Prize Committee: Nobel Prize Awardees in Chemistry, 2004, 2004. (Hozzáférés: 2006. december 11.)
- ↑ Etlinger JD, Goldberg AL (1977. január 1.). „A soluble ATP-dependent proteolytic system responsible for the degradation of abnormal proteins in reticulocytes”. Proceedings of the National Academy of Sciences of the United States of America 74 (1), 54–8. o. DOI:10.1073/pnas.74.1.54. PMID 264694.
- ↑ Ciehanover A, Hod Y, Hershko A (1978. április 1.). „A heat-stable polypeptide component of an ATP-dependent proteolytic system from reticulocytes”. Biochemical and Biophysical Research Communications 81 (4), 1100–5. o. DOI:10.1016/0006-291X(78)91249-4. PMID 666810.
- ↑ Goldknopf IL, Busch H (1977. március 1.). „Isopeptide linkage between nonhistone and histone 2A polypeptides of chromosomal conjugate-protein A24”. Proceedings of the National Academy of Sciences of the United States of America 74 (3), 864–8. o. DOI:10.1073/pnas.74.3.864. PMID 265581.
- ↑ Ciechanover A (2005. szeptember 1.). „Early work on the ubiquitin proteasome system, an interview with Aaron Ciechanover. Interview by CDD”. Cell Death and Differentiation 12 (9), 1167–77. o. DOI:10.1038/sj.cdd.4401691. PMID 16094393.
- ↑ Wilk S, Orlowski M (1980. november 1.). „Cation-sensitive neutral endopeptidase: isolation and specificity of the bovine pituitary enzyme”. Journal of Neurochemistry 35 (5), 1172–82. o. DOI:10.1111/j.1471-4159.1980.tb07873.x. PMID 6778972.
- ↑ Arrigo AP, Tanaka, K, Goldberg F, Welch WJ (1988). „Identity of 19S prosome particle with the large multifunctional protease complex of mammalian cells”. Nature 331 (6152), 192–94. o. DOI:10.1038/331192a0. PMID 3277060. Tanaka K, Waxman L, Goldberg AL (1983. június 1.). „ATP serves two distinct roles in protein degradation in reticulocytes, one requiring and one independent of ubiquitin”. The Journal of Cell Biology 96 (6), 1580–5. o. DOI:10.1083/jcb.96.6.1580. PMID 6304111.
- ↑ Hough R, Pratt G, Rechsteiner M (1987. június 1.). „Purification of two high molecular weight proteases from rabbit reticulocyte lysate”. The Journal of Biological Chemistry 262 (17), 8303–13. o. DOI:10.1016/S0021-9258(18)47564-3. PMID 3298229.
- ↑ Hershko A (2005. szeptember 1.). „Early work on the ubiquitin proteasome system, an interview with Avram Hershko. Interview by CDD”. Cell Death and Differentiation 12 (9), 1158–61. o. DOI:10.1038/sj.cdd.4401709. PMID 16094391.
- ↑ Kopp F, Steiner R, Dahlmann B, Kuehn L, Reinauer H (1986. augusztus 1.). „Size and shape of the multicatalytic proteinase from rat skeletal muscle”. Biochimica et Biophysica Acta (BBA) - Protein Structure and Molecular Enzymology 872 (3), 253–60. o. DOI:10.1016/0167-4838(86)90278-5. PMID 3524688.
- ↑ Löwe J, Stock D, Jap B, Zwickl P, Baumeister W, Huber R (1995. április 1.). „Crystal structure of the 20S proteasome from the archaeon T. acidophilum at 3.4 A resolution”. Science 268 (5210), 533–9. o. DOI:10.1126/science.7725097. PMID 7725097.
- ↑ a b c d e f g h i j k l Dong Y, Zhang S, Wu Z, Li X, Wang WL, Zhu Y, Stoilova-McPhie S, Lu Y, Finley D, Mao Y (2018. november 1.). „Cryo-EM structures and dynamics of substrate-engaged human 26S proteasome”. Nature 565 (7737), 49–55. o. DOI:10.1038/s41586-018-0736-4. PMID 30479383.
- ↑ a b c d Wang J, Maldonado MA (2006. augusztus 1.). „The ubiquitin-proteasome system and its role in inflammatory and autoimmune diseases”. Cellular & Molecular Immunology 3 (4), 255–61. o. PMID 16978533.
- ↑ a b c (2011. január 7.) „Proteasome activators”. Molecular Cell 41 (1), 8–19. o. DOI:10.1016/j.molcel.2010.12.020. PMID 21211719.
- ↑ a b c Smith DM, Chang SC, Park S, Finley D, Cheng Y, Goldberg AL (2007. szeptember 1.). „Docking of the proteasomal ATPases' carboxyl termini in the 20S proteasome's alpha ring opens the gate for substrate entry”. Molecular Cell 27 (5), 731–44. o. DOI:10.1016/j.molcel.2007.06.033. PMID 17803938.
- ↑ MEROPS Family T1. EMBL-EBI. (Hozzáférés: 2019. február 16.)
- ↑ Wilk S, Orlowski M (1983. március 1.). „Evidence that pituitary cation-sensitive neutral endopeptidase is a multicatalytic protease complex”. Journal of Neurochemistry 40 (3), 842–9. o. DOI:10.1111/j.1471-4159.1983.tb08056.x. PMID 6338156.
- ↑ a b Nandi D, Tahiliani P, Kumar A, Chandu D (2006. március 1.). „The ubiquitin-proteasome system”. Journal of Biosciences 31 (1), 137–55. o. DOI:10.1007/BF02705243. PMID 16595883.
- ↑ a b Heinemeyer W, Fischer M, Krimmer T, Stachon U, Wolf DH (1997. október 1.). „The active sites of the eukaryotic 20 S proteasome and their involvement in subunit precursor processing”. The Journal of Biological Chemistry 272 (40), 25200–9. o. DOI:10.1074/jbc.272.40.25200. PMID 9312134.
- ↑ a b Padmanabhan A, Vuong SA, Hochstrasser M (2016. március 1.). „Assembly of an Evolutionarily Conserved Alternative Proteasome Isoform in Human Cells”. Cell Reports 14 (12), 2962–74. o. DOI:10.1016/j.celrep.2016.02.068. PMID 26997268.
- ↑ Velichutina I, Connerly PL, Arendt CS, Li X, Hochstrasser M (2004. február 1.). „Plasticity in eucaryotic 20S proteasome ring assembly revealed by a subunit deletion in yeast”. The EMBO Journal 23 (3), 500–10. o. DOI:10.1038/sj.emboj.7600059. PMID 14739934.
- ↑ Kusmierczyk AR, Kunjappu MJ, Funakoshi M, Hochstrasser M (2008. március 1.). „A multimeric assembly factor controls the formation of alternative 20S proteasomes”. Nature Structural & Molecular Biology 15 (3), 237–44. o. DOI:10.1038/nsmb.1389. PMID 18278055.
- ↑ Zwickl P, Ng D, Woo KM, Klenk HP, Goldberg AL (1999. szeptember 1.). „An archaebacterial ATPase, homologous to ATPases in the eukaryotic 26 S proteasome, activates protein breakdown by 20 S proteasomes”. The Journal of Biological Chemistry 274 (37), 26008–14. o. DOI:10.1074/jbc.274.37.26008. PMID 10473546.
- ↑ a b c d Smith DM, Kafri G, Cheng Y, Ng D, Walz T, Goldberg AL (2005. december 1.). „ATP binding to PAN or the 26S ATPases causes association with the 20S proteasome, gate opening, and translocation of unfolded proteins”. Molecular Cell 20 (5), 687–98. o. DOI:10.1016/j.molcel.2005.10.019. PMID 16337593.
- ↑ a b c d e Liu CW, Li X, Thompson D, Wooding K, Chang TL, Tang Z, Yu H, Thomas PJ, DeMartino GN (2006. október 1.). „ATP binding and ATP hydrolysis play distinct roles in the function of 26S proteasome”. Molecular Cell 24 (1), 39–50. o. DOI:10.1016/j.molcel.2006.08.025. PMID 17018291.
- ↑ Lam YA, Lawson TG, Velayutham M, Zweier JL, Pickart CM (2002. április 1.). „A proteasomal ATPase subunit recognizes the polyubiquitin degradation signal”. Nature 416 (6882), 763–7. o. DOI:10.1038/416763a. PMID 11961560.
- ↑ a b Beck F, Unverdorben P, Bohn S, Schweitzer A, Pfeifer G, Sakata E, Nickell S, Plitzko JM, Villa E, Baumeister W, Förster F (2012. szeptember 1.). „Near-atomic resolution structural model of the yeast 26S proteasome”. Proceedings of the National Academy of Sciences of the United States of America 109 (37), 14870–5. o. DOI:10.1073/pnas.1213333109. PMID 22927375.
- ↑ a b Lander GC, Estrin E, Matyskiela ME, Bashore C, Nogales E, Martin A (2012. február 1.). „Complete subunit architecture of the proteasome regulatory particle”. Nature 482 (7384), 186–91. o. DOI:10.1038/nature10774. PMID 22237024.
- ↑ Lasker K, Förster F, Bohn S, Walzthoeni T, Villa E, Unverdorben P, Beck F, Aebersold R, Sali A, Baumeister W (2012. január 1.). „Molecular architecture of the 26S proteasome holocomplex determined by an integrative approach”. Proceedings of the National Academy of Sciences of the United States of America 109 (5), 1380–7. o. DOI:10.1073/pnas.1120559109. PMID 22307589.
- ↑ a b c Chen S, Wu J, Lu Y, Ma YB, Lee BH, Yu Z, Ouyang Q, Finley DJ, Kirschner MW, Mao Y (2016. november 1.). „Structural basis for dynamic regulation of the human 26S proteasome”. Proceedings of the National Academy of Sciences of the United States of America 113 (46), 12991–12996. o. DOI:10.1073/pnas.1614614113. PMID 27791164.
- ↑ Huang X, Luan B, Wu J, Shi Y (2016. szeptember 1.). „An atomic structure of the human 26S proteasome”. Nature Structural & Molecular Biology 23 (9), 778–785. o. DOI:10.1038/nsmb.3273. PMID 27428775.
- ↑ Schweitzer A, Aufderheide A, Rudack T, Beck F, Pfeifer G, Plitzko JM, Sakata E, Schulten K, Förster F, Baumeister W (2016. július 1.). „Structure of the human 26S proteasome at a resolution of 3.9 Å”. Proceedings of the National Academy of Sciences of the United States of America 113 (28), 7816–7821. o. DOI:10.1073/pnas.1614614113. PMID 27791164.
- ↑ a b c d Zhu Y, Wang WL, Yu D, Ouyang Q, Lu Y, Mao Y (2018. április 1.). „Structural mechanism for nucleotide-driven remodeling of the AAA-ATPase unfoldase in the activated human 26S proteasome”. Nature Communications 9 (1), 1360. o. DOI:10.1038/s41467-018-03785-w. PMID 29636472.
- ↑ a b c Unverdorben P, Beck F, Śledź P, Schweitzer A, Pfeifer G, Plitzko JM, Baumeister W, Förster F (2014. április 1.). „Deep classification of a large cryo-EM dataset defines the conformational landscape of the 26S proteasome”. Proceedings of the National Academy of Sciences of the United States of America 111 (15), 5544–9. o. DOI:10.1073/pnas.1403409111. PMID 24706844.
- ↑ Śledź P, Unverdorben P, Beck F, Pfeifer G, Schweitzer A, Förster F, Baumeister W (2013. április 1.). „Structure of the 26S proteasome with ATP-γS bound provides insights into the mechanism of nucleotide-dependent substrate translocation”. Proceedings of the National Academy of Sciences of the United States of America 110 (18), 7264–7269. o. DOI:10.1073/pnas.1305782110. PMID 23589842.
- ↑ Matyskiela ME, Lander GC, Martin A (2013. július 1.). „Conformational switching of the 26S proteasome enables substrate degradation”. Nature Structural & Molecular Biology 20 (7), 781–788. o. DOI:10.1038/nsmb.2616. PMID 23770819.
- ↑ Lu Y, Wu J, Dong Y, Chen S, Sun S, Ma YB, Ouyang Q, Finley D, Kirschner MW, Mao Y (2017. július 1.). „Conformational Landscape of the p28-Bound Human Proteasome Regulatory Particle”. Molecular Cell 67 (2), 322–333.e6. o. DOI:10.1016/j.molcel.2017.06.007. PMID 28689658.
- ↑ Köhler A, Cascio P, Leggett DS, Woo KM, Goldberg AL, Finley D (2001. június 1.). „The axial channel of the proteasome core particle is gated by the Rpt2 ATPase and controls both substrate entry and product release”. Molecular Cell 7 (6), 1143–52. o. DOI:10.1016/S1097-2765(01)00274-X. PMID 11430818.
- ↑ Förster A, Masters EI, Whitby FG, Robinson H, Hill CP (2005. május 1.). „The 1.9 A structure of a proteasome-11S activator complex and implications for proteasome-PAN/PA700 interactions”. Molecular Cell 18 (5), 589–99. o. DOI:10.1016/j.molcel.2005.04.016. PMID 15916965.
- ↑ Witt S, Kwon YD, Sharon M, Felderer K, Beuttler M, Robinson CV, Baumeister W, Jap BK (2006. július 1.). „Proteasome assembly triggers a switch required for active-site maturation”. Structure 14 (7), 1179–88. o. DOI:10.1016/j.str.2006.05.019. PMID 16843899.
- ↑ Krüger E, Kloetzel PM, Enenkel C (2001). „20S proteasome biogenesis”. Biochimie 83 (3–4), 289–93. o. DOI:10.1016/S0300-9084(01)01241-X. PMID 11295488.
- ↑ Murata S, Yashiroda H, Tanaka K (2009. február 1.). „Molecular mechanisms of proteasome assembly”. Nature Reviews Molecular Cell Biology 10 (2), 104–115. o. DOI:10.1038/nrm2630. PMID 19165213.
- ↑ Sakata E, Stengel F, Fukunaga K, Zhou M, Saeki Y, Förster F, Baumeister W, Tanaka K, Robinson CV (2011. június 1.). „The catalytic activity of Ubp6 enhances maturation of the proteasomal regulatory particle”. Molecular Cell 42 (5), 637–649. o. DOI:10.1016/j.molcel.2011.04.021. PMID 21658604.
- ↑ Fukunaga K, Kudo T, Toh-e A, Tanaka K, Saeki Y (2010. június 1.). „Dissection of the assembly pathway of the proteasome lid in Saccharomyces cerevisiae”. Biochemical and Biophysical Research Communications 396 (4), 1048–1053. o. DOI:10.1016/j.bbrc.2010.05.061. PMID 20471955.
- ↑ Haas AL, Warms JV, Hershko A, Rose IA (1982. március 1.). „Ubiquitin-activating enzyme. Mechanism and role in protein-ubiquitin conjugation”. The Journal of Biological Chemistry 257 (5), 2543–8. o. DOI:10.1016/S0021-9258(18)34958-5. PMID 6277905.
- ↑ Thrower JS, Hoffman L, Rechsteiner M, Pickart CM (2000. január 1.). „Recognition of the polyubiquitin proteolytic signal”. The EMBO Journal 19 (1), 94–102. o. DOI:10.1093/emboj/19.1.94. PMID 10619848.
- ↑ Risseeuw EP, Daskalchuk TE, Banks TW, Liu E, Cotelesage J, Hellmann H, Estelle M, Somers DE, Crosby WL (2003. június 1.). „Protein interaction analysis of SCF ubiquitin E3 ligase subunits from Arabidopsis”. The Plant Journal 34 (6), 753–67. o. DOI:10.1046/j.1365-313X.2003.01768.x. PMID 12795696.
- ↑ Elsasser S, Finley D (2005. augusztus 1.). „Delivery of ubiquitinated substrates to protein-unfolding machines”. Nature Cell Biology 7 (8), 742–9. o. DOI:10.1038/ncb0805-742. PMID 16056265.
- ↑ Sadanandom A, Bailey M, Ewan R, Lee J, Nelis S (2012. október 1.). „The ubiquitin-proteasome system: central modifier of plant signalling”. The New Phytologist 196 (1), 13–28. o. DOI:10.1111/j.1469-8137.2012.04266.x. PMID 22897362.
- ↑ Sharp PM, Li WH (1987). „Ubiquitin genes as a paradigm of concerted evolution of tandem repeats”. Journal of Molecular Evolution 25 (1), 58–64. o. DOI:10.1007/BF02100041. PMID 3041010.
- ↑ Pickart CM, Fushman D (2004. december 1.). „Polyubiquitin chains: polymeric protein signals”. Current Opinion in Chemical Biology 8 (6), 610–16. o. DOI:10.1016/j.cbpa.2004.09.009. PMID 15556404.
- ↑ Xu P, Duong DM, Seyfried NT, Cheng D, Xie Y, Robert J, Rush J, Hochstrasser M, Finley D, Peng J (2009. április 1.). „Quantitative proteomics reveals the function of unconventional ubiquitin chains in proteasomal degradation”. Cell 137 (1), 133–45. o. DOI:10.1016/j.cell.2009.01.041. PMID 19345192.
- ↑ a b Pickart CM (2000. november 1.). „Ubiquitin in chains”. Trends in Biochemical Sciences 25 (11), 544–8. o. DOI:10.1016/S0968-0004(00)01681-9. PMID 11084366.
- ↑ a b Zhang S, Zou S, Yin D, Zhao L, Finley D, Wu Z, Mao Y (2022. április 1.). „USP14-regulated allostery of the human proteasome by time-resolved cryo-EM”. Nature 605 (7910), 567–574. o. DOI:10.1038/s41586-022-04671-8. PMID 35477760.
- ↑ Zhu Q, Wani G, Wang QE, El-mahdy M, Snapka RM, Wani AA (2005. július 1.). „Deubiquitination by proteasome is coordinated with substrate translocation for proteolysis in vivo”. Experimental Cell Research 307 (2), 436–51. o. DOI:10.1016/j.yexcr.2005.03.031. PMID 15950624.
- ↑ Wenzel T, Baumeister W (1995. március 1.). „Conformational constraints in protein degradation by the 20S proteasome”. Nature Structural Biology 2 (3), 199–204. o. DOI:10.1038/nsb0395-199. PMID 7773788.
- ↑ Inobe T, Fishbain S, Prakash S, Matouschek A (2011. március 1.). „Defining the geometry of the two-component proteasome degron”. Nature Chemical Biology 7 (3), 161–7. o. DOI:10.1038/nchembio.521. PMID 21278740.
- ↑ van der Lee R, Lang B, Kruse K, Gsponer J, Sánchez de Groot N, Huynen MA, Matouschek A, Fuxreiter M, Babu MM (2014. szeptember 1.). „Intrinsically disordered segments affect protein half-life in the cell and during evolution”. Cell Reports 8 (6), 1832–44. o. DOI:10.1016/j.celrep.2014.07.055. PMID 25220455.
- ↑ Smith DM, Benaroudj N, Goldberg A (2006. október 1.). „Proteasomes and their associated ATPases: a destructive combination”. Journal of Structural Biology 156 (1), 72–83. o. DOI:10.1016/j.jsb.2006.04.012. PMID 16919475.
- ↑ Hoyt MA, Zich J, Takeuchi J, Zhang M, Govaerts C, Coffino P (2006. április 1.). „Glycine-alanine repeats impair proper substrate unfolding by the proteasome”. The EMBO Journal 25 (8), 1720–9. o. DOI:10.1038/sj.emboj.7601058. PMID 16601692.
- ↑ Zhang M, Coffino P (2004. március 1.). „Repeat sequence of Epstein–Barr virus-encoded nuclear antigen 1 protein interrupts proteasome substrate processing”. The Journal of Biological Chemistry 279 (10), 8635–41. o. DOI:10.1074/jbc.M310449200. PMID 14688254.
- ↑ Seemüller E, Lupas A, Stock D, Löwe J, Huber R, Baumeister W (1995. április 1.). „Proteasome from Thermoplasma acidophilum: a threonine protease”. Science 268 (5210), 579–82. o. DOI:10.1126/science.7725107. PMID 7725107.
- ↑ Coux O, Tanaka K, Goldberg AL (1996. április 25.). „Structure and functions of the 20S and 26S proteasomes”. Annual Review of Biochemistry 65, 801–47. o. DOI:10.1146/annurev.bi.65.070196.004101. PMID 8811196.
- ↑ Groll M, Ditzel L, Löwe J, Stock D, Bochtler M, Bartunik HD, Huber R (1997. április 1.). „Structure of 20S proteasome from yeast at 2.4 A resolution”. Nature 386 (6624), 463–71. o. DOI:10.1038/386463a0. PMID 9087403.
- ↑ Dick TP, Nussbaum AK, Deeg M, Heinemeyer W, Groll M, Schirle M, Keilholz W, Stevanović S, Wolf DH, Huber R, Rammensee HG, Schild H (1998. október 1.). „Contribution of proteasomal beta-subunits to the cleavage of peptide substrates analyzed with yeast mutants”. The Journal of Biological Chemistry 273 (40), 25637–46. o. DOI:10.1074/jbc.273.40.25637. PMID 9748229.
- ↑ Voges D, Zwickl P, Baumeister W (1999). „The 26S proteasome: a molecular machine designed for controlled proteolysis”. Annual Review of Biochemistry 68 (1), 1015–68. o. DOI:10.1146/annurev.biochem.68.1.1015. PMID 10872471.
- ↑ a b Rape M, Jentsch S (2002. május 1.). „Taking a bite: proteasomal protein processing”. Nature Cell Biology 4 (5), E113–6. o. DOI:10.1038/ncb0502-e113. PMID 11988749.
- ↑ Rape M, Jentsch S (2004. november 1.). „Productive RUPture: activation of transcription factors by proteasomal processing”. Biochimica et Biophysica Acta (BBA) - Molecular Cell Research 1695 (1–3), 209–13. o. DOI:10.1016/j.bbamcr.2004.09.022. PMID 15571816.
- ↑ Asher G, Reuven N, Shaul Y (2006. augusztus 1.). „20S proteasomes and protein degradation "by default"”. BioEssays 28 (8), 844–9. o. DOI:10.1002/bies.20447. PMID 16927316.
- ↑ Zhang M, Pickart CM, Coffino P (2003. április 1.). „Determinants of proteasome recognition of ornithine decarboxylase, a ubiquitin-independent substrate”. The EMBO Journal 22 (7), 1488–96. o. DOI:10.1093/emboj/cdg158. PMID 12660156.
- ↑ Asher G, Shaul Y (2005. augusztus 1.). „p53 proteasomal degradation: poly-ubiquitination is not the whole story”. Cell Cycle 4 (8), 1015–8. o. DOI:10.4161/cc.4.8.1900. PMID 16082197.
- ↑ a b Shringarpure R, Grune T, Mehlhase J, Davies KJ (2003. január 1.). „Ubiquitin conjugation is not required for the degradation of oxidized proteins by proteasome”. The Journal of Biological Chemistry 278 (1), 311–8. o. DOI:10.1074/jbc.M206279200. PMID 12401807.
- ↑ a b c Gille C, Goede A, Schlöetelburg C, Preissner R, Kloetzel PM, Göbel UB, Frömmel C (2003. március 1.). „A comprehensive view on proteasomal sequences: implications for the evolution of the proteasome”. Journal of Molecular Biology 326 (5), 1437–48. o. DOI:10.1016/S0022-2836(02)01470-5. PMID 12595256.
- ↑ Bochtler M, Ditzel L, Groll M, Hartmann C, Huber R (1999). „The proteasome”. Annual Review of Biophysics and Biomolecular Structure 28 (1), 295–317. o. DOI:10.1146/annurev.biophys.28.1.295. PMID 10410804.
- ↑ Chesnel F, Bazile F, Pascal A, Kubiak JZ (2006. augusztus 1.). „Cyclin B dissociation from CDK1 precedes its degradation upon MPF inactivation in mitotic extracts of Xenopus laevis embryos”. Cell Cycle 5 (15), 1687–98. o. DOI:10.4161/cc.5.15.3123. PMID 16921258.
- ↑ Brito DA, Rieder CL (2006. június 1.). „Mitotic checkpoint slippage in humans occurs via cyclin B destruction in the presence of an active checkpoint”. Current Biology 16 (12), 1194–200. o. DOI:10.1016/j.cub.2006.04.043. PMID 16782009.
- ↑ Havens CG, Ho A, Yoshioka N, Dowdy SF (2006. június 1.). „Regulation of late G1/S phase transition and APC Cdh1 by reactive oxygen species”. Molecular and Cellular Biology 26 (12), 4701–11. o. DOI:10.1128/MCB.00303-06. PMID 16738333.
- ↑ Bashir T, Dorrello NV, Amador V, Guardavaccaro D, Pagano M (2004. március 1.). „Control of the SCF(Skp2-Cks1) ubiquitin ligase by the APC/C(Cdh1) ubiquitin ligase”. Nature 428 (6979), 190–3. o. DOI:10.1038/nature02330. PMID 15014502.
- ↑ Higashitsuji H, Liu Y, Mayer RJ, Fujita J (2005. október 1.). „The oncoprotein gankyrin negatively regulates both p53 and RB by enhancing proteasomal degradation”. Cell Cycle 4 (10), 1335–7. o. DOI:10.4161/cc.4.10.2107. PMID 16177571.
- ↑ (2020-08-07020) „The proteasome controls ESCRT-III–mediated cell division in an archaeon”. Science 369 (6504), eaaz2532. o. DOI:10.1126/science.aaz2532. PMID 32764038.
- ↑ Dharmasiri S, Estelle M (2002). „The role of regulated protein degradation in auxin response”. Plant Molecular Biology 49 (3–4), 401–9. o. DOI:10.1023/A:1015203013208. PMID 12036263.
- ↑ Weijers D, Benkova E, Jäger KE, Schlereth A, Hamann T, Kientz M, Wilmoth JC, Reed JW, Jürgens G (2005. május 1.). „Developmental specificity of auxin response by pairs of ARF and Aux/IAA transcriptional regulators”. The EMBO Journal 24 (10), 1874–85. o. DOI:10.1038/sj.emboj.7600659. PMID 15889151.
- ↑ Haas AL, Baboshina O, Williams B, Schwartz LM (1995. április 1.). „Coordinated induction of the ubiquitin conjugation pathway accompanies the developmentally programmed death of insect skeletal muscle”. The Journal of Biological Chemistry 270 (16), 9407–12. o. DOI:10.1074/jbc.270.16.9407. PMID 7721865.
- ↑ Schwartz LM, Myer A, Kosz L, Engelstein M, Maier C (1990. október 1.). „Activation of polyubiquitin gene expression during developmentally programmed cell death”. Neuron 5 (4), 411–9. o. DOI:10.1016/0896-6273(90)90080-Y. PMID 2169771.
- ↑ Löw P, Bussell K, Dawson SP, Billett MA, Mayer RJ, Reynolds SE (1997. január 1.). „Expression of a 26S proteasome ATPase subunit, MS73, in muscles that undergo developmentally programmed cell death, and its control by ecdysteroid hormones in the insect Manduca sexta”. FEBS Letters 400 (3), 345–9. o. DOI:10.1016/S0014-5793(96)01413-5. PMID 9009228.
- ↑ Pitzer F, Dantes A, Fuchs T, Baumeister W, Amsterdam A (1996. szeptember 1.). „Removal of proteasomes from the nucleus and their accumulation in apoptotic blebs during programmed cell death”. FEBS Letters 394 (1), 47–50. o. DOI:10.1016/0014-5793(96)00920-9. PMID 8925925.
- ↑ a b Adams J, Palombella VJ, Sausville EA, Johnson J, Destree A, Lazarus DD, Maas J, Pien CS, Prakash S, Elliott PJ (1999. június 1.). „Proteasome inhibitors: a novel class of potent and effective antitumor agents”. Cancer Research 59 (11), 2615–22. o. PMID 10363983.
- ↑ Orlowski RZ (1999. április 1.). „The role of the ubiquitin-proteasome pathway in apoptosis”. Cell Death and Differentiation 6 (4), 303–13. o. DOI:10.1038/sj.cdd.4400505. PMID 10381632.
- ↑ Garrido C, Brunet M, Didelot C, Zermati Y, Schmitt E, Kroemer G (2006. november 1.). „Heat shock proteins 27 and 70: anti-apoptotic proteins with tumorigenic properties”. Cell Cycle 5 (22), 2592–2601. o. DOI:10.4161/cc.5.22.3448. PMID 17106261.
- ↑ Park SH, Bolender N, Eisele F, Kostova Z, Takeuchi J, Coffino P, Wolf DH (2007. január 1.). „The cytoplasmic Hsp70 chaperone machinery subjects misfolded and endoplasmic reticulum import-incompetent proteins to degradation via the ubiquitin-proteasome system”. Molecular Biology of the Cell 18 (1), 153–65. o. DOI:10.1091/mbc.E06-04-0338. PMID 17065559.
- ↑ Dai Q, Qian SB, Li HH, McDonough H, Borchers C, Huang D, Takayama S, Younger JM, Ren HY, Cyr DM, Patterson C (2005. november 1.). „Regulation of the cytoplasmic quality control protein degradation pathway by BAG2”. The Journal of Biological Chemistry 280 (46), 38673–81. o. DOI:10.1074/jbc.M507986200. PMID 16169850.
- ↑ Bader N, Grune T (2006). „Protein oxidation and proteolysis”. Biological Chemistry 387 (10–11), 1351–5. o. DOI:10.1515/BC.2006.169. PMID 17081106.
- ↑ Davies KJ (2003). „Degradation of oxidized proteins by the 20S proteasome”. Biochimie 83 (3–4), 301–10. o. DOI:10.1016/S0300-9084(01)01250-0. PMID 11295490.
- ↑ a b Lehman NL (2009. szeptember 1.). „The ubiquitin proteasome system in neuropathology”. Acta Neuropathologica 118 (3), 329–47. o. DOI:10.1007/s00401-009-0560-x. PMID 19597829.
- ↑ McNaught KS, Jackson T, JnoBaptiste R, Kapustin A, Olanow CW (2006. május 1.). „Proteasomal dysfunction in sporadic Parkinson's disease”. Neurology 66 (10 Suppl 4), S37–49. o. DOI:10.1212/01.wnl.0000221745.58886.2e. PMID 16717251.
- ↑ Sharma N, Brandis KA, Herrera SK, Johnson BE, Vaidya T, Shrestha R, Debburman SK (2006). „alpha-Synuclein budding yeast model: toxicity enhanced by impaired proteasome and oxidative stress”. Journal of Molecular Neuroscience 28 (2), 161–78. o. DOI:10.1385/JMN:28:2:161. PMID 16679556.
- ↑ Murata S, Sasaki K, Kishimoto T, Niwa S, Hayashi H, Takahama Y, Tanaka K (2007. június 1.). „Regulation of CD8+ T cell development by thymus-specific proteasomes”. Science 316 (5829), 1349–53. o. DOI:10.1126/science.1141915. PMID 17540904.
- ↑ Cascio P, Hilton C, Kisselev AF, Rock KL, Goldberg AL (2001. május 1.). „26S proteasomes and immunoproteasomes produce mainly N-extended versions of an antigenic peptide”. The EMBO Journal 20 (10), 2357–66. o. DOI:10.1093/emboj/20.10.2357. PMID 11350924.
- ↑ Mallery DL, McEwan WA, Bidgood SR, Towers GJ, Johnson CM, James LC (2010. november 1.). „Antibodies mediate intracellular immunity through tripartite motif-containing 21 (TRIM21)”. Proceedings of the National Academy of Sciences of the United States of America 107 (46), 19985–19990. o. DOI:10.1073/pnas.1014074107. PMID 21045130.
- ↑ Fenteany G, Standaert RF, Lane WS, Choi S, Corey EJ, Schreiber SL (1995. május 1.). „Inhibition of proteasome activities and subunit-specific amino-terminal threonine modification by lactacystin”. Science 268 (5211), 726–31. o. DOI:10.1126/science.7732382. PMID 7732382.
- ↑ "United States Food and Drug Administration press release". Sajtóközlemény. Elérés: 2006-12-29.
- ↑ Fisher RI, Bernstein SH, Kahl BS, Djulbegovic B, Robertson MJ, de Vos S, Epner E, Krishnan A, Leonard JP, Lonial S, Stadtmauer EA, O'Connor OA, Shi H, Boral AL, Goy A (2006. október 1.). „Multicenter phase II study of bortezomib in patients with relapsed or refractory mantle cell lymphoma”. Journal of Clinical Oncology 24 (30), 4867–74. o. DOI:10.1200/JCO.2006.07.9665. PMID 17001068.
- ↑ Jakob C, Egerer K, Liebisch P, Türkmen S, Zavrski I, Kuckelkorn U, Heider U, Kaiser M, Fleissner C, Sterz J, Kleeberg L, Feist E, Burmester GR, Kloetzel PM, Sezer O (2007. március 1.). „Circulating proteasome levels are an independent prognostic factor for survival in multiple myeloma”. Blood 109 (5), 2100–5. o. DOI:10.1182/blood-2006-04-016360. PMID 17095627.
- ↑ Shah SA, Potter MW, McDade TP, Ricciardi R, Perugini RA, Elliott PJ, Adams J, Callery MP (2001). „26S proteasome inhibition induces apoptosis and limits growth of human pancreatic cancer”. Journal of Cellular Biochemistry 82 (1), 110–22. o. DOI:10.1002/jcb.1150. PMID 11400168.
- ↑ Nawrocki ST, Sweeney-Gotsch B, Takamori R, McConkey DJ (2004. január 1.). „The proteasome inhibitor bortezomib enhances the activity of docetaxel in orthotopic human pancreatic tumor xenografts”. Molecular Cancer Therapeutics 3 (1), 59–70. o. DOI:10.1158/1535-7163.59.3.1. PMID 14749476.
- ↑ Schenkein D (2002. június 1.). „Proteasome inhibitors in the treatment of B-cell malignancies”. Clinical Lymphoma 3 (1), 49–55. o. DOI:10.3816/CLM.2002.n.011. PMID 12141956.
- ↑ O'Connor OA, Wright J, Moskowitz C, Muzzy J, MacGregor-Cortelli B, Stubblefield M, Straus D, Portlock C, Hamlin P, Choi E, Dumetrescu O, Esseltine D, Trehu E, Adams J, Schenkein D, Zelenetz AD (2005. február 1.). „Phase II clinical experience with the novel proteasome inhibitor bortezomib in patients with indolent non-Hodgkin's lymphoma and mantle cell lymphoma”. Journal of Clinical Oncology 23 (4), 676–84. o. DOI:10.1200/JCO.2005.02.050. PMID 15613699.
- ↑ Messinger YH, Gaynon PS, Sposto R, van der Giessen J, Eckroth E, Malvar J, Bostrom BC (2012. július 1.). „Bortezomib with chemotherapy is highly active in advanced B-precursor acute lymphoblastic leukemia: Therapeutic Advances in Childhood Leukemia & Lymphoma (TACL) Study”. Blood 120 (2), 285–90. o. DOI:10.1182/blood-2012-04-418640. PMID 22653976.
- ↑ Lambrou GI, Papadimitriou L, Chrousos GP, Vlahopoulos SA (2012. április 1.). „Glucocorticoid and proteasome inhibitor impact on the leukemic lymphoblast: multiple, diverse signals converging on a few key downstream regulators”. Molecular and Cellular Endocrinology 351 (2), 142–51. o. DOI:10.1016/j.mce.2012.01.003. PMID 22273806.
- ↑ Schmidtke G, Holzhütter HG, Bogyo M, Kairies N, Groll M, de Giuli R, Emch S, Groettrup M (1999. december 1.). „How an inhibitor of the HIV-I protease modulates proteasome activity”. The Journal of Biological Chemistry 274 (50), 35734–40. o. DOI:10.1074/jbc.274.50.35734. PMID 10585454.
- ↑ Laurent N, de Boüard S, Guillamo JS, Christov C, Zini R, Jouault H, Andre P, Lotteau V, Peschanski M (2004. február 1.). „Effects of the proteasome inhibitor ritonavir on glioma growth in vitro and in vivo”. Molecular Cancer Therapeutics 3 (2), 129–36. o. DOI:10.1158/1535-7163.129.3.2. PMID 14985453.
- ↑ Zollner TM, Podda M, Pien C, Elliott PJ, Kaufmann R, Boehncke WH (2002. március 1.). „Proteasome inhibition reduces superantigen-mediated T cell activation and the severity of psoriasis in a SCID-hu model”. The Journal of Clinical Investigation 109 (5), 671–9. o. DOI:10.1172/JCI12736. PMID 11877475.
- ↑ Elliott PJ, Pien CS, McCormack TA, Chapman ID, Adams J (1999. augusztus 1.). „Proteasome inhibition: A novel mechanism to combat asthma”. The Journal of Allergy and Clinical Immunology 104 (2 Pt 1), 294–300. o. DOI:10.1016/S0091-6749(99)70369-6. PMID 10452747.
- ↑ Verdoes M, Florea BI, Menendez-Benito V, Maynard CJ, Witte MD, van der Linden WA, van den Nieuwendijk AM, Hofmann T, Berkers CR, van Leeuwen FW, Groothuis TA, Leeuwenburgh MA, Ovaa H, Neefjes JJ, Filippov DV, van der Marel GA, Dantuma NP, Overkleeft HS (2006. november 1.). „A fluorescent broad-spectrum proteasome inhibitor for labeling proteasomes in vitro and in vivo”. Chemistry & Biology 13 (11), 1217–26. o. DOI:10.1016/j.chembiol.2006.09.013. PMID 17114003.
- ↑ Kleiger G, Mayor T (2014. június 1.). „Perilous journey: a tour of the ubiquitin-proteasome system”. Trends in Cell Biology 24 (6), 352–9. o. DOI:10.1016/j.tcb.2013.12.003. PMID 24457024.
- ↑ Goldberg AL, Stein R, Adams J (1995. augusztus 1.). „New insights into proteasome function: from archaebacteria to drug development”. Chemistry & Biology 2 (8), 503–8. o. DOI:10.1016/1074-5521(95)90182-5. PMID 9383453.
- ↑ Sulistio YA, Heese K (2015. január 1.). „The Ubiquitin–Proteasome System and Molecular Chaperone Deregulation in Alzheimer's Disease”. Molecular Neurobiology 53 (2), 905–31. o. DOI:10.1007/s12035-014-9063-4. PMID 25561438.
- ↑ Ortega Z, Lucas JJ (2014. április 25.). „Ubiquitin-proteasome system involvement in Huntington's disease”. Frontiers in Molecular Neuroscience 7, 77. o. DOI:10.3389/fnmol.2014.00077. PMID 25324717.
- ↑ Sandri M, Robbins J (2014. június 1.). „Proteotoxicity: an underappreciated pathology in cardiac disease”. Journal of Molecular and Cellular Cardiology 71, 3–10. o. DOI:10.1016/j.yjmcc.2013.12.015. PMID 24380730.
- ↑ Drews O, Taegtmeyer H (2014. december 1.). „Targeting the ubiquitin–proteasome system in heart disease: the basis for new therapeutic strategies”. Antioxidants & Redox Signaling 21 (17), 2322–43. o. DOI:10.1089/ars.2013.5823. PMID 25133688.
- ↑ Wang ZV, Hill JA (2015. február 1.). „Protein quality control and metabolism: bidirectional control in the heart”. Cell Metabolism 21 (2), 215–26. o. DOI:10.1016/j.cmet.2015.01.016. PMID 25651176.
- ↑ a b Karin M, Delhase M (2000. február 1.). „The I kappa B kinase (IKK) and NF-kappa B: key elements of proinflammatory signalling”. Seminars in Immunology 12 (1), 85–98. o. DOI:10.1006/smim.2000.0210. PMID 10723801.
- ↑ Ermolaeva MA, Dakhovnik A, Schumacher B (2015. január 1.). „Quality control mechanisms in cellular and systemic DNA damage responses”. Ageing Research Reviews 23 (Pt A), 3–11. o. DOI:10.1016/j.arr.2014.12.009. PMID 25560147.
- ↑ Checler F, da Costa CA, Ancolio K, Chevallier N, Lopez-Perez E, Marambaud P (2000. július 1.). „Role of the proteasome in Alzheimer's disease”. Biochimica et Biophysica Acta (BBA) – Molecular Basis of Disease 1502 (1), 133–8. o. DOI:10.1016/s0925-4439(00)00039-9. PMID 10899438.
- ↑ a b Chung KK, Dawson VL, Dawson TM (2001. november 1.). „The role of the ubiquitin-proteasomal pathway in Parkinson's disease and other neurodegenerative disorders”. Trends in Neurosciences 24 (11 Suppl), S7–14. o. DOI:10.1016/s0166-2236(00)01998-6. PMID 11881748.
- ↑ a b Ikeda K, Akiyama H, Arai T, Ueno H, Tsuchiya K, Kosaka K (2002. július 1.). „Morphometrical reappraisal of motor neuron system of Pick's disease and amyotrophic lateral sclerosis with dementia”. Acta Neuropathologica 104 (1), 21–8. o. DOI:10.1007/s00401-001-0513-5. PMID 12070660.
- ↑ Manaka H, Kato T, Kurita K, Katagiri T, Shikama Y, Kujirai K, Kawanami T, Suzuki Y, Nihei K, Sasaki H (1992. május 1.). „Marked increase in cerebrospinal fluid ubiquitin in Creutzfeldt–Jakob disease”. Neuroscience Letters 139 (1), 47–9. o. DOI:10.1016/0304-3940(92)90854-z. PMID 1328965.
- ↑ Mathews KD, Moore SA (2003. január 1.). „Limb-girdle muscular dystrophy”. Current Neurology and Neuroscience Reports 3 (1), 78–85. o. DOI:10.1007/s11910-003-0042-9. PMID 12507416.
- ↑ Mayer RJ (2003. március 1.). „From neurodegeneration to neurohomeostasis: the role of ubiquitin”. Drug News & Perspectives 16 (2), 103–8. o. DOI:10.1358/dnp.2003.16.2.829327. PMID 12792671.
- ↑ Calise J, Powell SR (2013. február 1.). „The ubiquitin proteasome system and myocardial ischemia”. American Journal of Physiology. Heart and Circulatory Physiology 304 (3), H337–49. o. DOI:10.1152/ajpheart.00604.2012. PMID 23220331.
- ↑ Predmore JM, Wang P, Davis F, Bartolone S, Westfall MV, Dyke DB, Pagani F, Powell SR, Day SM (2010. március 1.). „Ubiquitin proteasome dysfunction in human hypertrophic and dilated cardiomyopathies”. Circulation 121 (8), 997–1004. o. DOI:10.1161/CIRCULATIONAHA.109.904557. PMID 20159828.
- ↑ Powell SR (2006. július 1.). „The ubiquitin-proteasome system in cardiac physiology and pathology”. American Journal of Physiology. Heart and Circulatory Physiology 291 (1), H1–H19. o. DOI:10.1152/ajpheart.00062.2006. PMID 16501026.
- ↑ Adams J (2003. április 1.). „Potential for proteasome inhibition in the treatment of cancer”. Drug Discovery Today 8 (7), 307–15. o. DOI:10.1016/s1359-6446(03)02647-3. PMID 12654543.
- ↑ Ben-Neriah Y (2002. január 1.). „Regulatory functions of ubiquitination in the immune system”. Nature Immunology 3 (1), 20–26. o. DOI:10.1038/ni0102-20. PMID 11753406.
- ↑ Egerer K, Kuckelkorn U, Rudolph PE, Rückert JC, Dörner T, Burmester GR, Kloetzel PM, Feist E (2002. október 1.). „Circulating proteasomes are markers of cell damage and immunologic activity in autoimmune diseases”. The Journal of Rheumatology 29 (10), 2045–52. o. PMID 12375310.
Fordítás[szerkesztés]
Ez a szócikk részben vagy egészben a Proteasome című angol Wikipédia-szócikk ezen változatának fordításán alapul. Az eredeti cikk szerkesztőit annak laptörténete sorolja fel. Ez a jelzés csupán a megfogalmazás eredetét és a szerzői jogokat jelzi, nem szolgál a cikkben szereplő információk forrásmegjelöléseként.
További információk[szerkesztés]
- Glickman MH, Adir N (2004. január 1.). „The proteasome and the delicate balance between destruction and rescue”. PLOS Biology 2 (1), e13. o. DOI:10.1371/journal.pbio.0020013. PMID 14737189.
- The Yeast 26S Proteasome with list of subunits and pictures
- Ciechanover A (2005. szeptember 1.). „Early work on the ubiquitin proteasome system, an interview with Aaron Ciechanover. Interview by CDD”. Cell Death and Differentiation 12 (9), 1167–77. o. DOI:10.1038/sj.cdd.4401691. PMID 16094393.
- Hershko A (2005. szeptember 1.). „Early work on the ubiquitin proteasome system, an interview with Avram Hershko. Interview by CDD”. Cell Death and Differentiation 12 (9), 1158–61. o. DOI:10.1038/sj.cdd.4401709. PMID 16094391.
- Rose I (2005. szeptember 1.). „Early work on the ubiquitin proteasome system, an interview with Irwin Rose. Interview by CDD”. Cell Death and Differentiation 12 (9), 1162–6. o. DOI:10.1038/sj.cdd.4401700. PMID 16094392.
- Cvek B, Dvorak Z (2007). „Targeting of nuclear factor-kappaB and proteasome by dithiocarbamate complexes with metals”. Current Pharmaceutical Design 13 (30), 3155–67. o. [2012. július 29-i dátummal az eredetiből archiválva]. DOI:10.2174/138161207782110390. PMID 17979756. (Hozzáférés: 2024. február 3.)
- Proteasome subunit nomenclature guide
- 3D proteasome structures in the EM Data Bank (EMDB)
- A proteaszómaműködés fontos lépései Archiválva 2020. november 17-i dátummal a Wayback Machine-ben

