Papillomaviridae
| Papillomavirus | ||||
|---|---|---|---|---|
 Papillómavírusok elektronmikroszkópos képe
| ||||
| Vírusbesorolás | ||||
| ||||
| Nemzetségek | ||||
|
Alphapapillomavirus | ||||
| Hivatkozások | ||||
A Wikifajok tartalmaz Papillomavirus témájú rendszertani információt. A Wikimédia Commons tartalmaz Papillomavirus témájú kategóriát. |
A Papillomaviridae a vírusok egy családja, amelybe kettős szálú DNS-genommal rendelkező, lipidburok nélküli fajok, az ún. papillómavírusok tartoznak. A családba tartozó, majd száz vírusfaj elsősorban emlősöket, valamint madarakat és hüllőket fertőz meg.[1][2][3] A papillómavírus-infekció többnyire nem jár tünetekkel (pl. a legtöbb bétapapillómavírus esetében) vagy kis, jóindulatú bőrdaganatot, szemölcsöt v. papillómát okoz (ilyen a HPV-1, HPV-6 vagy HPV-11). Más típusok esetében, mint a HPV-16 vagy HPV-18 a sejtburjánzás rosszindulatú tumorrá is fajulhat.[4]
Az egyes papillómavírus-típusok gazda- és szövetspecifikusak és csak ritkán adódnak át egyik fajról a másikra.[5] Kizárólag a bőr és a nyálkahártyák bazális sejtrétegében szaporodnak és a fajok ragaszkodnak az adott testtájhoz.[6] Például a HPV-1 a talpat, a HPV-2 pedig a tenyeret támadja meg, ahol a fertőzés hatására szemölcsök nőnek.
A papillómavírusokat a 20. század elején fedezték fel, mikor kimutatták, hogy a szemölcsök átvihetők egyik emberről a másikra és egy mikrobiológiai szűrőkön áthatoló, fertőző ágens okozza őket. A későbbi Nobel-díjas Francis Peyton Rous 1935-ben fedezte fel, hogy a papillómavírusok nyulakban bőrtumort képesek indukálni.
Taxonómia
[szerkesztés]Régebben a papillómavírusokat a poliómavírusokkal együtt a Papovaviridae családba sorolták (papillóma, polióma és vakuolizáló az azóta átsorolt SV40-vírus után), de a DNS-vizsgálatok kimutatták, hogy ez a két csoport nem közeli rokona egymásnak. Ma a Nemzetközi Vírustaxonómiai Bizottság (International Committee on Taxonomy of Viruses, ICTV) a Papilloviridae családon belül 39 nemzetséget és 95 fajt ismer el.[7] Genomjuk szerkezete nagyon hasonló, bár a tényleges nukleotidszekvencia akár 50%-kal is eltérhet. A DNS-szekvenciáik alapján rokonsági fákat lehet szerkeszteni, amelyek alapján megállapítható, hogy ezek a vírusok nem váltanak gazdacsoportot, együtt fejlődnek az emlősökkel és madarakkal, nem rekombinálódnak és alapvető genetikai szerkezetük több mint százmillió éve változatlan. A DNS-szekvenciák alapján állította fel az ICTV az új taxonómiai rendszert is, amely eltér a hagyományos, gazdaszervezetekre alapozott megnevezésektől és amely nem is fajokkal, hanem típusokkal, altípusokkal és variánsokkal dolgozott.[8]
A papillómavírusok ICTV szerinti taxonómiája:
Család: Papillomaviridae
Nemzetség:
- Alphapapillomavirus 14 faj (elnevezésük alfapapillómavírus 1-től 14-ig)
- Betapapillomavirus 6 faj
- Chipapillomavirus 3 faj
- Deltapapillomavirus 6 faj
- Dyodeltapapillomavirus 1 faj
- Dyoepsilonpapillomavirus 1 faj
- Dyoetapapillomavirus 1 faj
- Dyoiotapapillomavirus 2 faj
- Dyokappapapillomavirus 1 faj
- Dyolambdapapillomavirus 1 faj
- Dyomupapillomavirus 1 faj
- Dyonupapillomavirus 1 faj
- Dyoomikronpapillomavirus 1 faj
- Dyopipapillomavirus 1 faj
- Dyorhopapillomavirus 1 faj
- Dyosigmapapillomavirus 1 faj
- Dyothetapapillomavirus 1 faj
- Dyoxipapillomavirus 1 faj
- Dyozetapapillomavirus 1 faj
- Epsilonpapillomavirus 1 faj
- Etapapillomavirus 1 faj
- Gammapapillomavirus 20 faj
- Iotapapillomavirus 1 faj
- Kappapapillomavirus 2 faj
- Lambdapapillomavirus 5 faj
- Mupapillomavirus 2 faj
- Nupapillomavirus 1 faj
- Omegapapillomavirus 1 faj
- Omikronpapillomavirus 1 faj
- Phipapillomavirus 1 faj
- Pipapillomavirus 2 faj
- Psipapillomavirus 1 faj
- Rhopapillomavirus 1 faj
- Sigmapapillomavirus 1 faj
- Taupapillomavirus 2 faj
- Thetapapillomavirus1 faj
- Upsilonpapillomavirus 3 faj
- Xipapillomavirus 2 faj
- Zetapapillomavirus 1 faj
Humán papillómavírusok
[szerkesztés]2013-ig több mint 130 különböző HPV-típust szekvenáltak meg,[9] amelyek 5 nemzetségbe (Alphapapillomavirus, Betapapillomavirus, Gammapapillomavirus, Mupapillomavirus és Nupapillomavirus) tartoznak; azonban legalább 200 további típus vár még szekvenálásra és besorolásra.
Állati papillómavírusok
[szerkesztés]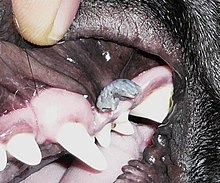
A tünetmentes fertőzést okozó papillómavírusok rendkívül elterjedtek az emlősök körében. Egy vizsgálat során 19 fajhoz tartozó 111 egészséges állatkerti állatról (főemlősökről és patásokról) vettek mintát, amikben 53, addig döntő többségben ismeretlen vírustípust fedeztek fel.[10] A majmok 75-87%-a volt fertőzött. Ezek a vírusok többnyire fajspecifikusak, a vizsgálatban az egyik gondozón is felfedezték a csimpánz-papillómavírust, de az csak felszíni szennyeződésnek bizonyult, néhány hónappal később már nem lehetett kimutatni.
Az észak-amerikai vattafarkú nyulakat (Sylvilagus) fertőző vattafarkú nyúl-papillómavírus (angolul Cottontail rabbit papillomavirus, CRPV; új nómenklatúrában kappapapillómavírus 2) a gazdaállaton egészen nagy felszíni kinövéseket okoz, esetenként a fejen is; talán ez lehetett a szarvasnyúl-legendák, az amerikai jackalope és a német wolpertinger hátterében.[11] A vírus házinyulakban is életben marad, de továbbfertőzni nem képes.[12]
A több faj megfertőzésére képes vírusok egyik példája a szarvasmarha-papillómavírus 1 (BPV-1),[13] amely marhák bőrén nagy szemölcsszerű kinövéseket, lovakon pedig szarkoidnak nevezett jóindulatú bőrtumort okoz. A BPV-1 ellen vakcinát is kidolgoztak.
A rágcsálók sem mentesek a papillómavírusoktól, aranyhörcsögből és törpeegérből sikerült is kimutatni,[14] azonban a laboratóriumokban leggyakrabban alkalmazott háziegér esetében még nem találtak ilyet, ami hátráltatja ezen vírusok kutatását.
Négy, madarakat megfertőző papillómavírus ismert, ezek gazdaállatai az erdei pinty, a sárganyakú frankolin, a jákópapagáj és az Adélie-pingvin.[15] Valamennyiük rendelkezik egy ismeretlen funkciójú génnel (E9), vagyis feltehetően egymás rokonai.
Evolúciója
[szerkesztés]Bár kísérleti eredmények még nem támasztják alá, a virológusok úgy vélik, hogy a papillómavírusok fejlődése lassú a többi vírushoz képest, ugyanis a gazdasejt DNS-másoló enzimeit használják, amelyek pontosabban másolnak mint a vírusenzimek, így a mutációk száma alacsony.
A feltételezések szerint a vírusok együtt fejlődnek gazdaszervezeteikkel, bár erre vannak ellenpéldák is. A HPV-16 jól követi az emberi populáció változásait és variációiból vissza lehet következtetni az emberi migrációra a kontinenseken.[16][17] Ezzel szemben a HPV-13 meglehetősen homogén, sőt nagyon hasonlít a törpecsimpánz papillómavírusára.[18] Lehetséges, hogy nem túl régen ugrott át egyik fajról a másikra, de az is elképzelhető, hogy maga a vírus nem változott sokat az alatt a kb. hatmillió év alatt, amióta az ember és a törpecsimpánz elvált egymástól.[17]
Szerkezete
[szerkesztés]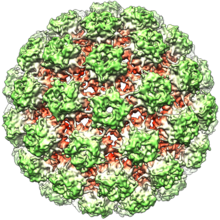
A papillómavírusok kis, 55-60 nanométer átmérőjű vírusok, külső lipidburokkal nem rendelkeznek. Ikozaéderes kapszidjuk az L1 proteinből épül fel. Ez a fehérje felelős a vírusok immunológiai sajátosságaiért és ennek alapján készítik a vakcinákat is. Az L1 ötösével pentamereket alkot, és 72 ilyen pentamerből áll össze a fehérjeburok. Ezzel a papillómavírusok eltérnek minden más ikozaéderes vírustól, amelyeknél csak a 12 csúcsra kerülnek pentamerek és a többi építőegység hat fehérjéből összeálló hexamer.[19]
A vírus ellenálló a környezeti hatásokkal szemben.
Genomja egy kettős szálú, cirkuláris DNS-molekula, amely kb. 8 ezer bázispár hosszú. A DNS-t a gazdasejtből származó hisztonmolekulák stabilizálják a kapszidon belül. A vírusrészecskén belül található még az L2 fehérje, amelynek pontos elhelyezkedése nem ismert, funkciója a vírusgenom kapszidba való csomagolásának, valamint az infekció korai fázisának elősegítése.
Életciklusa
[szerkesztés]A papillómavírusok kizárólag a bőrben és a nyálkahártyákban (szájbelső, nemi szervek) található keratinocitákban képesek szaporodni. A kezdeti infekció a bazális membránhoz közeli, kevéssé differenciált sejteket célozza és a vírus életciklusa szorosan kötődik a keratinociták érési folyamatához. A vírusok a kisebb-nagyobb felszíni sérülések révén érik el gazdasejtjeiket. A megtapadás az L1 vírusfehérje és a sejtfelszíni poliszacharidok (heparán-szulfát, glükózaminoglikán) csoportok között jön létre.[20][21] Ezután az alfa-6 integrin sejtreceptor segítségével endocitózissal bekerül a gazdasejtbe.[22][23] Odabent az L2 fehérje feltárja az endoszóma membránját és a kiszabaduló vírusgenom és a kapcsolódó L2 a sejt transzportmechanizmusait felhasználva a sejtmagba kerül.[24][25]
A sikeres infekció után a vírus elkezdi korai (early, E) génjeinek aktiválását; az E1 és E2 terméke a DNS-másolást és a genom stabilizálását szolgálják, míg az E6 és E7 a sejtosztódást elősegítő (és emiatt potenciálisan onkogén) fehérjéket termelnek, amelyek kikapcsolják a sejtciklust szabályozó és tumorszupresszor p53-at és retinoblasztóma-proteint. Ebben az állapotban a vírusok akár évtizedekig is megbújhatnak a bazális membránon elhelyezkedő keratinocita-őssejtekben.[6]
Egyes vírustípusok elősegítik az epiteliális sejtek rákos elváltozását (bár többségükre ez nem jellemző). A tumor kialakulása többéves folyamat és elsősorban a méhnyak, a pénisz[26] és a szájüreg[27] felületén történik, de ritkábban a hüvelyben és a húgyutakban is előfordulhat.[28]
Az életciklus kései (late, L) szakasza csak a differenciálódott keratinocitákban, a bőr (nyálkahártya) külső rétegeiben aktiválódik és megkezdődik az L1 és L2 fehérjék termelése és a vírusgenom tömeges másolása. Mivel ezek a keratinizálódott sejtrétegek alig elérhetőek az immunrendszer számára, feltételezhető, hogy a vírusok így akarják kikerülni a gazdaszervezet védekezési mechanizmusait.
Az újonnan összeállt vírusrészecskék a sejtmagban gyűlnek össze. Nem pusztítják el a sejtet a kiszabaduláshoz, mint más lipidburok nélküli sejtek teszik, hanem megvárják, míg a bőrfelszíni sejtek maguktól elpusztulnak és szétesnek.
Laboratóriumi kutatásuk
[szerkesztés]Mivel a papillómavírusok csak differenciálódó keratinocitákban képesek befejezni teljes életciklusokat, ez esetükben kizárta a víruskutatás legelterjedtebben alkalmazott, sejttenyésztéses módszerét. Sokáig a szarvasmarha-papillómavírus volt a modellszervezet, mert az állatok bőrén képződött szövetburjánzásokból nagy mennyiségű vírust lehetett kinyerni. Ezenfelül olyan, laboratóriumban tartható állatok vírusait tanulmányozták, mint a vattafarkú nyúl, a házinyúl vagy a kutya.
A szexuálisan átadódó humán papillómavírusok kutatására ún. xenograftokat, immunhiányos egerek bőrébe ültetett emberi bőrsejteket is használnak. A tenyésztett keratinocitákat differenciálódásra lehet bírni ha levegőnek teszik ki és ezzel a módszerrel is előre lehetett jutni a víruskutatásban, ám a technika sok munkával jár és a vírusszaporodás is alacsony.[29][30]
Molekuláris biológiai módszerekkel a vírusok egyes génjeit élesztőbe vitték, ahol a genom replikációja és kapszidba csomagolása jól tanulmányozható. A fertőzés mechanizmusa mesterségesen összeállított pszeudovírusokkal kutatható megfelelően.
A genom szerkezete
[szerkesztés]
A papillómavírus-genom két nagy részre oszlik: az E (early, korai) régióra, ahol közvetlenül az infekció után átíródó gének (hat ORF, E1, E2, E4, E5, E6 és E7) helyezkednek el, valamint az L (late, kései) régió, ahol a kapszidot felépítő L1 és L2 fehérje génje található. Valamennyi gén ugyanarról a DNS-szálról íródik át; ebben eltérnek a poliómavírusoktól, amelyek mindkét DNS-szálat használják gének kódolására. Ez a különbség volt a fő oka annak, hogy a két víruscsoportot külön családba helyezték.
A gazdasejt megfertőzése után aktiválódik az E régió promotere és mind a hat E-génről egyetlen közös, hosszú mRNS íródik át. Ez az RNS három exont és két intront tartalmaz és alternatív splicing révén különböző érett hírvivő RNS-ek képződnek belőle. Az egyik ilyen alternatív formáról, az E6*I-ről íródik át a tumorképzésért felelős egyik onkoprotein, az E7. Az L régió promotere csak differenciált sejtekben lesz aktív, és szintén egy hosszú, közös RNS képződik róla, amely két intront és három exont tartalmaz.
Az egyes gének funkciója a következő:
- E1 – a vírusgenom másolásának megkezdése a feladata, a fehérje a replikációs kezdőponthoz köt és szétcsavarja a DNS szálait, hogy a gazdasejt másolóenzimjei hozzáférhessenek.
- E2 – szabályozó protein; a vírus génjeinek kifejeződését regulálja, ő felelős a látens infekcióért, gátolja az E6 és E7 onkogének átírását, lehetővé teszi, hogy a sejtosztódáskor a vírus-DNS-ek egyenlően oszoljanak el a leánysejtekben[31]
- E3 – csak néhány fajban található meg, funkciója ismeretlen
- E4 – bár az E régióban van, nagy mennyiségben csak a fertőzés késői fázisában képződik. Feladata a sejt citoszkeletonjának bontása és a vírusrészecskék kiszabadulásának elősegítése. Azt is kimutatták, hogy gátolja a sejtosztódást, a G2 fázisban megállítja a sejtciklust.
- E5 – kis, hidrofób proteinek, amelyek a sejt saját, membránkötött fehérjéit képesek gátolni.[32] Egyes vírusok (BPV-1) esetében onkogénként is szolgálnak a sejtosztódást szabályozó szignálproteinekhez való kötődéssel. Gátolja a fő hisztokompatibilitási komplex sejtfelszíni kifejeződését és ezzel segíti elrejteni a fertőzött sejteket az immunrendszer elől
- E6- onkogén, fő funkciója a sejtbeli p53 tumorszupresszor fehérje gátlása. Ezenkívül aktiválja a telomeráz enzimet (ami lehetővé teszi a végtelen számú osztódást a sejt kromoszómavégeinek javítása révén) és szabályzófehérjéket (főleg kinázokat) is megköt.
- E7 – onkogén, a tumorképződést gátló retinoblasztóma-proteint (pRb) inaktiválja. Gátolja az apoptózist és elősegíti a sejtosztódást. Szintén képes aktiválni a telomerázt.
- E8 – csak néhány vírusban található meg. A HPV-knél az E5-höz hasonlít és E5/E8 a neve, a BPV-4 esetében az E6-ot helyettesíti
- L1 – a kapszid fő komponense. Az L1 alapegységek spontán öt proteinből álló pentamerekké állnak össze, amelyek szintén külön behatás nélkül kész víruskapszidot képesek alkotni, amelyeket az L1 fehérjék közötti diszulfid kötések stabilizálnak. Szekvenciája a többi vírusgénhez képest konzervatív, kivéve a felszíni polipeptid-hurkokat, amelyek változékonyak, hogy a gazdaszervezet védekezőrendszerét könnyebben kikerülhesse.[33]
- L2 – az L2 segít a vírusgenom kapszidba való csomagolásában, valamint a fertőzés kezdeti szakaszában, miután a vírus endocitózissal bekerült a sejt belsejébe, a savassá váló környezet jelére az L2 destabilizálja az endoszóma membránját és a vírus beszabadul a citoplazmába.
Jegyzetek
[szerkesztés]- ↑ Herbst LH, Lenz J, Van Doorslaer K, Chen Z, Stacy BA, Wellehan JF Jr, Manire CA, Burk RD (2009). „Genomic characterization of two novel reptilian papillomaviruses, Chelonia mydas papillomavirus 1 and Caretta caretta papillomavirus 1”. Virology 383 (1), 131–5. o. DOI:10.1016/j.virol.2008.09.022. PMID 18973915.
- ↑ Drury SE, Gough RE, McArthur S, Jessop M (1998). „Detection of herpesvirus-like and papillomavirus-like particles associated with diseases of tortoises”. Vet Rec. 143 (23), 639. o. PMID 9881444.
- ↑ Lange CE, Favrot C, Ackermann M, Gull J, Vetsch E, Tobler K. (2011). „Novel snake papillomavirus does not cluster with other non-mammalian papillomaviruses”. Virol J. 8, 436. o. DOI:10.1186/1743-422X-8-436. PMID 21910860.
- ↑ Muñoza N, Castellsaguéb X, Berrington de Gonzálezc A, Gissmann L (2006). „Chapter 1: HPV in the etiology of human cancer”. Vaccine 24 (3), S1–S10. o. DOI:10.1016/j.vaccine.2006.05.115. PMID 16949995.
- ↑ Mistry N, Wibom C, Evander M (2008). „Cutaneous and mucosal human papillomaviruses differ in net surface charge, potential impact on tropism”. Virol J. 5, 118. o. DOI:10.1186/1743-422X-5-118. PMID 18854037.
- ↑ a b Doorbar J (2005). „The papillomavirus life cycle”. J. Clin. Virol. 32 Suppl 1, S7–15. o. DOI:10.1016/j.jcv.2004.12.006. PMID 15753007.
- ↑ Virus Taxonomy: 2014 Release. (Hozzáférés: 2015. június 15.)
- ↑ Campo MS (editor).. Papillomavirus Research: From Natural History To Vaccines and Beyond. Caister Academic Press. [1] (2006). ISBN 978-1-904455-04-2
- ↑ Chouhy D, Bolatti EM, Perez GR, Giri AA (2013) Analysis of the genetic diversity and phylogenetic relationships of putative human papillomavirus types. J Gen Virol
- ↑ Antonsson A, Hansson BG (2002). „Healthy skin of many animal species harbors papillomaviruses which are closely related to their human counterparts”. J. Virol. 76 (24), 12537–42. o. DOI:10.1128/JVI.76.24.12537-12542.2002. PMID 12438579.
- ↑ Holliday, Chuck: Prof. Chuck Holliday's www page at Lafayette College » Jackalopes. [2014. július 18-i dátummal az eredetiből archiválva]. (Hozzáférés: 2014. július 13.)
- ↑ Christensen ND (2005). „Cottontail rabbit papillomavirus (CRPV) model system to test antiviral and immunotherapeutic strategies”. Antivir. Chem. Chemother. 16 (6), 355–62. o. PMID 16331841.
- ↑ Coggins LW, Ma JQ, Slater AA, Campo MS (1985). „Sequence homologies between bovine papillomavirus genomes mapped by a novel low-stringency heteroduplex method”. Virology 143 (2), 603–11. o. DOI:10.1016/0042-6822(85)90398-8. PMID 2998027.
- ↑ Van Ranst M, Tachezy R, Pruss J, Burk RD (1992). „Primary structure of the E6 protein of Micromys minutus papillomavirus and Mastomys natalensis papillomavirus”. Nucleic Acids Res. 20 (11), 2889. o. DOI:10.1093/nar/20.11.2889. PMID 1319576.
- ↑ Varsani A1, Kraberger S, Jennings S, Porzig EL, Julian L, Massaro M, Pollard A, Ballard G, Ainley DG (2014) A novel papillomavirus in Adelie penguin (Pygoscelis adeliae) faeces sampled at the Cape Crozier colony, Antarctica. J Gen Virol doi: 10.1099/vir.0.064436-0
- ↑ Ho L, Chan SY, Burk RD, et al. (1993). „The genetic drift of human papillomavirus type 16 is a means of reconstructing prehistoric viral spread and the movement of ancient human populations”. J. Virol. 67 (11), 6413–23. o. PMID 8411343.
- ↑ a b Calleja-Macias IE, Villa LL, Prado JC, et al. (2005). „Worldwide genomic diversity of the high-risk human papillomavirus types 31, 35, 52, and 58, four close relatives of human papillomavirus type 16”. J. Virol. 79 (21), 13630–40. o. DOI:10.1128/JVI.79.21.13630-13640.2005. PMID 16227283.
- ↑ Van Ranst M, Fuse A, Fiten P, et al. (1992). „Human papillomavirus type 13 and pygmy chimpanzee papillomavirus type 1: comparison of the genome organizations”. Virology 190 (2), 587–96. o. DOI:10.1016/0042-6822(92)90896-W. PMID 1325697.
- ↑ Rayment, I., et al. "Polyoma virus capsid structure at 22.5 A resolution." Nature 295.5845 (1982): 110-115.
- ↑ Joyce JG, Tung JS, Przysiecki CT, et al. (1999). „The L1 major capsid protein of human papillomavirus type 11 recombinant virus-like particles interacts with heparin and cell-surface glycosaminoglycans on human keratinocytes”. J. Biol. Chem. 274 (9), 5810–22. o. DOI:10.1074/jbc.274.9.5810. PMID 10026203.
- ↑ Giroglou T, Florin L, Schäfer F, Streeck RE, Sapp M (2001). „Human papillomavirus infection requires cell surface heparan sulfate”. J. Virol. 75 (3), 1565–70. o. DOI:10.1128/JVI.75.3.1565-1570.2001. PMID 11152531.
- ↑ Evander M, Frazer IH, Payne E, Qi YM, Hengst K, McMillan NA (1997). „Identification of the alpha6 integrin as a candidate receptor for papillomaviruses”. J. Virol. 71 (3), 2449–56. o. PMID 9032382.
- ↑ McMillan NA, Payne E, Frazer IH, Evander M (1999). „Expression of the alpha6 integrin confers papillomavirus binding upon receptor-negative B-cells”. Virology 261 (2), 271–9. o. DOI:10.1006/viro.1999.9825. PMID 10497112.
- ↑ Kämper N, Day PM, Nowak T, et al. (2006). „A membrane-destabilizing peptide in capsid protein L2 is required for egress of papillomavirus genomes from endosomes”. J. Virol. 80 (2), 759–68. o. DOI:10.1128/JVI.80.2.759-768.2006. PMID 16378978.
- ↑ Day PM, Baker CC, Lowy DR, Schiller JT (2004). „Establishment of papillomavirus infection is enhanced by promyelocytic leukemia protein (PML) expression”. Proc. Natl. Acad. Sci. U.S.A. 101 (39), 14252–7. o. DOI:10.1073/pnas.0404229101. PMID 15383670.
- ↑ Do HT, Koriyama C, Khan NA, Higashi M, Kato T, Le NT, Matsushita S, Kanekura T, Akiba S (2013) The etiologic role of human papillomavirus in penile cancers: a study in Vietnam. Br J Cancer doi: 10.1038/bjc.2012.583
- ↑ Gogilashvili K, Shonia N, Burkadze G (2012) The role of human papillomavirus in oral squamous cell carcinoma (review). Georgian Med News (213):32-36
- ↑ Tolstov Y, Hadaschik B, Pahernik S, Hohenfellner M, Duensing S (2013) Human papillomaviruses in urological malignancies: A critical assessment. Urol Oncol. 2013 Oct 17. pii: S1078-1439(13)00282-2. doi: 10.1016/j.urolonc.2013.06.012
- ↑ Meyers C, Frattini MG, Hudson JB, Laimins LA (1992). „Biosynthesis of human papillomavirus from a continuous cell line upon epithelial differentiation”. Science 257 (5072), 971–3. o. DOI:10.1126/science.1323879. PMID 1323879.
- ↑ McLaughlin-Drubin ME, Christensen ND, Meyers C (2004). „Propagation, infection, and neutralization of authentic HPV16 virus”. Virology 322 (2), 213–9. o. DOI:10.1016/j.virol.2004.02.011. PMID 15110519.
- ↑ McBride AA, McPhillips MG, Oliveira JG (2004). „Brd4: tethering, segregation and beyond”. Trends Microbiol. 12 (12), 527–9. o. DOI:10.1016/j.tim.2004.10.002. PMID 15539109.
- ↑ Bravo IG, Alonso A (2004). „Mucosal human papillomaviruses encode four different E5 proteins whose chemistry and phylogeny correlate with malignant or benign growth”. J. Virol. 78 (24), 13613–26. o. DOI:10.1128/JVI.78.24.13613-13626.2004. PMID 15564472.
- ↑ Carter JJ, Wipf GC, Madeleine MM, Schwartz SM, Koutsky LA, Galloway DA (2006). „Identification of human papillomavirus type 16 L1 surface loops required for neutralization by human sera”. J. Virol. 80 (10), 4664–72. o. DOI:10.1128/JVI.80.10.4664-4672.2006. PMID 16641259.
További információk
[szerkesztés]- Viralzone: Papillomaviridae
- 00.099. Papillomaviridae description In: ICTVdB – The Universal Virus Database, version 4. Büchen-Osmond, C. (Ed), Columbia University, New York, USA
- Human papillomavirus particle and genome visualization
- ICTV
Fordítás
[szerkesztés]- Ez a szócikk részben vagy egészben a Papillomaviridae című angol Wikipédia-szócikk ezen változatának fordításán alapul. Az eredeti cikk szerkesztőit annak laptörténete sorolja fel. Ez a jelzés csupán a megfogalmazás eredetét és a szerzői jogokat jelzi, nem szolgál a cikkben szereplő információk forrásmegjelöléseként.



