Alternatív splicing

Az alternatív splicing a génexpresszió egyik szabályozott folyamata. Ezen folyamat során a génből kivágódhatnak bizonyos exonok, amelyek így nem vesznek részt a végső, transzláció előtt álló messenger RNS képzésében. Ebből az következik, hogy azok a proteinek, amelyek alternatív splicing-on átesett mRNS-ről képződtek, különbözhetnek aminosav szekvenciájukban, és ezért még biológiai funkciójukban is annak ellenére, hogy egy génről íródtak át (lásd ábra). Különösen igaz ez a humán genomra, amely alternatív splicinggal képes arra, hogy sokkal több fehérjét szintetizáljon annál, mint amennyi a 20.000 fehérjét kódoló génjétől elvárható volna. Az alternatív splicingot néha "differential splicingnek" is nevezik, de ez a kifejezés nem tartalmazza a folyamat géntermék növelő hatását.
Az eukariótáknál az alternatív splicing egy természetes jelenség, amely nagyon megnövelheti az egy genom által kódolt proteinek biodiverzitását; az embereknél a többexonos (multiexonos) gének 95%-a átmegy ezen a folyamaton. A kutatók számos módját fedezték már fel az alternatív splicingnak, de mindközül az exonátugrás (exon skipping) a legáltalánosabb. Ekkor bizonyos exonok bizonyos körülmények között bizonyos szövetekben átíródnak az mRNS-ekbe, míg más esetekben egyszerűen kihagyják, átugorják őket.[1]
Az alternatív splicing szabályozása az elsődleges transzkriptumon történik, a cisz regulátor (cis-acting) helyekkel kapcsolatban lévő transz regulátor (trans-acting) fehérjékkel. A splicing aktivátor fehérjék megnövelik egyes splicing helyek aktivitását, míg más splicing represszorok lecsökkentik azt. Az alternatív splicing mechanizmusa nagyon sokrétű, a tudósok a technika (high-throughput) segítségével folyamatosan fedeznek fel újakat. A kutatók remélik, hogy a regulációs rendszer teljes megfejtésével képesek lesznek ún. "splicing kódokkal" megjósolni, hogy egy adott génről, bizonyos körülmények között az alternatív splicing során milyen géntermék jön létre.[2][3]
A splicing során létrejövő abnormális változatok szintén okozhatnak betegséget; az emberi genetikai betegségek nagy része egy ilyen hibás splicing variánsból származik. Ezek a variánsok (splicoformok) akár rák[4][5][6] kifejlődéséhez is vezethetnek, habár egy egészséges szervezetben egy poszttranszkripciós ellenőrző mechanizmuson keresztül hatástalanítódnak vagy eltávolításra kerülnek.[5][6][7]
Felfedezése
Az alternatív splicing folyamatát először 1977-ben jegyezték le.[8][9] Az adenovírusok a fertőzési ciklusuk elején 5 elsődleges transzkriptumot szintetizálnak, még a DNS-ük megkettőződése előtt, majd további egyet a DNS replikáció megkezdése után. Az előbbiek képződése folytatódik a DNS megkettőződés megkezdődése után is. Az utóbbi, amelyik már a kései fertőzési szakaszban szintetizálódik, nagy, az 5/6-át adja a teljes 32kb hosszúságú adenovírus genomnak. Ez sokkal nagyobb azoknál az mRNS-eknél, amit egy adenovírus megfertőzött sejtben képes létrehozni. A kutatók rájöttek arra, hogy a 2-es típusú adenovírus által létrehozott elsődleges transzkriptumok a kései fázisban különbözőképpen illesztődnek össze, ezzel olyan mRNS-eket eredményeznek, amelyek számos különböző vírusfehérjét szintetizálnak. Továbbá az elsődleges transzkriptum több poliadenilációs helyet tartalmaz, amely eltérő 3’ végeket eredményezhet a képződő mRNS-eknek.[10][11][12]
1981-ben fedezték fel először egy normál, endogén eredetű gén által kódolt transzkriptumban az alternatív splicingot.[10] Ez a gén a calcitonin nevű thyroid hormont kódolja, amely keletkezése során alternatív splicingon megy keresztül az emlős sejtekben. Az erről a génről képződő elsődleges transzkriptum 6 exont tartalmaz; a calcitonin mRNS-e 1-től 4-ig tartalmazza ezeket az exonokat, és a 4. exonban található poliadenilációs rész után befejeződik. Egy ugyanerről a pre-mRNS-ről képződő mRNS esetében a 4-es exon kihagyódik (exon skipping), így ez az 1-es 2-es 3-as, 5-ös, 6-os exonokat tartalmazza. Ez a termék már CGRP (calcitonin gene related peptide) névű proteint kódolja.[13][14] Szintén a 80-as évek elején fedezték fel az emlősök immunglobulin génjeinek transzkriptumaiban végbemenő alternatív splicingot.[10][15]
Ettől kezdve ez a folyamat általánossá vált a kutatók számára az eukariótákban.[1] Az alternatív splicing „rekordtartója” az ecetmuslica (Drosophila melanogaster) Dscam-nak nevezett génje, amely potenciálisan 38,016 splicing variánst képes képezni.[16]
Fajtái

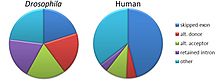
5 általánosan elfogadott fajtáját ismerjük az alternatív splicingnak.[1][2][17][18]
- Exonátugrás (exon skipping vagy kazettás exonok): ebben az esetben egy exon vagy kivágódik az elsődleges transzkriptumból, vagy bennmarad. Ez a legelterjedtebb az emlősök sejtjeiben.[17]
- Egymást kizáró exonok (mutually exclusive exons): egy vagy két exon bennmarad, de nem mindkettő.
- Alternatív donor hely: : egy exon 5’ végéhez (donor hely) már alternatívan kötődött egy másik (sárga) exon, ezért az ezzel történő csatlakozás során alternatívan változik egy különálló (5' vég felőli, upstream) exon 3’ vége.
- Alternatív akceptor hely: egy exon 3’ végéhez (akceptor hely) már alternatívan kötődött egy másik (sárga) exon, így az ehhez kapcsolódó (3’ vég felőli, downstream) exonnak alternatívan változik az 5’ vége.
- Intron retenció: egy szekvencia egyszerűen kivágódhat, mint egy intron, vagy bennmaradhat a transzkriptumban. Az különbözteti meg az exonátugrástól, hogy itt a bennmaradt exonokat nem szegélyezik intronok. Ha a bennmaradt intron a kódoló régióhoz tartozik, akkor vagy fehérjét szintetizál a mellette lévő exonokkal együtt, vagy egy stop kodont tartalmaz, vagy elmozdíthatja a leolvasási keretet, ami a képződő fehérje funkcióvesztésével is járhat. Ez a legritkább eset az emlősökben.[17]
A fent említettek csak az elsődleges fajtái voltak az alternatív splicing-nek, ezenkívül további két másik fő mechanizmus is ismert, amelyek szintén különböző mRNS-eket eredményeznek ugyanarról a génről; a többszörös promóterek valamint a többszörös poliadenilációs helyek. A többszörös promótereket inkább tekinthetjük egyfajta transzkripciós szabályozó mechanizmusnak, mint alternatív splicingnak; a transzkripció különböző helyeken való elkezdése, amelyre a legtöbb exon képessé tehető, eltérő 5’ végeket eredményez. Hasonlóan a másik mechazimus a poliadenilációs helyekkel különböző 3’ végeket képezhet. Mindkét folyamat egyfajta keveréket alkot az alternatív splicinggal, amely tovább növeli az egy génről származó mRNS-ek sokféleségét.[1][2]

A fentiek alap splicing mechanizmusok, ezért lehet, hogy nem elegendőek ahhoz, hogy összetett splicing folyamatokat tudjunk értelmezni. Például a jobb oldali ábrán 3 különböző transzkriptumot (spliceformot) az egér 3-as hyaluronidáz génjéről. Az exon struktúrák összehasonlítása során látható, hogy a második (sárga) és az első (zöld) transzkriptum között exon retenció jöhetett létre, míg a harmadik (kék) és a második spliceform között egy exonátugrás (exon skipping) okozhatja a különbséget. Egy újonnan javasolt modell nevezéktan egyedileg nevez meg minden lehetséges splicing mintázatot.[17]
Az alternatív splicing mechanizmusai
Általános splicing mechanizmusok

Amikor a DNS-ről egy pre-mRNS képződik, akkor az még számos intront és exont tartalmaz. A fonálférgeknél (Nematoda sp.) ez 4-5 intront és exont jelent, míg a musclicánál (Drosophila melanogaster) több mint 100 intron és exon is lehet egyben). Hogy melyik exonok kerülnek a kész mRNS-be, az a splicing folyamata alatt dől el. A szabályozását, és a kiválogatását ezeknek a splicing helyeknek transz regulátor (trans-acting) aktivátor és represszor fehérjék végzik.
Egy tipikus eukarióta sejtmagban a legfontosabb régiók intronjai konszenzus szekvenciákat tartalmaznak. Minden intron az 5’ végén GU-val (guanin, uracil) fejeződik be. A 3’ véghez közel egy elágazási hely (branch site) található. Az elágazási pontban (branch point) általában egy A (adenin) nukleotid található; itt valamelyest különböznek a konszenzus szekvenciák. Az emberek esetében a bázissorrend itt yUnAy.[19] Az elágazási helyet egy polipirimidin (Y) sor követ – A polipirimidin szakasz – amely egészen a 3’ véget lezáró AG részig tart.[2]
Az mRNS splicing olyan RNS és fehérje komplexek között jön létre, amelyek az U1,U2, U4, U5, U6 (U3 nem vesz részt az mRNS splicingban) jelű snRNP-ket (small nuclear ribonucleic particles) tartalmazzák, és amelyek a transzkriptumhoz csatlakozva létrehozzák a spliceoszómának is nevezett képződményt.[20] Az U1 az intron 5’ végén található GU részhez, valamint az U2-höz is csatlakozik, amely az U2AF komplexen keresztül az elágazási pont A (adenin) nukleotidjához kötődik (lásd ábra). Ezt a csatlakozást ebben a szakaszban spliceoszóma A komplexnek nevezik. A komplex A létrejötte általában egy olyan kulcsfontosságú lépés, amely már meghatározza a kivágásra szánt intron végeit, és ezzel a bennmaradt exon végeit is.[2]
Az U4, U5, U6 komplex bekötődésével az U6 elfoglalja az U1 helyét, mire az az U4 együtt leválik. Az így létrejött komplex ezután két transzészterezési reakciót végez el. Az első reakció során az intron 5’ vége levágódik a transzkriptum 5’ végéhez közelebb lévő (upstream) exonról (kék) és egy 2’,5’ foszfodiészter kötéssel kapcsolódik az elágazási pont adeninjéhez. A második reakcióban az intron 3’ vége is levágódik immár a 3’ véghez közelebbi (downstream) exonról (sárga), majd a két exon egy foszfodiészter kötéssel összekapcsolódik. A kivágódott intron ezután egy lasszószerű formát vesz fel, majd degradálódik.[1]
A szabályozó elemek és fehérjék

A splicing szabályozása a transz regulátor (trans-acting) fehérjék (represszorok és aktivátorok) és a velük kapcsolatban lévő pre-mRNS-en található cisz regulátor (cis-acting) helyeken (silencer, és enhancer) keresztül történik. Azonban az alternatív splicing bonyolultságát növeli az a kimutatott tény is, miszerint a splicing-faktorok hatása erősen függ a helytől, ahová csatlakozik. Azaz, ha egy splicing faktor aktivátorként funkcionál, amikor egy intronon lévő enhancer (erősítő) részhez csatlakozik, akkor lehet, hogy represszorként viselkedik amikor egy exonon lévő helyhez csatlakozik, és fordítva.[21] A pre-mRNS másodlagos szerkezete a splicing elemek mozgatásával, valamint bizonyos splicing faktor kötőhelyek eltakarásával szintén szerepet játszik a folyamat szabályozásában.[22][23] A fent említettek együtt alkotják az ún. „splicing kódot” amely meghatározza, hogy bizonyos sejtekben a splicing miként menjen végbe.[24][25]
Két fő típusa van a pre-mRNSen található cisz-regulációs RNS elemeknek, amelyek a transz-regulációs RNS kötő proteinekkel is kapcsolatban állnak. Az egyik splicing silencer, amely olyan hely, ahová a represszor kötődik lecsökkentve ezzel annak az esélyét, hogy a hozzá közeli helyek egy splicing kapcsolatban szerepeljenek. Ezek a helyek az intronban (intronic splicing silencers, ISS) vagy a vele szomszédos exonban (exonic splicing silencers, ESS) találhatók. Közel annyi féle ilyen szekvencia van, ahány féle protein kötődhet oda. A splicing represszorok többsége hnRNP (heterogeneous nuclear ribonucleoprotein) mint például a hnRNPA1 és a polipirimidin szakaszhoz kötődő PTB (polypyrimidine tract binding) fehérje. .[2][24] A másik, a splicing enhancer hely, ahová a splicing aktivátor kötődik, ezzel megnövelve annak az esélyét, hogy a hozzá közeli helyek egy splicing kapcsolatban szerepeljenek. Ezek szintén lehetnek egy intronon belül (intronic splicing enhancers, ISE) vagy egy exonban (exonic splicing enhancers, ESE). A legtöbb aktivátor fehérje, amely az ISE vagy az ESE helyekhez kötődik az SR protein családhoz tartozik. Az ilyen proteinek RNS felismerő részeket valamint argininben és szerinben gazdag (RS) doménokat hordoznak.[2][24]
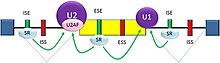
Általában a splicing determinánsok egy a környezet által befolyásolt összefüggő rendszert alkotnak, amely megszabja, hogy hogyan legyen a folyamat szabályozva a splicing kód által[25] A kontextustól függően bizonyos cisz-regulációs RNS szekvencia részeknek a jelenléte egyes esetekben megnöveli annak a valószínűségét, hogy a közelében levő szekvenciák kivágódjanak, míg más esetekben lecsökkenti azt. Egy ilyen környezetben az egyes szabályozó elemek (pl.: cisz-reguláció) kapcsolata más RNS szekvenciák és transz-regulációs elemek jelenlétének köszönhető, amelyek viszont a sejt állapotától függenek. Például néhány cisz-regulátor RNS szekvencia csak akkor befolyásolja a splicingot ha több más elem is jelen van ugyanabban a régióban, hogy a kapcsolatot stabilizálják. Egy másik esetben a cisz-regulátor elemek folyamatra gyakorolt hatása attól függ, hogy éppen milyen fehérjék szintetizálódtak a sejtben (pl.: neurális és nem neurális PTB). A splicing silencer-ek és enhancerek adaptív szignifikanciáját, egy tanulmány bizonyította, miszerint az emberi génekben nagy szelekciós nyomás nehezedik azokra a mutációkra, amelyek úgy silencereket képeznek vagy a létező exonokat inaktiválnak.[26][27]
Jegyzetek
- ↑ a b c d e Black, Douglas L. (2003). „Mechanisms of alternative pre-messenger RNA splicing”. Annual Reviews of Biochemistry 72 (1), 291–336. o. DOI:10.1146/annurev.biochem.72.121801.161720. PMID 12626338.
- ↑ a b c d e f g h Matlin, AJ, Clark F, Smith, CWJ (2005. május 1.). „Understanding alternative splicing: towards a cellular code”. Nature Reviews 6 (5), 386–398. o. DOI:10.1038/nrm1645. PMID 15956978.
- ↑ (2008) „The search for alternative splicing regulators: new approaches offer a path to a splicing code”. Genes & Development 22 (3), 279–85. o. DOI:10.1101/gad.1643108. PMID 18245441.
- ↑ Skotheim, R I and Nees, M (2007). „Alternative splicing in cancer: noise, functional, or systematic?”. The international journal of biochemistry & cell biology 39 (7-8), 1432–49. o. DOI:10.1016/j.biocel.2007.02.016. PMID 17416541.
- ↑ a b (2009) „A Global View of Cancer-Specific Transcript Variants by Subtractive Transcriptome-Wide Analysis”. PLoS ONE 4 (3), e4732. o. DOI:10.1371/journal.pone.0004732. PMID 19266097.
- ↑ a b Fackenthal, Jd; Godley, La (2008). „Aberrant RNA splicing and its functional consequences in cancer cells” (Free full text). Disease models & mechanisms 1 (1), 37–42. o. DOI:10.1242/dmm.000331. PMID 19048051.
- ↑ Danckwardt S, Neu-Yilik G, Thermann R, Frede U, Hentze MW, Kulozik AE (2002). „Abnormally spliced beta-globin mRNAs: a single point mutation generates transcripts sensitive and insensitive to nonsense-mediated mRNA decay”. Blood 99 (5), 1811–6. o. [2009. február 3-i dátummal az eredetiből archiválva]. DOI:10.1182/blood.V99.5.1811. PMID 11861299. (Hozzáférés: 2014. augusztus 13.)
- ↑ Chow LT, Gelinas RE, Broker TR, Roberts RJ (1977). „An amazing sequence arrangement at the 5' ends of adenovirus 2 messenger RNA”. Cell 12 (1), 1–8. o. DOI:10.1016/0092-8674(77)90180-5. PMID 902310.
- ↑ Berget SM, Moore C, Sharp PA (1977). „Spliced segments at the 5' terminus of adenovirus 2 late mRNA”. Proc. Natl. Acad. Sci. U.S.A. 74 (8), 3171–5. o. DOI:10.1073/pnas.74.8.3171. PMID 269380.
- ↑ a b c Leff SE, Rosenfeld MG, Evans RM (1986). „Complex transcriptional units: diversity in gene expression by alternative RNA processing”. Annu. Rev. Biochem. 55 (1), 1091–117. o. DOI:10.1146/annurev.bi.55.070186.005303. PMID 3017190.
- ↑ Chow LT, Broker TR (1978). „The spliced structures of adenovirus 2 fiber message and the other late mRNAs”. Cell 15 (2), 497–510. o. DOI:10.1016/0092-8674(78)90019-3. PMID 719751.
- ↑ Nevins JR, Darnell JE (1978). „Steps in the processing of Ad2 mRNA: poly(A)+ nuclear sequences are conserved and poly(A) addition precedes splicing”. Cell 15 (4), 1477–93. o. DOI:10.1016/0092-8674(78)90071-5. PMID 729004.
- ↑ Rosenfeld MG, Amara SG, Roos BA, Ong ES, Evans RM (1981). „Altered expression of the calcitonin gene associated with RNA polymorphism”. Nature 290 (5801), 63–5. o. DOI:10.1038/290063a0. PMID 7207587.
- ↑ Rosenfeld MG, Lin CR, Amara SG, et al. (1982). „Calcitonin mRNA polymorphism: peptide switching associated with alternative RNA splicing events”. Proc. Natl. Acad. Sci. U.S.A. 79 (6), 1717–21. o. DOI:10.1073/pnas.79.6.1717. PMID 6952224.
- ↑ Maki R, Roeder W, Traunecker A, et al. (1981). „The role of DNA rearrangement and alternative RNA processing in the expression of immunoglobulin delta genes”. Cell 24 (2), 353–65. o. DOI:10.1016/0092-8674(81)90325-1. PMID 6786756.
- ↑ Schmucker D, Clemens JC, Shu H, Worby CA, Xiao J, Muda M, Dixon JE, Zipursky SL (2000). „Drosophila Dscam is an axon guidance receptor exhibiting extraordinary molecular diversity”. Cell 101 (6), 671–84. o. DOI:10.1016/S0092-8674(00)80878-8. PMID 10892653.
- ↑ a b c d e Michael Sammeth (2008). „A general definition and nomenclature for alternative splicing events”. PLoS Comput Biol. 4 (8), e1000147. o. DOI:10.1371/journal.pcbi.1000147. PMID 18688268.
- ↑ Pan, Q (2008. december 1.). „Deep surveying of alternative splicing complexity in the human transcriptome by high-throughput sequencing”. Nature Genetics 40 (12), 1413–1415. o. DOI:10.1038/ng.259. PMID 18978789.
- ↑ (2008) „Human branch point consensus sequence is yUnAy”. Nucleic Acids Research 36 (7), 2257–67. o. DOI:10.1093/nar/gkn073. PMID 18285363.
- ↑ Clark, David. Molecular biology. Amsterdam: Elsevier Academic Press (2005). ISBN 0-12-175551-7
- ↑ Lim KH, Ferraris L, Filloux ME, Raphael BJ, Fairbrother WG (2011). "Using positional distribution to identify splicing elements and predict pre-mRNA processing defects in human genes". Proc. Natl. Acad. Sci. USA 108 (27): 11093–11098.
- ↑ Warf MB, Berglund JA (2010). "Role of RNA structure in regulating pre-mRNA splicing". Trends Biochem. Sci. 35 (3): 169–178.
- ↑ Reid DC, Chang BL, Gunderson SI, Alpert L, Thompson WA, Fairbrother WG (2009). "Next-generation SELEX identifies sequence and structural determinants of splicing factor binding in human pre-mRNA sequence". RNA 15 (12): 2385–2397.
- ↑ a b c Wang, Z; Burge, Cb (2008). „Splicing regulation: from a parts list of regulatory elements to an integrated splicing code” (Free full text). RNA 14 (5), 802–13. o. DOI:10.1261/rna.876308. PMID 18369186.
- ↑ a b Barash, Y; et al (2010). „Deciphering the splicing code”. Nature 465 (7294), 53–59. o. DOI:10.1038/nature09000. PMID 20445623.
- ↑ Ke S, Zhang XH, Chasin LA (2008). „Positive selection acting on splicing motifs reflects compensatory evolution”. Genome Res. 18 (4), 533–43. o. DOI:10.1101/gr.070268.107. PMID 18204002.
- ↑ Fairbrother WG, Holste D, Burge CB, Sharp PA (2004). "Single nucleotide polymorphism–based validation of exonic splicing enhancers". PLoS Biol 2 (9): e268.
Fordítás
- Ez a szócikk részben vagy egészben az Alternative splicing című angol Wikipédia-szócikk ezen változatának fordításán alapul. Az eredeti cikk szerkesztőit annak laptörténete sorolja fel. Ez a jelzés csupán a megfogalmazás eredetét és a szerzői jogokat jelzi, nem szolgál a cikkben szereplő információk forrásmegjelöléseként.
