RNS-polimeráz II
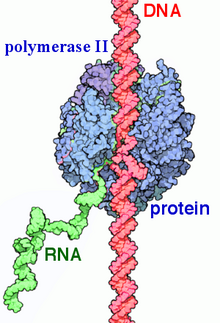
Az RNS-polimeráz II (RNSP II, Pol II DNS-t pre-mRNS-re és a legtöbb kis magi RNS-re és mikro-RNS-re átíró fehérjekomplex.[1][2] Az eukarióták 3 RNS-polimerázának egyike.[3] 550 kDa-os, 12 alegységes komplex, és az egyik leginkább tanulmányozott RNS-polimeráz-típus. Számos transzkripciós faktor kell a korábbi promoterek kötéséhez és a transzkripció elindításához.
Felfedezés[szerkesztés]

A korai tanulmányok legalább 2 RNSP-t feltételeztek: egy rRNS-előállítót a nukleóluszban és egy más RNS-t a nukleoplazmában – a sejtmag nukleóluszon kívüli részén – előállítót.[5] 1969-ben Robert G. Roeder és William J. Rutter felfedezték, hogy 3 különböző magi RNS-polimeráz van – egy 3. felelt bizonyos RNS-ek nukleoplazmabeli átírásáért.[6] Ezt ioncsere-kromatográfia révén érték el DEAE-vel burkolt Sephadex gyöngyökkel. Ez az enzimeket megfelelő elúciók szerint választotta el I, II, III sorrendben az ammónium-szulfát-koncentráció növelésével. Az enzimeket elúciós sorrendben nevezték el RNS-polimeráz I-nek, II-nek és III-nak.[3] E felfedezés kimutatta, hogy van egy további enzim a nukleoplazmában, lehetővé téve az RNSP II és III megkülönböztetését.[7]
Az RNS-polimeráz II (RNSP2) szabályzott transzkripciós szüneteket tart az elongáció elején. Több tanulmány kimutatta az elongációs zavarok kapcsolatát a rákkal, a neurodegenerációval, a HIV-látenciával stb.[8]
Alegységek[szerkesztés]

Az eukarióta RNS-polimeráz II-magot először transzkripciós assay-kkel mutatták ki.[10] A tiszta enzim általában 10–12 alegységet (emberben és élesztőben 12-t) tartalmaz, és nem képes specifikus promoterfelismerésre.[11] Many subunit-subunit interactions are known.[12]
- RPB1 DNS-irányított RNS-polimeráz II-alegység: az emberben a POLR2A, élesztőben az RPO21 által kódolt enzim. A legnagyobb RNS-polimeráz II-egység. Maximum 52 YSPTSPS-ismétlődésből álló, a polimerázaktivitáshoz szükséges C-terminális doménje van.[13] Ezt először a Torontói Egyetemen dolgozó C. J. Ingles és a Johns Hopkins Egyetemen dolgozó J. L. Corden mutatták ki.A többi alegységgel együtt alkotja a polimeráz DNS-kötő doménjét, ahol a templát RNS-be való transzkripciója történik.[14] Erősen kölcsönhat az RPB8-cal.[12]
- RPB2 (POLR2B): a másodig legnagyobb alegység, mely legalább 2 másik polimerázzal kölcsönhat, szerkezetet alkot a polimerázban, mely az aktív helen érintkezést létesít a DNS-templát és az új RNS közt.[15]
- RPB3 (POLR2C): a harmadik legnagyobb alegység. Heterodimert alkot a POLR2Jvel magi egységet alkot. Az RPB1-gyel, 2-vel, 4-gyel, 5-tel, 7-tel és 10–12-vel kölcsönhat.[12]
- RNS-polimeráz II, B4 alegység (RPB4): a POLR2D kódolja,[16] a 4. legnagyobb, és védhet a stressztől.
- RPB5: A POLR2E gén kódolja. 2 van belőle minden RNS-polimeráz II-ben.[17] Az RPB1-gyel, 3-mal és 6-tal kölcsönhat erősen.[12]
- RPB6 (POLR2F): az átíró polimerázt a DNS-templáton stabilizáló komplexet alkot legalább 2 másik fehérjével.[18]
- RPB7: a POLR2G gén kódolja, és a polimerázfunkciót szabályozza.[19] Az RPB1-gyel és 5-tel erősen kölcsönhat.[12]
- RPB8R (POLR2H): az RPB1–3-mal, 5-tel és 7-tel kölcsönhat.[12]
- RPB9: a bemélyedés, ahol a DNS-templát transzkripciója történik, az RPB9-ből (POLR2I) és az RPB1-ből áll.
- RPB10: a POLR2L gén kódolja. Az RPB1–3-mal és 5-tel kölcsönhat, az RPB3-mal erősen.[12]
- RPB11: 3 alegységből áll, ezek a POLR2J (RPB11-a), a POLR2J2 (RPB11-b) és a POLR2J3 (RPB11-c).[20]
- RPB12 (POLR2K): Az RPB3-mal kölcsönhat.[12]
Szerkezet[szerkesztés]
Az RPB3 fontos az RNS-polimeráz II-szerkezetben.[21] Az alegységszintézis után hamar létrejön az RPB2–3 komplex.[21] Ez az RPB1-gyel kölcsönhat.[21] Az RPB3, 5 és 7 homodimereket alkotnak, és az RPB3 és 5 mindegyik alegységhez tud kapcsolódni, kivéve az RPB9-et.[12] Csak az RPB1 köt erősen az RPB5-höz.[12] Az RPB1 az RPB7-hez, az RPB10-hez, és gyengébben, de leghatékonyabban az RPB8-hoz köt.[12] Miután az RPB1 a komplexbe kerül, más egységek, például az RPB5 és az RPB7 bekerülhetnek, az RPB5 az RPB6-hoz és 8-hoz köt, az RPB3 az RPB10–12-höz.[12] Mikor a komplex nagyrészt összeállt, bekerül az RPB4 és 9. Az RPB4 az RPB7-tel komplexet alkot.[12]
Kinetika[szerkesztés]

Az RNS-polimeráz II (RNSP II, Pol II DNS-t pre-mRNS-re és a legtöbb kis magi RNS-re és mikro-RNS-re átíró fehérjekomplex.[1][22] Az eukarióták 3 RNS-polimerázának egyike.[3] 550 kDa-os, 12 alegységes komplex, és az egyik leginkább tanulmányozott RNS-polimeráz-típus. Számos transzkripciós faktor kell a korábbi promoterek kötéséhez és a transzkripció elindításához.
Felfedezés[szerkesztés]

A korai tanulmányok legalább 2 RNSP-t feltételeztek: egy rRNS-előállítót a nukleóluszban és egy más RNS-t a nukleoplazmában – a sejtmag nukleóluszon kívüli részén – előállítót.[5] 1969-ben Robert G. Roeder és William J. Rutter felfedezték, hogy 3 különböző magi RNS-polimeráz van – egy 3. felelt bizonyos RNS-ek nukleoplazmabeli átírásáért.[6] Ezt ioncsere-kromatográfia révén érték el DEAE-vel burkolt Sephadex gyöngyökkel. Ez az enzimeket megfelelő elúciók szerint választotta el I, II, III sorrendben az ammónium-szulfát-koncentráció növelésével. Az enzimeket elúciós sorrendben nevezték el RNS-polimeráz I-nek, II-nek és III-nak.[3] E felfedezés kimutatta, hogy van egy további enzim a nukleoplazmában, lehetővé téve az RNSP II és III megkülönböztetését.[23]
Az RNS-polimeráz II (RNSP2) szabályzott transzkripciós szüneteket tart az elongáció elején. Több tanulmány kimutatta az elongációs zavarok kapcsolatát a rákkal, a neurodegenerációval, a HIV-látenciával stb.[24]
Alegységek[szerkesztés]

Az eukarióta RNS-polimeráz II-magot először transzkripciós assay-kkel mutatták ki.[10] A tiszta enzim általában 10–12 alegységet (emberben és élesztőben 12-t) tartalmaz, és nem képes specifikus promoterfelismerésre.[11] Many subunit-subunit interactions are known.[12]
- RPB1 DNS-irányított RNS-polimeráz II-alegység: az emberben a POLR2A, élesztőben az RPO21 által kódolt enzim. A legnagyobb RNS-polimeráz II-egység. Maximum 52 YSPTSPS-ismétlődésből álló, a polimerázaktivitáshoz szükséges C-terminális doménje van.[13] Ezt először a Torontói Egyetemen dolgozó C. J. Ingles és a Johns Hopkins Egyetemen dolgozó J. L. Corden mutatták ki.A többi alegységgel együtt alkotja a polimeráz DNS-kötő doménjét, ahol a templát RNS-be való transzkripciója történik.[14] Erősen kölcsönhat az RPB8-cal.[12]
- RPB2 (POLR2B): a másodig legnagyobb alegység, mely legalább 2 másik polimerázzal kölcsönhat, szerkezetet alkot a polimerázban, mely az aktív helen érintkezést létesít a DNS-templát és az új RNS közt.[15]
- RPB3 (POLR2C): a harmadik legnagyobb alegység. Heterodimert alkot a POLR2Jvel magi egységet alkot. Az RPB1-gyel, 2-vel, 4-gyel, 5-tel, 7-tel és 10–12-vel kölcsönhat.[12]
- RNS-polimeráz II, B4 alegység (RPB4): a POLR2D kódolja,[16] a 4. legnagyobb, és védhet a stressztől.
- RPB5: A POLR2E gén kódolja. 2 van belőle minden RNS-polimeráz II-ben.[17] Az RPB1-gyel, 3-mal és 6-tal kölcsönhat erősen.[12]
- RPB6 (POLR2F): az átíró polimerázt a DNS-templáton stabilizáló komplexet alkot legalább 2 másik fehérjével.[18]
- RPB7: a POLR2G gén kódolja, és a polimerázfunkciót szabályozza.[19] Az RPB1-gyel és 5-tel erősen kölcsönhat.[12]
- RPB8R (POLR2H): az RPB1–3-mal, 5-tel és 7-tel kölcsönhat.[12]
- RPB9: a bemélyedés, ahol a DNS-templát transzkripciója történik, az RPB9-ből (POLR2I) és az RPB1-ből áll.
- RPB10: a POLR2L gén kódolja. Az RPB1–3-mal és 5-tel kölcsönhat, az RPB3-mal erősen.[12]
- RPB11: 3 alegységből áll, ezek a POLR2J (RPB11-a), a POLR2J2 (RPB11-b) és a POLR2J3 (RPB11-c).[20]
- RPB12 (POLR2K): Az RPB3-mal kölcsönhat.[12]
Szerkezet[szerkesztés]
Az RPB3 fontos az RNS-polimeráz II-szerkezetben.[21] Az alegységszintézis után hamar létrejön az RPB2–3 komplex.[21] Ez az RPB1-gyel kölcsönhat.[21] Az RPB3, 5 és 7 homodimereket alkotnak, és az RPB3 és 5 mindegyik alegységhez tud kapcsolódni, kivéve az RPB9-et.[12] Csak az RPB1 köt erősen az RPB5-höz.[12] Az RPB1 az RPB7-hez, az RPB10-hez, és gyengébben, de leghatékonyabban az RPB8-hoz köt.[12] Miután az RPB1 a komplexbe kerül, más egységek, például az RPB5 és az RPB7 bekerülhetnek, az RPB5 az RPB6-hoz és 8-hoz köt, az RPB3 az RPB10–12-höz.[12] Mikor a komplex nagyrészt összeállt, bekerül az RPB4 és 9. Az RPB4 az RPB7-tel komplexet alkot.[12]
Kinetika[szerkesztés]
Az enzimek akár több millió reakciót is katalizálhatnak másodpercenként. Sebességük az oldat és a szubsztrát koncentrációjától függ. Más enzimekhez hasonlóan van szaturációs görbéje; maximális sebessége ),[25] eltérő nukleotidok esetén Michaelis–Menten-állandója (a fél -hoz szükséges koncentráció), specificitási állandója (az egy aktív hely által másodpercenként kezelt szubsztrátok száma) eltérő. Az elméleti maximális specificitási állandó a 108 és 109 M−1s−1 közti diffúziós határ, ahol minden enzim-szubsztrát ütközés katalízist okoz. Élesztőben a legnagyobb alegység kapcsoló-kör doménjének mutációja módosíthatja az enzimkinetikát.[25]
Az RNS-polimerázzal rokon bakteriális RNS-polimeráz inaktív és aktív állapotok közt váltakozik a DNS-en való előre-hátra mozgással.[26] esetén az átlagos elongációs arány a bakteriális RNSP esetén körülbelül 1 bp/NTP.[26]
Az RNS-polimeráz II az elongáció során jelentős kotranszkripciós szüneteket tart.[27][28] Ez különösen nukleoszómáknál jelentős, és részben a polimeráz transzkripcióra kevésbé képes állapotba lépése okozza.[27] E szünetek időtartama másodpercektől percekig vagy még tovább is tarthat, a hosszú szünetekből való kilépést elongációs faktorok, például a TFIIS könnyíthetik.[29] A transzkripciós sebesség meghatározza, hogy az átírt nukleoszómák hisztonjai kikerülnek a kromatinból, vagy visszakerülnek a polimeráz mögött.[30]
α-Amanitin[szerkesztés]
Az RNS-polimeráz II-tt az α-amanitin[31] és más amatoxinok inhibeálják. Az α-amanitin sok gombában megtalálható erős méreg.[5] Az egyes RNS-polimerázokra eltérő hatása van: az RNSP I teljesen érintetlen, és normálisan működik, az RNSP III érzékenysége közepes. Az RNSP II-t azonban teljesen inhibeálja a toxin. Az α-amanitin az enzim RPB-1 egységének tölcsér-, bemélyedés és híd-α-hélix-régióiba ágyazódva inhibeálja az RNS-polimeráz II-t.[32]

Holoenzim[szerkesztés]
Az RNS-polimeráz II-holoenzim az eukarióta RNS-polimeráz II fehérjekódoló gének promotereinél aktivált RNS-polimeráz II-változat.[11] RNS-polimeráz II-ből, általános transzkripciós faktorokból és szabályzó SRB fehérjékből áll.
A holoenzim része a preiniciációs komplex, mivel létrejötte promoteren történik a transzkripció elindítása előtt. A mediátorkomplex a transzkripciós faktorok és az RNS-polimeráz II közti híd.
Kromatinszerkezet általi irányítás[szerkesztés]
Az utat a transzkripció szakaszaiban szabályzó fehérjék például:
- Preiniciáció (Bre1 általi segítés, hisztonmódosítás)[33]
- Iniciáció (TFIIH segíti; COMPASS általi Pol II-módosítás és -segítés, hisztonmódosítás)[34]
- Elongáció (SET2 általi segítés, hisztonmódosítás)[35]
Ez a folyamat szakaszaira szabályzási lépésekként tekint. Nem bizonyított, de valószínű szabályzó funkciójuk. A Pol II-elongációspromoterek 3 osztályba sorolhatók:
- Gyógyszer/szekvenciadependens leállás által érintett (Több interferáló fehérje)
- Kromatinszerkezet-orientált faktorok (hiszton-poszttranszkripciósmódosítók, például hiszton-metiltranszferázok)
- Pol II-katalízist-javító faktorok ctors (több kölcsönható fehérje és Pol II-kofaktor).
Transzkripciós mechanizmusok[szerkesztés]
- Kromatinstruktúra-orientált faktorok: HMT-k (hiszton-metiltranszferáz); COMPASS (Set1-asszociált fehérjék komplexe): a H3 hiszton 4. lizinjét metilálja; a transzkripciórepresszióért (csendesítés) felel. A sejtnövekedés- és transzkripciószabályzás normál része az RNSP II-ben.[36]
- Set2: a H3 hiszton 36. lizinjét metlálja, elongációszabályzó a CTD-vel való érintkezés révén.[37] A Dot1 a H3 79. lizinjét metilálja.
- Bre1: Ubikvitinálja a H2B hiszton 123. lizinjét, a preiniciációval függ össze és lehetővé teszi az RNSP II-kötést.
C-terminális domén[szerkesztés]
Az RPB1 C-terminusa alkotja a C-terminális domént (CTD). Ez általában a Tyr-Ser-Pro-Thr-Ser-Pro-Ser maximum 52 ismétlődéséből áll.[38] A domén az enzim magjától a kilépési csatornáig tart, ami az RNS-feldolgozó reakciók az RNS-feldolgozó szerkezettel való közvetlen és közvetett kölcsönhatások miatti indukciója miatt hatásos.[39] Az RNSP I-ben és III-ban nincs CTD.[3] Az RNS-polimeráz II CTD-jét először C. J. Ingles és J. Corden laboratóriumaiban fedezték fel az élesztő-, illetve egér-RPB1-et kódoló DNS szekvenálása során. Más fehérjék általában a polimerázaktiváláshoz kötnek a CTD-hez. Ez érintett a transzkripció iniciációjában, az 5’-sapka elhelyezésében és a spliceoszómához helyezésben a splicinghoz.[13]
CTD-foszforiláció[szerkesztés]
Az RNS-polimeráz II két formája a foszforilálatlan (IIA) és a foszforilált (IIO).[5][3] A kettő közti átmenet a transzkripció különböző funkcióit könnyíti meg. A CTD-foszforilációt a 6 általános transzkripciós faktor egyike, a TFIIH katalizálja. 2 célja van: az egyik a transzkripció kezdetén történő DNS-elválasztás, a másik a foszforiláció. Az IIA-forma a kerül a preiniciációs komplexben, feltehetően mert nagyobb a TATA-box-kötőfehérje-affinitása, mely a TFIID általános transzkripciós faktor alegysége, mint az IIO-formáé. Az IIO-forma katalizálja az RNS-lánc-elongációt.[5] Az elongáció az 5. helyen történő szerin TFIIH általi foszforilációjával történik. Ez enzimeket aktivál az 5’-vég lezárásához és a 3’-feldolgozófaktorokhoz a poli(A)-helyeknél.[39] A második szerin foszforilációja után aktiválódik az elongáció, ennek megállításához defoszforiláció kell. A domén teljes defoszforilációja után az RNSP II „újrahasznosul” és más iniciációs helyen katalizálja a folyamatot.[39]
Transzkripciókapcsolt rekombinációs javítás[szerkesztés]
A DNS-oxidáció akadályozhatja az RNSP II-transzkripciót és száltöréseket okozhat. Egy RNS-templátú transzkripciókapcsolt rekombinációs folyamatról leírták, hogy védhet a DNS-károsodással szemben.[40] A sejtciklus G1/G0 szakaszai során a sejtek homológ rekombinációs faktorokat hoznak létre a kettősszál-töréseknél az aktívan átírt részekben. Ez a DNS-kettősszál-törések javításával egybefügg az RNS-templátú homológ rekombináció révén. E folyamat hatékonyan és pontosan illeszti össze az RNS-polimeráz II által átírt gének kettősszál-töréseknél lévő szakaszokat.
Jegyzetek[szerkesztés]
- ↑ a b Kornberg RD (1999. december 1.). „Eukaryotic transcriptional control”. Trends in Cell Biology 9 (12), M46–M49. o. DOI:10.1016/S0962-8924(99)01679-7. PMID 10611681.
- ↑ Sims RJ, Mandal SS, Reinberg D (2004. június 1.). „Recent highlights of RNA-polymerase-II-mediated transcription”. Current Opinion in Cell Biology 16 (3), 263–271. o. DOI:10.1016/j.ceb.2004.04.004. PMID 15145350.
- ↑ a b c d e f Young, Richard A. (2003. november 28.). „RNA Polymerase II” (angol nyelven). Annual Review of Biochemistry 60 (1), 689–715. o. DOI:10.1146/annurev.bi.60.070191.003353. PMID 1883205.
- ↑ a b Meyer PA, Ye P, Zhang M, Suh MH, Fu J (2006. június 1.). „Phasing RNA polymerase II using intrinsically bound Zn atoms: an updated structural model”. Structure 14 (6), 973–82. o. DOI:10.1016/j.str.2006.04.003. PMID 16765890.
- ↑ a b c d e Weaver, Robert Franklin. Molecular biology. McGraw-Hill (2012. január 1.). ISBN 9780073525327. OCLC 789601172
- ↑ a b Roeder RG, Rutter WJ (1969. október 1.). „Multiple forms of DNA-dependent RNA polymerase in eukaryotic organisms”. Nature 224 (5216), 234–237. o. DOI:10.1038/224234a0. PMID 5344598.
- ↑ Roeder RG, Rutter WJ (1969. október 1.). „Multiple forms of DNA-dependent RNA polymerase in eukaryotic organisms”. Nature 224 (5216), 234–7. o. DOI:10.1038/224234a0. PMID 5344598.
- ↑ Cermakova, Katerina (2021. november 26.). „A ubiquitous disordered protein interaction module orchestrates transcription elongation”. Science 374 (6571), 1113–1121. o. DOI:10.1126/science.abe2913. PMID 34822292.
- ↑ a b Armache, Karim-Jean (2019). „Structures of complete RNA polymerase II and its subcomplex, Rpb4/7”. Journal of Biological Chemistry 280 (8), 7131–1734. o. DOI:10.2210/pdb1wcm/pdb. PMID 15591044.
- ↑ a b Sawadogo M, Sentenac A (1990). „RNA polymerase B (II) and general transcription factors”. Annual Review of Biochemistry 59, 711–754. o. DOI:10.1146/annurev.bi.59.070190.003431. PMID 2197989.
- ↑ a b c Myer VE, Young RA (1998. október 1.). „RNA polymerase II holoenzymes and subcomplexes”. The Journal of Biological Chemistry 273 (43), 27757–27760. o. DOI:10.1074/jbc.273.43.27757. PMID 9774381.
- ↑ a b c d e f g h i j k l m n o p q r s t u v w x y z Acker J, de Graaff M, Cheynel I, Khazak V, Kedinger C, Vigneron M (1997. július 1.). „Interactions between the human RNA polymerase II subunits”. The Journal of Biological Chemistry 272 (27), 16815–16821. o. DOI:10.1074/jbc.272.27.16815. PMID 9201987.
- ↑ a b c Brickey WJ, Greenleaf AL (1995. június 1.). „Functional studies of the carboxy-terminal repeat domain of Drosophila RNA polymerase II in vivo”. Genetics 140 (2), 599–613. o. DOI:10.1093/genetics/140.2.599. PMID 7498740.
- ↑ a b Entrez Gene: POLR2A polymerase (RNA) II (DNA directed) polypeptide A, 220kDa
- ↑ a b Entrez Gene: POLR2B polymerase (RNA) II (DNA directed) polypeptide B, 140kDa
- ↑ a b Khazak V, Estojak J, Cho H, Majors J, Sonoda G, Testa JR, Golemis EA (1998. április 1.). „Analysis of the interaction of the novel RNA polymerase II (pol II) subunit hsRPB4 with its partner hsRPB7 and with pol II”. Molecular and Cellular Biology 18 (4), 1935–1945. o. DOI:10.1128/mcb.18.4.1935. PMID 9528765.
- ↑ a b Entrez Gene: POLR2E polymerase (RNA) II (DNA directed) polypeptide E, 25kDa
- ↑ a b Entrez Gene: POLR2F polymerase (RNA) II (DNA directed) polypeptide F
- ↑ a b Entrez Gene: POLR2G polymerase (RNA) II (DNA directed) polypeptide G
- ↑ a b POLR2J3 polymerase (RNA) II (DNA directed) polypeptide J3
- ↑ a b c d e f Kolodziej PA, Young RA (1991. szeptember 1.). „Mutations in the three largest subunits of yeast RNA polymerase II that affect enzyme assembly”. Molecular and Cellular Biology 11 (9), 4669–4678. o. DOI:10.1128/mcb.11.9.4669. PMID 1715023.
- ↑ Sims RJ, Mandal SS, Reinberg D (2004. június 1.). „Recent highlights of RNA-polymerase-II-mediated transcription”. Current Opinion in Cell Biology 16 (3), 263–271. o. DOI:10.1016/j.ceb.2004.04.004. PMID 15145350.
- ↑ Roeder RG, Rutter WJ (1969. október 1.). „Multiple forms of DNA-dependent RNA polymerase in eukaryotic organisms”. Nature 224 (5216), 234–7. o. DOI:10.1038/224234a0. PMID 5344598.
- ↑ Cermakova, Katerina (2021. november 26.). „A ubiquitous disordered protein interaction module orchestrates transcription elongation”. Science 374 (6571), 1113–1121. o. DOI:10.1126/science.abe2913. PMID 34822292.
- ↑ a b Kaplan CD, Jin H, Zhang IL, Belyanin A (2012. április 12.). „Dissection of Pol II trigger loop function and Pol II activity-dependent control of start site selection in vivo”. PLOS Genetics 8 (4), e1002627. o. DOI:10.1371/journal.pgen.1002627. PMID 22511879.
- ↑ a b Abbondanzieri EA, Greenleaf WJ, Shaevitz JW, Landick R, Block SM (2005. november 1.). „Direct observation of base-pair stepping by RNA polymerase”. Nature 438 (7067), 460–5. o. DOI:10.1038/nature04268. PMID 16284617.
- ↑ a b Hodges, Courtney (2009. július 31.). „Nucleosomal fluctuations govern the transcription dynamics of RNA polymerase II”. Science 325 (5940), 626–628. o. DOI:10.1126/science.1172926. ISSN 1095-9203. PMID 19644123.
- ↑ Churchman, L. Stirling (2011. január 20.). „Nascent transcript sequencing visualizes transcription at nucleotide resolution”. Nature 469 (7330), 368–373. o. DOI:10.1038/nature09652. ISSN 1476-4687. PMID 21248844.
- ↑ Galburt, Eric A. (2007. április 12.). „Backtracking determines the force sensitivity of RNAP II in a factor-dependent manner”. Nature 446 (7137), 820–823. o. DOI:10.1038/nature05701. ISSN 1476-4687. PMID 17361130.
- ↑ Bintu, Lacramioara (2011. november 13.). „The elongation rate of RNA polymerase determines the fate of transcribed nucleosomes”. Nature Structural & Molecular Biology 18 (12), 1394–1399. o. DOI:10.1038/nsmb.2164. ISSN 1545-9985. PMID 22081017.
- ↑ Kaplan CD, Larsson KM, Kornberg RD (2008. június 1.). „The RNA polymerase II trigger loop functions in substrate selection and is directly targeted by alpha-amanitin”. Molecular Cell 30 (5), 547–556. o. DOI:10.1016/j.molcel.2008.04.023. PMID 18538653.
- ↑ Gong, Xue Q. (2004. június 25.). „α-Amanitin Blocks Translocation by Human RNA Polymerase II” (angol nyelven). Journal of Biological Chemistry 279 (26), 27422–27427. o. DOI:10.1074/jbc.M402163200. ISSN 0021-9258. PMID 15096519.
- ↑ Wood A, Schneider J, Dover J, Johnston M, Shilatifard A (2003. szeptember 12.). „f1 complex is essential for histone monoubiquitination by the Rad6-Bre1 complex, which signals for histone methylation by COMPASS and Dot1p”. J Biol Chem 278 (37), 34739–34742. o. DOI:10.1074/jbc.C300269200. PMID 12876294.

- ↑ Ebmeier CC, Erickson B, Allen BL, Allen MA, Kim H, Fong N, Jacobsen JR, Liang K, Shilatifard A, Dowell RD, Old WM, Bentley DL, Taatjes DJ (2017. augusztus 1.). „Human TFIIH Kinase CDK7 Regulates Transcription-Associated Chromatin Modifications”. Cell Rep 20 (5), 1173–1186. o. DOI:10.1016/j.celrep.2017.07.021. PMID 28768201.

- ↑ Carrozza MJ, Li B, Florens L, Suganuma T, Swanson SK, Lee KK, Shia WJ, Anderson S, Yates J, Washburn MP, Workman JL. Histone H3 methylation by Set2 directs deacetylation of coding regions by Rpd3S to suppress spurious intragenic transcription (2005. november 18.). „{{{title}}}”. Cell 123 (4), 581–592. o. DOI:10.1016/j.cell.2005.10.023. PMID 16286007.

- ↑ Briggs, Scott D. (2001. december 15.). „Histone H3 lysine 4 methylation is mediated by Set1 and required for cell growth and rDNA silencing in Saccharomyces cerevisiae” (angol nyelven). Genes & Development 15 (24), 3286–3295. o. DOI:10.1101/gad.940201. ISSN 0890-9369. PMID 11751634.
- ↑ Li, Bing (2003. március 14.). „The Set2 Histone Methyltransferase Functions through the Phosphorylated Carboxyl-terminal Domain of RNA Polymerase II” (angol nyelven). Journal of Biological Chemistry 278 (11), 8897–8903. o. DOI:10.1074/jbc.M212134200. ISSN 0021-9258. PMID 12511561.
- ↑ Meinhart A, Cramer P (2004. július 1.). „Recognition of RNA polymerase II carboxy-terminal domain by 3'-RNA-processing factors”. Nature 430 (6996), 223–226. o. DOI:10.1038/nature02679. PMID 15241417.
- ↑ a b c Egloff, Sylvain (2008). „Cracking the RNA polymerase II CTD code”. Trends in Genetics 24 (6), 280–288. o. DOI:10.1016/j.tig.2008.03.008. PMID 18457900.
- ↑ Wei L, Levine AS, Lan L (2016). „Transcription-coupled homologous recombination after oxidative damage”. DNA Repair (Amst.) 44, 76–80. o. DOI:10.1016/j.dnarep.2016.05.009. PMID 27233112.
Fordítás[szerkesztés]
Ez a szócikk részben vagy egészben a RNA polymerase II című angol Wikipédia-szócikk ezen változatának fordításán alapul. Az eredeti cikk szerkesztőit annak laptörténete sorolja fel. Ez a jelzés csupán a megfogalmazás eredetét és a szerzői jogokat jelzi, nem szolgál a cikkben szereplő információk forrásmegjelöléseként.
További információk[szerkesztés]
- More information at Berkeley National Lab (Wayback Machine copy)
- RNA+Polymerase+II a U.S. National Library of Medicine Medical Subject Headings (MeSH) honlapján
| Az itt található információk kizárólag tájékoztató jellegűek, nem minősülnek orvosi szakvéleménynek, nem pótolják az orvosi kivizsgálást és kezelést. A cikk tartalmát a Wikipédia önkéntes szerkesztői alakítják ki, és bármikor módosulhat. |




![{\displaystyle \mathrm {[NTP]_{e}q=10\mu {M}GTP,10\mu {M}UTP,5\mu {M}ATP,2,5\mu {M}CTP} }](https://wikimedia.org/api/rest_v1/media/math/render/svg/f3e27894b3116532f308543727cc8bca6895bc4a)