PA proteázklán
A PA klán (vegyes nukleofilú Proteázok, A szupercsalád) a szerkezeti homológia alapján azonosított közös őssel rendelkező legnagyobb proteázcsalád. Tagjai chimotripszinszerű szerkezettel és hasonló proteolízis-mechanizmussal rendelkeznek, de hasonlóságuk 10%-nál is kisebb lehet. Cisztein- és szerinproteázok (eltérő nukleofilok) is tagjai.[1][2] A család tagjai találhatók növényekben,[3] állatokban,[3] gombákban,[3] eubaktériumokban,[4] archeákban[5][6] és vírusokban.[2]
A katalitikus hármas elnevezés több hidrolízist végző proteázklánra való használata a konvergens evolúció példája.[7] A PA klán katalitikus hármasának eltérései az enzimek aktív helyeinek divergens evolúciójára példák.[2]
Története[szerkesztés]
Az 1960-as években egyes proteázok hasonló szekvenciáját fedezték fel, mely az evolúciós hasonlóságukat jelentette.[8] Ezeket a chimotripszinszerű szerinproteázokhoz (ma S1 család) sorolták.[9] Miután ezek és más proteázok szerkezetét röntgenkrisztallográfiával az 1970-es és 1980-as években felismerték, kiderült, hogy egyes vírusproteázok, például a dohányfoltosságvírus-proteáz szerkezeti homológiát mutattak, bár nem volt a szekvencia hasonló, és a nukleofil is eltért.[2][10][11] Szerkezeti homológia alapján létrehoztak egy fehérje-szupercsaládot, melyet később PA klánnak neveztek el a MEROPS osztályozási rendszer alapján. A további szerkezetek megismerésével további családok kerültek a PA klánba.[12][13]
Etimológia[szerkesztés]
A P a vegyes nukleofilú proteázokat jelenti. Az A azt jelenti, hogy ez volt az első azonosított proteázklán (vannak továbbá PB, PC, PD és PE klánok is).[1]
Szerkezet[szerkesztés]
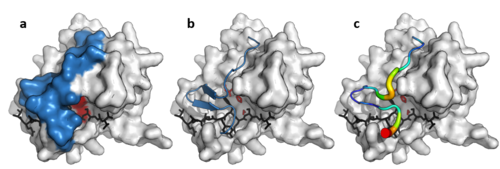
Bár csak 10%-os szekvenciahasonlóságot mutatnak, a PA-klán vírusokból, prokariótákból és eukariótákból izolált tagjai szerkezeti homológiát mutatnak, és szerkezeti hasonlóság alapján elrendezhetők (például DALI-val).[3]
Kettős β-redő[szerkesztés]
A PA-klán proteázainak közös jellemzője a két β-redő, ahol a kovalens katalízist sav-hisztidin-nukleofil katalitikus hármas végzi. A hordók egymásra merőlegesen helyezkednek el, és hidrofób molekularészek tartják össze. A hármas részei a két hordó közt megoszlanak, így a katalízis a felületen történik.[14]
Virális proteázgyűrű[szerkesztés]
A kettős β-redő mellett egyes vírusproteázok (például a TEV proteáz) hosszú, rugalmas C-terminális gyűrűvel rendelkeznek, mely a szubsztrátot teljesen elfedi, és kötőalagutat hoz létre. Ez szorosan kötődő térrészeket hoz létre, így a szubsztrát oldalláncai (P6–P1’) egy komplementer helyhez (S6–S1’) kötődik, a specificitást az enzim és a szubsztrát nagy érintkezési felülete biztosítja.[11] Az ezzel nem rendelkező sejtproteázok, például a tripszin specificitása kisebb.
Evolúció és funkció[szerkesztés]
Katalitikus aktivitás[szerkesztés]
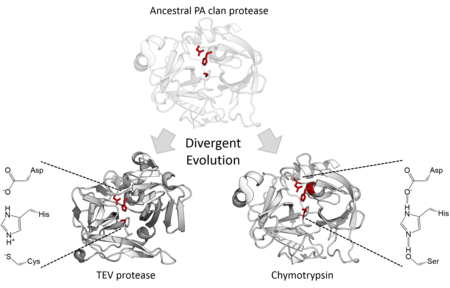
A strukturális homológia alapján a PA klán tagjai azonos szerkezetű közös ősből származnak. Bár mindegyik tagja kétlépéses nukleofil katalízist végez,[7] egyes családok szerint, mások ciszteint használnak nukleofilként.[2] A szupercsalád így különleges példa a divergens enzimevolúcióra, mivel a központi katalitikus molekularészlet megváltozott egyes családokban.[15] Emellett az irányított evolúcióról kiderült, hogy képes ciszteinproteázt aktív szerinproteázzá alakítani.[16] A PA klán minden sejtben aktív tagja szerinproteáz, azonban a vírusproteázok közt vannak szerin- és ciszteinproteáz-családok.[7] Ezek többsége endopeptidáz, az exopeptidázok S46 családja kivétel.[17][18]
Biológiai szerep és szubsztrátspecificitás[szerkesztés]
A központi katalitikus részletben való divergencia mellett jelentős divergens evolúció történt a funkcióban. A PA klán tagjai megtalálhatók eukariótákban, prokariótákban és vírusokban, és számos funkciót töltenek be. Az emlősökben egy részük a véralvadásban fontos (például a trombin), így nagymértékű a szubsztrátspecificitásuk, míg mások az emésztésben (például a tripszin), így szubsztrátspecificitásuk kisebb. Egyes kígyómérgek is ilyen fehérjék, például a Crotalinae hemotoxinja, és a véralvadásba avatkoznak be. Ezenkívül a baktériumok (például a Staphylococcus aureus) exfoliatint bocsátanak ki, mellyel a gazdaszervezet szöveteit sértik és emésztik meg. Sok vírus genomját egyetlen poliproteinként expresszálja, és a PA-klánba tartozó proteázt használja ennek működésképes részekre bontásában (például ilyen vírusok a poliovírus, a norovírus, ilyen proteázok például a TEV proteázok).[19][20]
Ezenkívül a szupercsaládba tartoznak pszeudoenzimek, ahol a katalitikus hármas tagjai megváltoztak, és kötőfehérjékként működnek.[21] Például a heparinkötő azurocidin esetén a nukleofil helyén glicin, a hisztidin helyén szerin van.[22]
Családok[szerkesztés]
A PA klánban a családokat katalitikus nukleofilnak megfelelően jelölik. A teljes PA klán szekvenciahomológiájának hiánya ellenére szekvenciahasonlósággal azonosíthatók az egyes családjai.
| Család | Példák | Ismert szerkezet? |
|---|---|---|
| C03 | polio-type picornain 3C (poliovirus) | Igen |
| C04 | dohányfoltosságvírus-proteáz (dohányfoltosság-vírus) | Igen |
| C24 | nyúlvérzésvírus 3C-szerű peptidáz (nyúlvérzésvírus) | Nem |
| C30 | terjedőgasztroenteritiszvírus-szerű főpeptidáz (terjedőgasztroenteritisz-vírus) | Igen |
| C37 | kalicivirin (Southampton-vírus) | Igen |
| C62 | okavírus 3C-szerű peptidáz (okavírus) | Nem |
| C74 | pestivírus N52 peptidáz (marhahasmenés-vírus 1) | Nem |
| C99 | iflavírus-feldolgozópeptidáz (Ectropis obliqua picorna-szerű vírus) | Nem |
| C107 | alfamezonivírus 3C-szeű peptidáz (Cavally-vírus) | Nem |
| S01 | chimotripszin A (Bos taurus) | Igen |
| S03 | togavirin (Sindbis-vírus) | Igen |
| S06 | IgA-specifikus szerin-endopeptidáz (Neisseria gonorrhoeae) | Igen |
| S07 | flavivirin (sárgalázvírus) | Nem |
| S29 | hepacivirin (hepatitis C-vírus) | Igen |
| S30 | potivírus P1 peptidáz (szilvahimlővírus) | Nem |
| S31 | pestivírus NS3-poliprotein-peptidáz (marhahasmenés-vírus 1) | Nem |
| S32 | lóarterivírus-szerinpeptidáz (lóarteritisz-vírus) | Igen |
| S39 | szobemovírus-peptidáz (Dactylis-mozaikvírus) | Igen |
| S46 | dipeptidil-peptidáz 7 (Porphyromonas gingivalis) | Nem |
| S55 | SpoIVB-peptidáz (Bacillus subtilis) | Nem |
| S64 | Ssy5 peptidáz (Saccharomyces cerevisiae) | Nem |
| S65 | pikornainszerű ciszteinpeptidáz (Breda-1 torovírus) | Nem |
| S75 | karikakeszegvírus-szerinpeptidáz (karikakeszeg-vírus) | Nem |
Jegyzetek[szerkesztés]
- ↑ a b N. D. Rawlings, A. J. Barrett, A. Bateman (2012. január 1.). „MEROPS: the database of proteolytic enzymes, their substrates and inhibitors”. Nucleic Acids Research 40 (Adatbázis), D343-350. o. DOI:10.1093/nar/gkr987. PMID 22086950.
- ↑ a b c d e J. F. Bazan, R. J. Fletterick (1988. november 1.). „Viral cysteine proteases are homologous to the trypsin-like family of serine proteases: structural and functional implications”. Proceedings of the National Academy of Sciences of the United States of America 85 (21), 7872–6. o. DOI:10.1073/pnas.85.21.7872. PMID 3186696.
- ↑ a b c d A. Laskar, E. J. Rodger, A. Chatterjee, C. Mandal (2012. május 1.). „Modeling and structural analysis of PA clan serine proteases”. BMC Research Notes 5, 256. o. DOI:10.1186/1756-0500-5-256. PMID 22624962.
- ↑ J. A. Barbosa, J. W. Saldanha, R. C. Garratt (1996. július 1.). „Novel features of serine protease active sites and specificity pockets: sequence analysis and modelling studies of glutamate-specific endopeptidases and epidermolytic toxins”. Protein Engineering 9 (7), 591–601. o. DOI:10.1093/protein/9.7.591. PMID 8844831.
- ↑ MEROPS - Archaeal S01 proteases
- ↑ (2014. március 1.) „Bacterial serine proteases secreted by the autotransporter pathway: classification, specificity, and role in virulence”. Cellular and Molecular Life Sciences 71 (5), 745–70. o. DOI:10.1007/s00018-013-1355-8. PMID 23689588.
- ↑ a b c A. R. Buller, C. A. Townsend (2013. február 1.). „Intrinsic evolutionary constraints on protease structure, enzyme acylation, and the identity of the catalytic triad”. Proceedings of the National Academy of Sciences of the United States of America 110 (8), E653-61. o. DOI:10.1073/pnas.1221050110. PMID 23382230.
- ↑ C. de Haën, H. Neurath, D. C. Teller (1975. február 1.). „The phylogeny of trypsin-related serine proteases and their zymogens. New methods for the investigation of distant evolutionary relationships”. Journal of Molecular Biology 92 (2), 225–259. o. DOI:10.1016/0022-2836(75)90225-9. PMID 1142424.
- ↑ (1996. május 1.) „Conservation and variability in the structures of serine proteinases of the chymotrypsin family”. Journal of Molecular Biology 258 (3), 501–37. o. DOI:10.1006/jmbi.1996.0264. PMID 8642605.
- ↑ (1986. január 1.) „Poliovirus-encoded proteinase 3C: a possible evolutionary link between cellular serine and cysteine proteinase families”. FEBS Letters 194 (2), 253–7. o. DOI:10.1016/0014-5793(86)80095-3. PMID 3000829.
- ↑ a b J. Phan, A. Zdanov, A. G. Evdokimov, J. E. Tropea, H. K. Peters, R. B. Kapust, M. Li, A. Wlodawer, D. S. Waugh (2002. december 1.). „Structural basis for the substrate specificity of tobacco etch virus protease”. The Journal of Biological Chemistry 277 (52), 50564–72. o. DOI:10.1074/jbc.M207224200. PMID 12377789.
- ↑ (1994. május 1.) „Picornaviral 3C cysteine proteinases have a fold similar to chymotrypsin-like serine proteinases”. Nature 369 (6475), 72–6. o. DOI:10.1038/369072a0. PMID 8164744.
- ↑ (1996. március 1.) „The arterivirus nsp4 protease is the prototype of a novel group of chymotrypsin-like enzymes, the 3C-like serine proteases”. The Journal of Biological Chemistry 271 (9), 4864–71. o. DOI:10.1074/jbc.271.9.4864. PMID 8617757.
- ↑ (1989. szeptember 1.) „Characterization of the catalytic residues of the tobacco etch virus 49-kDa proteinase”. Virology 172 (1), 302–310. o. DOI:10.1016/0042-6822(89)90132-3. PMID 2475971.
- ↑ A. Laskar, E. J. Rodger, A. Chatterjee, C. Mandal (2012. május 1.). „Modeling and structural analysis of PA clan serine proteases” (angol nyelven). BMC Research Notes 5 (1), 256. o. DOI:10.1186/1756-0500-5-256. PMID 22624962.
- ↑ T. Shafee, P. Gatti-Lafranconi, R. Minter, F. Hollfelder (2015. szeptember 1.). „Handicap-Recover Evolution Leads to a Chemically Versatile, Nucleophile-Permissive Protease”. ChemBioChem 16 (13), 1866–1869. o. DOI:10.1002/cbic.201500295. PMID 26097079.
- ↑ Y. Suzuki, Y. Sakamoto, N. Tanaka, H. Okada, Y. Morikawa, W. Ogasawara (2014. március 1.). „Identification of the catalytic triad of family S46 exopeptidases, closely related to clan PA endopeptidases”. Scientific Reports 4, 4292. o. DOI:10.1038/srep04292. PMID 24598890.
- ↑ Y. Sakamoto, Y. Suzuki, I. Iizuka, C. Tateoka, S. Roppongi, M. Fujimoto, K. Inaka, H. Tanaka, M. Masaki, K. Ohta, H. Okada, T. Nonaka, Y. Morikawa, K. T. Nakamura, W. Ogasawara, N. Tanaka (2014. május 1.). „S46 peptidases are the first exopeptidases to be members of clan PA”. Scientific Reports 4, 4977. o. DOI:10.1038/srep04977. PMID 24827749.
- ↑ Handbook of proteolytic enzymes. Boston: Academic Press (2013). ISBN 9780123822192
- ↑ L. Polgár (2005. október 1.). „The catalytic triad of serine peptidases”. Cellular and Molecular Life Sciences 62 (19–20), 2161–72. o. DOI:10.1007/s00018-005-5160-x. PMID 16003488.
- ↑ (2002. október 1.) „Sequence and structural differences between enzyme and nonenzyme homologs”. Structure 10 (10), 1435–51. o. DOI:10.1016/s0969-2126(02)00861-4. PMID 12377129.
- ↑ (1997. április 1.) „Structure of HBP, a multifunctional protein with a serine proteinase fold”. Nature Structural Biology 4 (4), 265–8. o. DOI:10.1038/nsb0497-265. PMID 9095193.
Fordítás[szerkesztés]
Ez a szócikk részben vagy egészben a PA clan of proteases című angol Wikipédia-szócikk ezen változatának fordításán alapul. Az eredeti cikk szerkesztőit annak laptörténete sorolja fel. Ez a jelzés csupán a megfogalmazás eredetét és a szerzői jogokat jelzi, nem szolgál a cikkben szereplő információk forrásmegjelöléseként.
Kapcsolódó szócikkek[szerkesztés]
További információk[szerkesztés]
- MEROPS: fehérje-adatbázis
- Superfamily Archiválva 2016. június 24-i dátummal a Wayback Machine-ben: fehérjeszerkezet-adatbázis


